-
传统的生物量估测主要运用模型估计法,过去的几十年中建立的生物量模型超过2 000个,涉及100多个树种[1-3]。虽精度高、适用性强,却耗时费力,破坏性较大,且难以进行大范围、长时间跨度的生物量动态监测[4]。遥感技术的飞速发展,使得快速获取区域尺度森林生物量的空间分布成为了可能。许多学者运用光学和雷达数据构建生物量反演模型,取得了一定的效果,但易饱和、不稳定等缺陷使其在区域范围应用时难以保证足够的精度[5-9]。
激光雷达(LiDAR)是一项主动遥感技术,因其能精准地获取森林结构参数,大幅提高了生物量的估测精度[10-11]。利用LiDAR估测生物量一般可以分为直接法和间接法:直接法是将LiDAR提取的高度、密度、强度等变量作为输入,直接建立与生物量的回归关系;间接法是先利用LiDAR变量估计胸径、郁闭度、叶面积指数、蓄积量等森林参数,再通过林木生长方程或生物量转换因子(BEF)等计算生物量[12-13]。已有的成果大都针对森林生物量或地上部分生物量,而对组分生物量的研究较少。庞勇等[14]利用LiDAR点云数据,采用逐步回归法估测了小兴安岭森林组分生物量,取得了较高的精度。然而,遥感参数与生物量存在复杂的非线性关系,传统的回归方法并不能进行有效和完整的表达,且推导的关系一般仅适用于研究区,模型的泛化能力不强[15]。本研究以黑龙江省长白落叶松(Larix olgensis Henry.)为研究对象,以机载LiDAR提取的高度、密度变量组为基础,分别采用多元线性回归(MLR)和随机森林(RF)方法,构建组分生物量反演模型,讨论与评价不同方法对组分生物量估测精度的影响。
-
研究区位于黑龙江省孟家岗林场(46°20′46°30′ N,130°32′ 130°52′ E),地处完达山西麓余脉,以低山丘陵为主,地势东北高,西南低,海拔170575 m,平均海拔为250 m,坡度位于10°20°之间,较为平缓。土壤种类以典型暗棕壤为主,其次为白浆化暗棕壤,另有少量的潜育暗棕壤、原始暗棕壤、草甸暗棕壤等。
根据2016年森林资源二类调查,林场经营面积15 503 hm2,林地面积13 671 hm2,森林覆盖率达86.3%,活立木总蓄积量1 464 508 m3。主要树种以长白落叶松、樟子松(Pinus sylvestris L. var. mongolica Litv.)和红松(Pinus koraiensis Sieb. et Zucc.)等人工林为主,面积约占2/3,其他天然次生林约占1/3。
-
2016年6—7月,采用LiCHy(LiDAR, CCD and Hyperspectral)系统在研究区进行LiDAR点云和高清影像的采集,具体参数见1/2。使用“运-5”飞机作为飞行平台,平均飞行高度2 500 m,飞行相对地面速度约200 km·h-1。总覆盖面积约300 km2,共获取83条航带数据,航带平均扫描宽度约1 000 m,航带重叠率60%。
为了精确提取森林垂直结构参数,须对LiDAR点云数据进行预处理。航带拼接时对重叠区域点云予以剔除,以保证点云在研究区密度水平的一致性。采用改进的渐进加密三角网(IPTD)滤波算法[17],对点云进行分类,即区分地面点和非地面点;利用高程阈值法(Elevation>2 m)结合人工编辑,将非地面点区分为植被点和其他类型点云。分类后的地面点通过TIN插值方法生成数字高程模型(DEM),对植被点(DSM)与DEM取差值得到冠层高度模型(CHM),由此对植被点的高度进行了归一化处理,消除了地形变化的影响。
以样地为统计单元提取LiDAR点云数据的高度、密度变量组作为落叶松生物量反演模型的输入变量[18]。高度变量组(46个)包括:高度百分位数(H1, H5, H10, …, H95, H99)15个、累计高度百分位数(AIH1, AIH5, AIH10, …, AIH95, AIH99)15个、最大高度(Hmax)、最低高度(Hmin)、平均高度(Hmean)、高度中位数(Hmedian)、高度百分位数四分位数间距(Hinterval)以及高度的峰度(Hkurtosis)、平均偏差(Hmab)、变异系数(Hcv)、标准差(Hstddev)等。密度变量组(10个)为将点云(>2 m)从低到高分成十个相同的高度层,每层回波数的比例即为相应的密度变量(D5, D10, D20, …, D80, D90)。
-
2016年6—7月,与飞行实验同步进行地面样地调查。根据《黑龙江省森林资源规划设计调查技术操作细则》的龄组划分标准,按幼龄林(≤20 a)、中龄林(21~30 a)、近熟林(31~40 a)、成熟林(≥41 a,成熟林与过熟林合并处理)选择典型长白落叶松人工林,设置0.06 hm2(20 m×30 m)样地40块。对样地进行每木检尺,起测胸径为5 cm。使用DGPS对样地4个角进行定位,精度控制在cm级(10 cm以内),地面调查与机载实验的差分基站相同,保证外业调查数据与机载飞行数据的位置有可靠的一致性。
在样地调查基础上,分别4个林龄各选取4块标准地,每块标准地按优势木、劣势木各1株,标准木2株共选取64株解析样木(表 1)。地上部分采用Monsic分层切割法测定样木的干、皮、树、叶的鲜质量。地下部分采用全根挖掘法,分别测定小根(≤2 cm)、粗根(25 cm)、根茎(≥5 cm)的鲜质量。各器官分别取样后,在105 ℃下进行30 min的杀青处理,然后将烘箱的温度调到80 ℃烘干至恒质量,测出各组分样品的含水率。根据样品含水率乘以总鲜质量推算出各组分生物量[19]。
表 1 样木数据概况
Table 1. Summary statistics of sampling trees
林龄
Ages/a样木数量
Numbers胸径
D/cm树高
Height/m树干
Stem/kg树皮
Bark/kg树枝
Branch/kg树叶
Leaf/kg树根
Under/kg幼龄林(419)
Young stand16 3.49.2 3.713.8 1.715.9 0.32.9 0.74.8 0.62.0 0.85.2 中龄林(2129)
Middle-age stand16 9.213.1 6.517.1 10.148.6 2.17.1 3.79.0 1.53.1 4.711.9 近熟林(3139)
Near-mature stand16 12.916.7 11.318.4 26.9106.4 4.012.3 5.412.3 1.83.5 7.420.1 成熟林(4155)
Mature stand16 17.223.0 11.720.8 64.0185.7 7.919.2 8.719.4 2.55.6 15.849.9 -
经对数转换的异速生长模型是生物量估测的常用方法(式1),本研究以64株解析木数据为基础,利用似然无关法构建研究区落叶松人工林生物量相容性模型(式2)。
$ {\rm{ln}}\;M = {\rm{ln}}(a) + b \times {\rm{ln}}DBH $
(1) $ \left\{ \begin{align} & \text{ln}\ {{M}_{stem}}=\text{ln}({{a}_{1}})+{{b}_{1}}\times \text{ln}D~ \\ & \text{ln}\ {{M}_{Bark}}=\text{ln}({{a}_{2}})+{{b}_{2}}\times \text{ln}D~ \\ & \text{ln}\ {{M}_{Branch}}=\text{ln}({{a}_{3}})+{{b}_{3}}\times \text{ln}D~ \\ & \text{ln}\ {{M}_{Leaf}}=\text{ln}({{a}_{4}})+{{b}_{4}}\times \text{ln}D~ \\ & \text{ln}\ {{M}_{Under}}=\text{ln}({{a}_{5}})+{{b}_{5}}\times \text{ln}D \\ & ~\text{ln}\ {{M}_{Above}}=\text{ln}({{a}_{6}})+{{b}_{6}}\times \text{ln}D \\ & ~\text{ln}\ {{M}_{Total}}=\text{ln}({{a}_{7}})+{{b}_{7}}\times \text{ln}D \\ \end{align} \right. $
(2) 式中,Mstem,MBrak,MBranch,MLeaf,MUnder,MAbove,MTotal分别代表树干、树皮、树枝、树叶、树根、地上生物量、全株生物量,D为胸径,ai、bi为模型参数。
根据40个样地每木检尺数据,利用建立的单木水平生物量模型计算得到各单木的总生物量和各组分生物量,再分别汇总得到各样地的总生物量和各组分生物量,见表 2。
表 2 模型参数估计值及评价统计指标
Table 2. Parameter estimates and evaluation statistics of biomass model
组分
Components参数估计值
Parameter estimates统计指标
Evaluation statisticsa b Radj2 SEE/t TRE/% MPE/% MSE/% MPSE/% 树干Stem 0.055 0 2.512 5 0.968 4 14.59 9.03 7.03 6.31 18.47 树皮Bark 0.028 5 1.993 0 0.954 5 1.78 7.70 7.21 6.52 17.34 树枝Branch 0.161 6 1.456 5 0.917 3 1.69 5.91 5.40 5.98 16.81 树叶Leaf 0.235 6 0.905 5 0.842 9 0.49 5.69 4.73 5.69 16.21 树根Under 0.043 4 2.102 0 0.957 7 4.20 10.85 8.01 7.50 17.15 地上Above 0.962 2 18.01 8.41 6.58 6.38 16.74 全株Total 0.963 6 21.02 8.80 6.44 6.53 16.38 注:MAbove=MStem+MBark+MBranch+MLeaf ; MTotal=MAbove+MUnder.指标说明见参考1/2。 -
本研究采用经对数转换的线性回归来构建生物量反演模型[21]。
$ \begin{array}{l} {\rm{ln}}\;{M_{components}} = a + {b_1}\;{\rm{ln}}{x_1} + {b_2}\;{\rm{ln}}{x_2}{ + _{ \ldots \ldots }} + \\ \;\;\;\;\;\;\;\;\;\;\;\;\;\;\;\;\;\;\;\;{b_n}\;{\rm{ln}}{x_n} + \varepsilon \end{array} $
(3) 其中,Mcomponents为全株及各组分生物量,x1, x2, …, xn为LiDAR建模变量,a为常数项,b1, b2, …, bn为回归参数,ε为误差项。
采用逐步回归法(Stepwise)筛选变量,自变量使统计量F值过小并且t检验达不到显著水平(p>0.1),则予以剔除;F值较大且t检验达到显著水平(p < 0.05)则可以进入。重复进行直到回归方程中所有的自变量均符合进入模型的要求,方程外的自变量均不符合进入模型的要求为止。采用自变量相关稀疏矩阵的条件数k检查变量的多重共线性:k < 100,多重共线性程度很小;100 < k < 1 000,共线性程度较强;k>1 000,共线性程度严重。
-
随机森林(RF)作为一种改进的决策树方法,自Leo Breiman提出以来,被广泛地应用于数据挖掘、生物信息学等研究中[22]。
模型变量重要性程度通常使用平均不纯度减少(MDI)或平均精确率减少(MDA)来评估。
平均不纯度减少计算方法为:
$ G{I_m} = 1 - \sum {p^2}_{mk} $
(5) $ VI{M_{jm}} = G{I_m} - G{I_{left}} - G{I_{right}} $
(6) $ VI{M_{gini}} = {\sum ^N}_{i = 1}\left( {{\sum ^M}_{j = 1}VI{M_{jm}}} \right) $
(7) 其中,GIm为基尼指数,pmk为节点m中类别k所占的比例,VIMjm为特征Xj在节点m的重要性,GIleft和GIright分别表示节点分枝后两个新节点的基尼指数,M为节点总数,N为决策树总数。VIMgini为平均不纯度减少方法计算得到的重要性程度。
平均精确率减少计算方法为:
$ VI{M_{err}} = \sum \left( {er{r_{OOB2}} - er{r_{OOB1}}} \right)/N $
(8) 其中,VIMerr为平均精确率减少计算方法得到的重要性程度;errOOB1为每一棵决策树,使用OOB数据计算得到预测值与观测值的差值;errOOB2为随机地对所有OOB样本特征加入噪声干扰,再次计算得到预测值与观测值的差值;N为决策树总数。
使用R软件的Random Forest包进行生物量模型拟合需要确定mtry和ntree两个重要参数,本研究参考Leo Breiman的推荐默认值[22],结合调试结果,最终确定mtry取18,ntree取1 000。
-
采用决定系数(R2)、均方根误差(RMSE)、相对均方根误差(rRMSE)、总相对误差(TRE)作为模型的评价统计指标,指标含义及公式见1/2。本研究拟合模型使用全部数据,采用“刀切法”计算评价统计指标,每次取39个样本拟合,1个样本测试,如此循环直至遍历所有样本,以测试结果的平均值作为反映模型泛化能力的依据。生物量反演模型构建及统计分析均采用R软件完成。
-
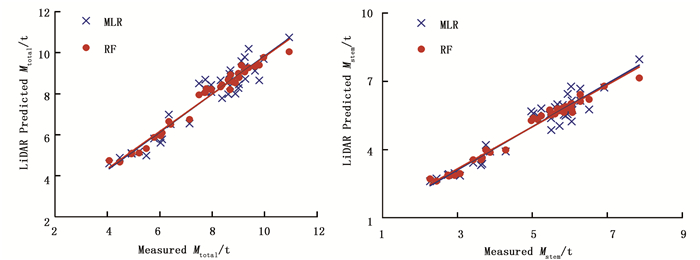
表 3、表 4分别表达了MLR和RF模型拟合的结果,LiDAR变量与生物量有较强的相关性,经逐步回归得到的Hinterval、H80、D10、D20与各组分生物量的相关性普遍表现为显著(P < 0.05)或极显著水平(P < 0.01)。MLR方法构建的生物量模型除了树枝、树叶外,其余组分模型的R2均高于0.82,树干、地上及全株模型的R2均高于0.90;RF方法构建的生物量模型R2均高于0.91,同时拥有较小的rRMSE和TRE值,表明两种方法构建的模型均有较好的拟合效果,可解释的变异占总变异比例较高。
表 3 MLR参数估计值及评价统计指标
Table 3. Parameter estimates and evaluation statistics of MLR
组分
Components参数估计值
Parameter estimates统计指标
Evaluation statisticsa b1 b2 b3 R2 RMSE/t rRMSE TRE/% 树干Stem -2.762 3** -0.632 4** 1.702 8** 11.850 6** 0.914 3 0.409 3 0.077 7 0.60 树皮Bark -3.121 8** -0.882 2** 1.187 9** 10.783 8** 0.825 1 0.048 9 0.081 7 0.66 树枝Branch -0.872 6* -0.788 1** 0.442 0** 7.347 6* 0.541 4 0.069 3 0.096 1 0.91 树叶Leaf 0.114 1 -1.100 8** -0.169 2 5.864 6 0.636 1 0.152 2 0.678 6 0.96 树根Under -2.574 5** -0.597 2** 1.149 7** 8.376 5** 0.873 3 0.083 7 0.068 0.45 地上Above -1.586 2** -0.651 3** 1.403 5** 10.583 6** 0.907 7 0.458 6 0.067 3 0.45 全株Total -1.304 4** -0.641 9** 1.362 6** 10.217 7** 0.905 0 0.537 0 0.066 8 0.45 注:*:P < 0.05;**:P < 0.01。ln MBranch=a0+b1 ln Hinterval+b2 ln H80+b3 ln D20。其余组分模型形式均为:ln Mcomponents=a0+b1 lnHinterval+b2 ln H80+b3 ln D10。 表 4 RF评价统计指标
Table 4. Evaluation statistics of RF
组分Components R2 RMSE/t rRMSE TRE/% 树干Stem 0.973 4 0.536 7 0.107 7 1.066 5 树皮Bark 0.964 8 0.051 4 0.087 5 0.725 9 树枝Branch 0.919 4 0.071 3 0.098 9 0.944 9 树叶Leaf 0.912 8 0.032 5 0.143 0 1.941 5 树根Under 0.964 6 0.104 0 0.086 7 0.713 3 地上Above 0.971 1 0.623 7 0.095 5 0.853 2 全株Total 0.970 6 0.720 6 0.093 2 0.814 7 对比两种方法,RF拥有更好的拟合效果。Mstem、Mabove和Mtotal组分生物量模型方面,RF的R2≈0.97,MLR的R2≈0.91,rRMSE和TRE相近。树枝和树叶模型方面,MLR的R2仅有0.5~0.6,RF则仍维持在0.9以上,表明RF有更好的拟合效果。此外,无论是MLR还是RF,生物量Mtotal>8 t的区间低估情况明显增多(图 1),说明随着林龄的增加,林分郁闭度逐渐降低(自然稀疏或人为间伐),LiDAR点云密度不高的情况下,冠层回波信号不够,容易树顶及树冠边缘错失现象,导致生物量被低估[18]。
图 2表达了RF组分生物量模型中各LiDAR变量归一化后的相对重要性程度。相同的组分模型中,MDI和MDA各变量的相对重要性排序总体趋势基本一致,尤其是前15个变量,重要程度几乎完全一致,即表明两种方法虽然选择指标和算法有所区别,但均是反映自变量对因变量的重要性影响。MDI与MDA相比,各变量的相对重要性表现出更大的差异性,一般前5~10个变量的重要性程度要显著高于其余变量;MDA虽然排序趋势与MDI一致,但各变量重要性程度差异较小,变化平缓。因此,若需要根据相对重要性程度筛选变量优化RF模型,MDI方法在趋势性判断和阈值设定方面更具优势。
从表 5可以看出,4种林分状态下,林分密度、平均高均与生物量有显著的相关性。其中,类型Ⅱ反映了同一水平林分间伐前后的生物量变化情况,表明LiDAR变量能较好地反映林分的现势性,除了能表达林分自然生长变化,也能表达人为干扰的情况。因此,利用LiDAR的高度变量和密度变量进行森林生物量的反演是十分有效的[10]。树枝、树叶的拟合效果较差,一方面因冠层交错导致生物量低估,另一方面可能与外业样本采集中容易造成树枝、树叶生物量的耗损有关[23]。
表 5 不同林分特征的反演效果比较
Table 5. Comparison of inversion effects of different forest characteristics
类型
Types样地号
PlotsMTotal实测值
MTotal Measured Value/tMLR预测值
MLR Predicted Value/tRF预测值/t
RF Predicted Value林分密度
Density/(株·hm-2)平均胸径
D/ cm平均树高
Height/mⅠ 17_02 9.270 2 8.713 9 9.172 4 1 217 18.2 22.2 17_711 7.974 9 8.069 1 8.191 4 547 23.9 22.8 Ⅱ 17_20 4.939 3 5.062 0 5.092 9 1 650 12.4 13.6 17_16 7.144 1 6.534 9 6.740 0 1 950 13.2 13.7 Ⅲ 17_704 10.943 1 10.738 3 10.040 0 420 31.5 28.9 17_12 7.504 5 8.494 7 7.936 3 483 23.7 20.7 Ⅳ 16_F2_1 8.664 2 8.963 7 8.617 2 1 256 17.2 19.3 17_22 4.078 8 4.587 5 4.733 4 1 283 13.0 14.0 类型Ⅰ:相同树高不同密度(高树高水平);类型Ⅱ:相同树高不同密度(低树高水平);类型Ⅲ:相同密度不同树高(低密度水平);类型Ⅳ:相同密度不同树高(高密度水平)。TypeⅠ:Same tree height and different density when tree height is high level; TypeⅡ:Same tree height and different density when tree height is low level; TypeⅢ:Same density and different tree height when density is low level; TypeⅣ:Same density and different tree height when density is high level. -
LiDAR变量与组分生物量均有显著的相关性,构建的模型具有较好的拟合效果和较高的预估精度,R2普遍高于0.82,树干、地上和全株等组分生物量模型的R2更是优于0.90。利用“刀切法”对模型的泛化能力进行评价,rRMSE值介于6%10%,TRE值介于0.4%1.0%,结果令人满意。这一方面由于LiDAR数据能高精度提取林分结构参数,有利于生物量反演精度提升;另一方面可能与研究区落叶松全部为人工林有关,林相整齐、均质度高,LiDAR数据能较好地代表林分整体状态[14]。
比较而言,RF拥有更好的拟合效果。利用“刀切法”评价RF模型的泛化能力,与穆喜云等[24]的研究结论一致。这一方面归功于RF自身强大的学习能力;另一方面,与森林生物量与LiDAR数据之间复杂的非线性关系有关[25]。MLR筛选出的变量Hinterval、H80、D10、D20与各组分生物量的相关性普遍表现为显著(P < 0.05)或极显著水平(P < 0.01),说明LiDAR变量拥有对组分生物量很好的解释性[15]。虽然RF的变量相对重要性排序也可以表达出各变量的贡献度,但由于LiDAR变量自身拥有的多重相关性,导致各组分生物量模型的变量相对重要性排序差异较大,下一步研究中,应当尝试深入分析变量相量相对重要性程度,并通过交互进一步优化RF模型。
-
本研究结果表明:
(1) 机载LiDAR提取的自变量与生物量均具有显著的相关性,普遍表现为显著(P < 0.05)或极显著水平(P < 0.01)。利用LiDAR变量构建的生物量反演模型能够较好地反映林分的现势特征,除了能表达林分的自然生长过程,还能很好的体现人为干扰的信息。
(2) MLR与RF方法均适用于生物量反演模型构建,除了树枝、树叶外,模型的R2均高于0.8。其中,MLR方法优势是模型更加简单明确,LiDAR自变量对生物量有更明确的解释能力。RF模型拥有更好的拟合效果和预估精度。
(3)低林分密度水平下,由于LiDAR点云与冠顶的错失机率增大,会出现对生物量被低估的现象,且这一情况会随着林龄的增加而逐步增大。
基于机载激光雷达的落叶松组分生物量反演
Inversion of Biomass Components for Larix olgensis Plantation Using Airborne LiDAR
-
摘要:
目的 构建基于机载LiDAR的落叶松组分生物量反演模型,讨论不同方法对模型构建的影响。 方法 以地面实测样地数据和同步获取的机载LiDAR点云数据为数据源,分别采用多元线性回归(MLR)和随机森林(RF)方法,估测了长白落叶松的组分生物量,利用"刀切法"评价了模型的泛化能力。 结果 表明:(1)MLR筛选得到的Hinterval、H80、D10、D20与各组分生物量普遍表现为显著(P < 0.05)或极显著水平(P < 0.01)。(2)MLR模型的R2高于0.82(枝、叶除外);RF模型的R2均高于0.91,且均拥有较小rRMSE、TRE值。(3)MDI和MDA方法的变量相对重要值排序均能较好地体现LiDAR变量与生物量之间的关系,MDI在趋势性判断和阈值设定方面更具优势。 结论 LiDAR变量与组分生物量具有显著的相关性。RF拥有更好的拟合效果和泛化能力,MLR则对LiDAR和组分生物量的关系有更明确的解释能力。反演模型能较好地反映林分的现势特征,生物量被低估的现象会随着林龄的增加而逐步增多。 Abstract:Objective To establish the inversion models of biomass components for Larix olgensis plantation using Airborne LiDAR. Method Compatible models were established by combining dummy variable and nonlinear seemingly unrelated regression based on the biomass data of 64 trees in 40 sample plots of L. olgensis plantation. The canopy height indices and density indices were calculated from LiDAR point cloud data. Then the models based on these biomass components from field data and LiDAR indices were built using multivariate linear regression(MLR) and random forest regression (RF). The biomass model validation was accomplished by Jackknifing technique. Result (1) There was a significant (P < 0.05) or extremely significant (P < 0.01) correlation between biomass components and Hinterval, H80, D10, and D20 which were screened using MLR. (2) The models had good estimation precision with R2>0.82 and R2>0.91 for MLR and RF, respectively. (3) The relationship between LiDAR variables and biomass could be reflected well by relative importance ranking of variables by both MDI and MDA method. Conclusion There is a good correlations between biomass components and LiDAR indices. RF shows stronger ability about fitting goodness and generalization, while MLR can make clearer interpretation of the relationship between LiDAR indices and biomass components. The present situation of L. olgensis plantation can be accurately reflected using inversion models, and the underestimation of component biomass will increase with the increase of stand age. -
Key words:
- biomass inversion
- / airborne LiDAR
- / biomass component
- / Larix olgensis
-
表 1 样木数据概况
Table 1. Summary statistics of sampling trees
林龄
Ages/a样木数量
Numbers胸径
D/cm树高
Height/m树干
Stem/kg树皮
Bark/kg树枝
Branch/kg树叶
Leaf/kg树根
Under/kg幼龄林(419)
Young stand16 3.49.2 3.713.8 1.715.9 0.32.9 0.74.8 0.62.0 0.85.2 中龄林(2129)
Middle-age stand16 9.213.1 6.517.1 10.148.6 2.17.1 3.79.0 1.53.1 4.711.9 近熟林(3139)
Near-mature stand16 12.916.7 11.318.4 26.9106.4 4.012.3 5.412.3 1.83.5 7.420.1 成熟林(4155)
Mature stand16 17.223.0 11.720.8 64.0185.7 7.919.2 8.719.4 2.55.6 15.849.9 表 2 模型参数估计值及评价统计指标
Table 2. Parameter estimates and evaluation statistics of biomass model
组分
Components参数估计值
Parameter estimates统计指标
Evaluation statisticsa b Radj2 SEE/t TRE/% MPE/% MSE/% MPSE/% 树干Stem 0.055 0 2.512 5 0.968 4 14.59 9.03 7.03 6.31 18.47 树皮Bark 0.028 5 1.993 0 0.954 5 1.78 7.70 7.21 6.52 17.34 树枝Branch 0.161 6 1.456 5 0.917 3 1.69 5.91 5.40 5.98 16.81 树叶Leaf 0.235 6 0.905 5 0.842 9 0.49 5.69 4.73 5.69 16.21 树根Under 0.043 4 2.102 0 0.957 7 4.20 10.85 8.01 7.50 17.15 地上Above 0.962 2 18.01 8.41 6.58 6.38 16.74 全株Total 0.963 6 21.02 8.80 6.44 6.53 16.38 注:MAbove=MStem+MBark+MBranch+MLeaf ; MTotal=MAbove+MUnder.指标说明见参考1/2。 表 3 MLR参数估计值及评价统计指标
Table 3. Parameter estimates and evaluation statistics of MLR
组分
Components参数估计值
Parameter estimates统计指标
Evaluation statisticsa b1 b2 b3 R2 RMSE/t rRMSE TRE/% 树干Stem -2.762 3** -0.632 4** 1.702 8** 11.850 6** 0.914 3 0.409 3 0.077 7 0.60 树皮Bark -3.121 8** -0.882 2** 1.187 9** 10.783 8** 0.825 1 0.048 9 0.081 7 0.66 树枝Branch -0.872 6* -0.788 1** 0.442 0** 7.347 6* 0.541 4 0.069 3 0.096 1 0.91 树叶Leaf 0.114 1 -1.100 8** -0.169 2 5.864 6 0.636 1 0.152 2 0.678 6 0.96 树根Under -2.574 5** -0.597 2** 1.149 7** 8.376 5** 0.873 3 0.083 7 0.068 0.45 地上Above -1.586 2** -0.651 3** 1.403 5** 10.583 6** 0.907 7 0.458 6 0.067 3 0.45 全株Total -1.304 4** -0.641 9** 1.362 6** 10.217 7** 0.905 0 0.537 0 0.066 8 0.45 注:*:P < 0.05;**:P < 0.01。ln MBranch=a0+b1 ln Hinterval+b2 ln H80+b3 ln D20。其余组分模型形式均为:ln Mcomponents=a0+b1 lnHinterval+b2 ln H80+b3 ln D10。 表 4 RF评价统计指标
Table 4. Evaluation statistics of RF
组分Components R2 RMSE/t rRMSE TRE/% 树干Stem 0.973 4 0.536 7 0.107 7 1.066 5 树皮Bark 0.964 8 0.051 4 0.087 5 0.725 9 树枝Branch 0.919 4 0.071 3 0.098 9 0.944 9 树叶Leaf 0.912 8 0.032 5 0.143 0 1.941 5 树根Under 0.964 6 0.104 0 0.086 7 0.713 3 地上Above 0.971 1 0.623 7 0.095 5 0.853 2 全株Total 0.970 6 0.720 6 0.093 2 0.814 7 表 5 不同林分特征的反演效果比较
Table 5. Comparison of inversion effects of different forest characteristics
类型
Types样地号
PlotsMTotal实测值
MTotal Measured Value/tMLR预测值
MLR Predicted Value/tRF预测值/t
RF Predicted Value林分密度
Density/(株·hm-2)平均胸径
D/ cm平均树高
Height/mⅠ 17_02 9.270 2 8.713 9 9.172 4 1 217 18.2 22.2 17_711 7.974 9 8.069 1 8.191 4 547 23.9 22.8 Ⅱ 17_20 4.939 3 5.062 0 5.092 9 1 650 12.4 13.6 17_16 7.144 1 6.534 9 6.740 0 1 950 13.2 13.7 Ⅲ 17_704 10.943 1 10.738 3 10.040 0 420 31.5 28.9 17_12 7.504 5 8.494 7 7.936 3 483 23.7 20.7 Ⅳ 16_F2_1 8.664 2 8.963 7 8.617 2 1 256 17.2 19.3 17_22 4.078 8 4.587 5 4.733 4 1 283 13.0 14.0 类型Ⅰ:相同树高不同密度(高树高水平);类型Ⅱ:相同树高不同密度(低树高水平);类型Ⅲ:相同密度不同树高(低密度水平);类型Ⅳ:相同密度不同树高(高密度水平)。TypeⅠ:Same tree height and different density when tree height is high level; TypeⅡ:Same tree height and different density when tree height is low level; TypeⅢ:Same density and different tree height when density is low level; TypeⅣ:Same density and different tree height when density is high level. -
[1] Lambert M, Ung C, Raulier F. Canadian national tree aboveground biomass equations[J]. Canadian Journal of Forest Research, 2005, 35(8):1996-2018. doi: 10.1139/x05-112 [2] 沈亚洲, 孙晓梅, 张江涛, 等.甘肃小陇山林区日本落叶松人工林单株生物量的研究[J].林业科学研究, 2011, 24(4):517-522. [3] 曾伟生, 唐守正, 夏忠胜, 等.利用线性混合模型和哑变量模型方法建立贵州省通用性生物量方程[J].林业科学研究, 2011, 24(3):285-291. [4] Haara A, Leskinen P. The assessment of the uncertainty of updated stand-level inventory data[J]. Silva Fennica, 2009, 43(1):87-112. [5] 徐天蜀, 岳彩荣.基于印度遥感卫星IRS-P6的森林生物量估测模型研究[J].中南林业调查规划, 2008, 27(1):42-45. doi: 10.3969/j.issn.1003-6075.2008.01.012 [6] 郑光, 田庆久, 陈镜明, 等.结合树龄信息的遥感森林生态系统生物量制图[J].遥感学报, 2006, 10(6):932-940. [7] Smith-Jonforsen G, Folkesson K, Hallberg B, et al. Effects of Forest Biomass and Stand Consolidation on P-Band Backscatter[J]. IEEE Geoscience and Remote Sensing Letters, 2007, 4(4):669-673. doi: 10.1109/LGRS.2007.905120 [8] Paloscia S, Macelloni G, Pampaloni P, et al. The potential of C- and L-band SAR in estimating vegetation biomass:the ERS-1 and JERS-1 experiments[J]. IEEE Transactions on Geoscience and Remote Sensing, 1999, 37(4):2107-2110. doi: 10.1109/36.774723 [9] 曹庆先, 徐大平, 鞠洪波.基于TM影像纹理与光谱特征和KNN方法估算5种红树林群落生物量[J].林业科学研究, 2011, 24(2):144-150. [10] Næsset E, Gobakken T. Estimation of above- and below-ground biomass across regions of the boreal forest zone using airborne laser[J]. Remote Sensing of Environment, 2008, 112(6):3079-3090. doi: 10.1016/j.rse.2008.03.004 [11] 刘清旺, 李增元, 陈尔学, 等.机载LIDAR点云数据估测单株木生物量[J].高技术通讯, 2010, 20(7):765-770. doi: 10.3772/j.issn.1002-0470.2010.07.019 [12] 曹林, 佘光辉, 代劲松, 等.激光雷达技术估测森林生物量的研究现状及展望[J].南京林业大学学报:自然科学版, 2013, 37(3):163-169. [13] 尹惠妍, 李海奎.基于蓄积的森林生物量估算方法的对比分析[J].林业科学研究, 2014, 27(6):848-853. [14] 庞勇, 李增元.基于机载激光雷达的小兴安岭温带森林组分生物量反演[J].植物生态学报, 2012, 36(10):1095-1105. [15] 刘茜, 杨乐, 柳钦火, 等.森林地上生物量遥感反演方法综述[J].遥感学报, 2015, 19(1):62-74. [16] Yong P, Li Z, Ju H, et al. LiCHy:The CAF's LiDAR, CCD and hyperspectral integrated airborne observation system[J]. Remote Sensing, 2016, 8(5):398-414. doi: 10.3390/rs8050398 [17] Zhao X, Guo Q, Su Y, et al. Improved progressive TIN densification filtering algorithm for airborne LiDAR data in forested areas[J]. ISPRS Journal of Photogrammetry and Remote Sensing, 2016, 117:79-91. doi: 10.1016/j.isprsjprs.2016.03.016 [18] 李增元, 刘清旺, 庞勇.激光雷达森林参数反演研究进展[J].遥感学报, 2016, 20(5):1138-1150. [19] 莫德祥.桂东南不同密度柳杉人工林生态系统生物量及碳氮格局研究[D].南宁: 广西大学, 2013. [20] 曾伟生, 唐守正.立木生物量方程的优度评价和精度分析[J].林业科学, 2011, 47(11):106-113. doi: 10.11707/j.1001-7488.20111117 [21] 穆喜云.森林地上生物量遥感估测方法研究[D].呼和浩特: 内蒙古农业大学, 2015. [22] Breiman L. Random forests[J]. Machine Learning, 2001, 45:5-32. doi: 10.1023/A:1010933404324 [23] 黄兴召, 孙晓梅, 张守攻, 等.辽东山区日本落叶松生物量相容性模型的研究[J].林业科学研究, 2014, 27(2):142-148. [24] 穆喜云, 刘清旺, 庞勇, 等.基于机载激光雷达的森林地上碳储量估测[J].东北林业大学学报, 2016, 44(11):52-56. doi: 10.3969/j.issn.1000-5382.2016.11.011 [25] Chen Q. Modeling aboveground tree woody biomass using national-scale allometric methods and airborne lidar[J]. ISPRS Journal of Photogrammetry and Remote Sensing, 2015, 106(3):95-106. -





 下载:
下载: