-
杨树(Populus L.)具有速生、易繁殖、适应性强和生产力高等特性,在木材加工、碳汇造林、制浆造纸和生物燃料等方面发挥着重要作用[1]。杨树提供了大量木材,但是杨树人工林土壤氮素缺乏导致其木材产量受到严重制约[2-4],因此选育高产量的杨树品种具有重要意义。作物育种的关键步骤是选择,而常规育种是以表型选择为基础的。基于亲本杂交和后代表型选择的传统育种费时费力,挖掘和公布一个新的品种需要至少10 a以上的时间。基因组选择(Genomic selection, GS)也被称为基因组预测,通过在早期阶段淘汰潜力较小的个体来降低育种成本[5-7]。基因组选择现在被广泛用于数量性状的遗传改良,GS可以减少植物育种中表型观察所需的成本和工作量[8]。通过使用详细的基因组信息揭示一个基因型的基因优势,可以使农业和林业生产发生革命性的变化[9]。
GS的预测准确度影响因素包括群体的大小、群体结构、亲缘关系、分子标记、表型的精度、目标性状的遗传力和统计模型等[10-11]。分子标记的数量和密度影响基因组预测的准确度和效率,因此需要足够的分子标记并且选择适合模型才能得到准确的育种值[12]。统计模型在GS研究中具有重要地位,其对表型和基因型数据的训练效果决定着标记效应是否估计准确,进而对后续的育种计划产生影响[13]。采用固定和随机效应的混合线性模型(Mixed linear model,MLM)直接预测个体的遗传优点,被称为最佳线性无偏预测(Best Linear Unbiased Prediction,BLUP)。VanRaden借助BLUP于2008年提出了基于G矩阵的gBLUP(Genomic BLUP)方法[14]。目前gBLUP已经广泛应用于动植物育种研究中,并且因为它的高效、稳健等优点,现在仍饱受青睐。Wang等人基于gBLUP,压缩个体分成不同的组构建了cBLUP (Compressed BLUP)模型和区段化标记(bin 标记)构建了sBLUP(SUPER BLUP)模型[15]。
育种计划的成功取决于对遗传参数的精确估计,包括对育种值的可靠预测[16-17]。育种值是遗传效应对该性状表型值的加性效应[18],它消除了环境影响,反映了真实的遗传效应,提高了选择的准确性。通过估算亲本和杂交后代的育种值进行基因型选择可以代替表型选择,从而提高选择的效率和准确性。育种值的估算对重要造林树种的遗传改良起到了重要作用。有效地建立基因型-表型关系,以便作出可靠的预测,指导探索巨大的遗传选择空间。对于杂交作物来说尤其如此,因为潜在杂交品种的数量太高,无法进行广泛的测试。由于GS在提高动物育种遗传收益方面取得的巨大成功,因此GS被引入到植物育种研究的许多方面,如自交系性能预测、亲本选择和杂交预测[19-20]。利用GS对植物的重要的经济性状进行预测育种值,加快育种计划具有重要的意义。目前在杨树的经济相关性状的全基因组选择方面的研究十分欠缺,亟须展开基因组选择相关的研究工作。
在F1杂交育种中,随着自交系数量的增加,需要测试的亲本组合数量呈指数增长。因此,利用GS对有杂种优势的F1代进行预选,可以实现高效育种。本研究对高氮和低氮环境下的地径、株高和茎生物量等性状进行全基因组选择研究,利用3个全基因组选择模型(gBLUP、sBLUP、cBLUP)和已经观测364个基因型的表型观测值(包含2个亲本和362个杂交F1代)对502个基因型进行预测育种值,为杨树遗传育种工作奠定基础。
-
美洲黑杨丹红杨(Populus deltoides ‘Danhong’)具有速生和干形通直等优良特性[21-22]。青杨派小叶杨优树通辽1号杨(Populus simonii ‘Tongliao1’)具有抗旱、抗冻和抗病虫害等特点,但是生长缓慢[23-24]。以丹红杨为母本,通辽1号杨为父本的F1群体种植于中国林业科学研究院试验田,包括2个亲本和500个杂交F1代。于2020年4月采集亲本及杂交子代1年生枝条进行扦插繁殖,5月选择生长一致的杨树幼苗移栽大田。田间试验采用随机区组设计,设施氮肥处理(与对照相比定义为高氮条件)和对照为不施氮肥处理(与处理组相比定义为低氮条件)2个区组,种植株行距为30 cm × 50 cm。6月、7月和8月在高氮处理区每株追施尿素(CON2H4,含氮量46.0%) 4 g。干旱季节和雨后需要正常灌溉和除草。试验设计了两个处理条件,3次生物学重复,364个基因型(包括2个父母和362个F1代),每次重复3株幼苗,共计6 552棵树。
-
11月份杨树生长季节结束后进行所有杂交后代表型测定。地径:利用电子卡尺在根基部以上5 cm处,从垂直的两个方向测定地径;株高:从茎基部5 cm处测量苗高;茎生物量:茎砍伐后自然风干,称取茎的质量。
两个环境中的F1种群的广义遗传力(H)计算公式如下:
$ H=\frac{{V}_{\mathrm{g}}}{{V}_{\mathrm{g}} + \frac{{V}_{\mathrm{e}}}{L}} $
Vg代表遗传方差,Ve代表残差,L代表环境的个数。
-
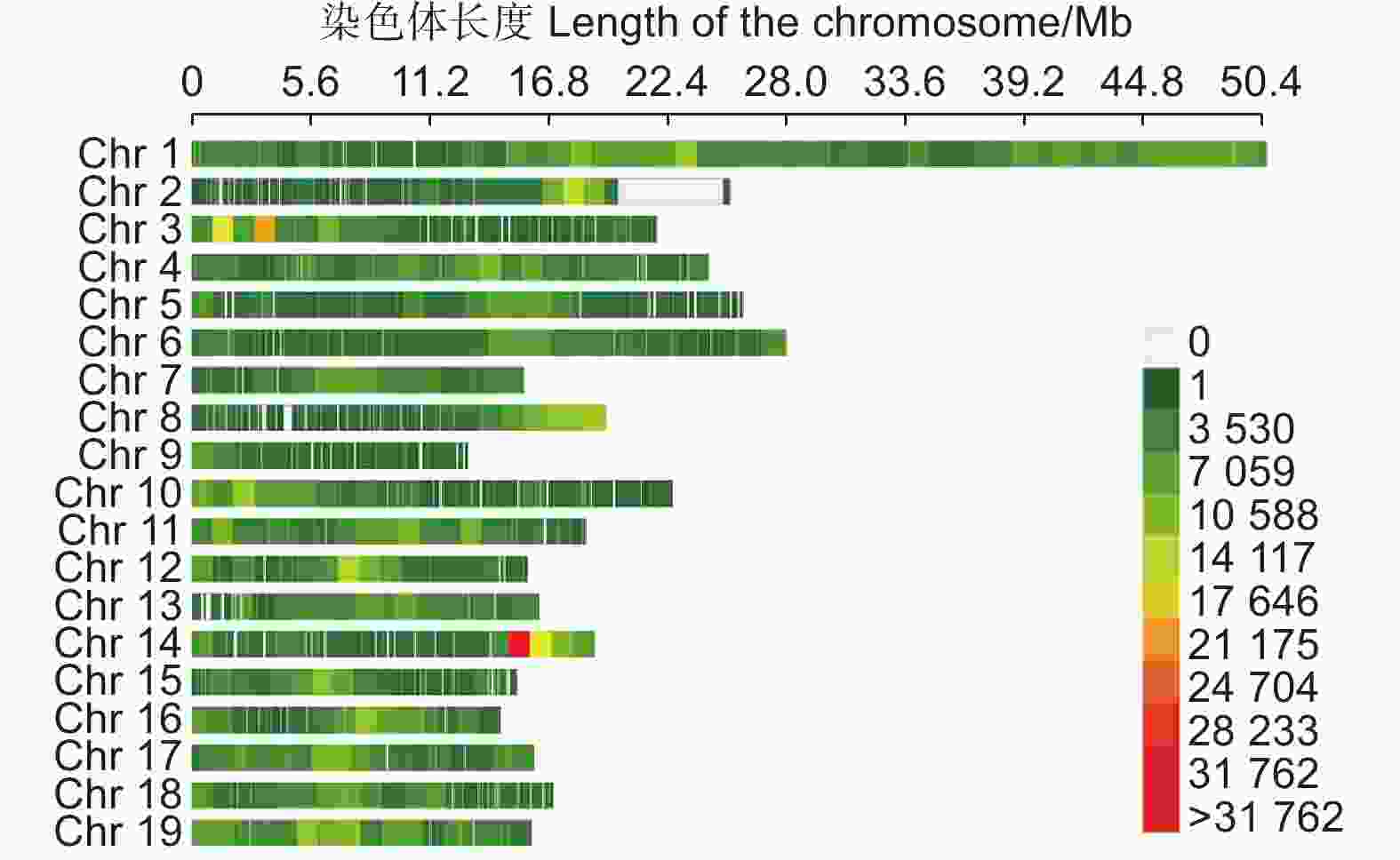
全基因组重测序数据来自于2个亲本和500个杂交群体[23]。毛果杨(Populus trichocarpa Torr. & Gray)基因组V3.1作为参考基因组。对SNP(Single Nucleotide Polymorphism, SNP)位点进行过滤,以确定标记缺失率<10%,次要等位基因频率(MAF)>5%。为了获得独立的SNP标记,根据LD值进行过滤,窗口为50 kb,步长为2个SNP,R2阈值为0.7。最终保留总共1 447 341个高质量的SNP用于GS分析。通过TASSAL5.0软件对502基因型的过滤后的重测序数据进行主成分分析(Principal component analysis,PCA),利用R软件ggplot2包绘制PCA的散点分布图。
-
统计模型是GS的核心,极大地影响了基因组预测的准确度和效率。利用gBLUP、cBLUP和sBLUP模型进行GS分析。通过 R软件的GAPIT3包进行3个模型的基因组选择分析[25]。
-
gBLUP模型公式如下:
$ y=Xb + Zg + e $
y是表型向量,X是固定效应系数关联矩阵,b是固定效应,Z是随机加性遗传效应的关联矩阵,g是随机加性遗传效应,e是残差向量。
混合模型方程组如下:
$ \left[\begin{array}{c}X\;{'}X\\ Z\;{'}X\end{array}\begin{array}{c}X\;{'}Z\\ Z\;{'}Z + k{G}^{-1}\end{array}\right]\left[\begin{array}{c}\breve{b}\\ \breve{g}\end{array}\right]=\left[\begin{array}{c}X\;{'}{y}^{*}\\ Z\;{'}{y}^{*}\end{array}\right] $
其中,
$ k={\sigma }_{e}^{2}/{\sigma }_{\mu }^{2} $ ,G阵是基因组关系矩阵,计算模型如下:$ G=\frac{(M-P)(M-P)\;{'}}{2{\sum }_{i=1}^{m}{p}_{i}(1-{p}_{i})} $
其中,m是标记数目,M是个体基因型信息矩阵。Pi是第i位点的第二等位基因频率。P矩阵是按照每个位点的第二等位基因频率减去0.5然后乘以2规则构建。
-
cBLUP[15]由相应的全基因组关联分析(Genome-wide association study, GWAS)方法压缩混合线性模型(Compressed mixed linear model, CMLM)开发而来。sBLUP[15]由相应的SUPER GWAS方法开发而来。
-
育种值的准确性是基因组预测育种值(GEBV)和真实的育种值(True Breeding Values, TBV或观测值)的相关系数,计算公式为
$ r=\frac{GEBV}{TBV} $
-
所有数据经过excel、R语言和SPSS软件进行统计分析和相关性分析,并且作图。
-
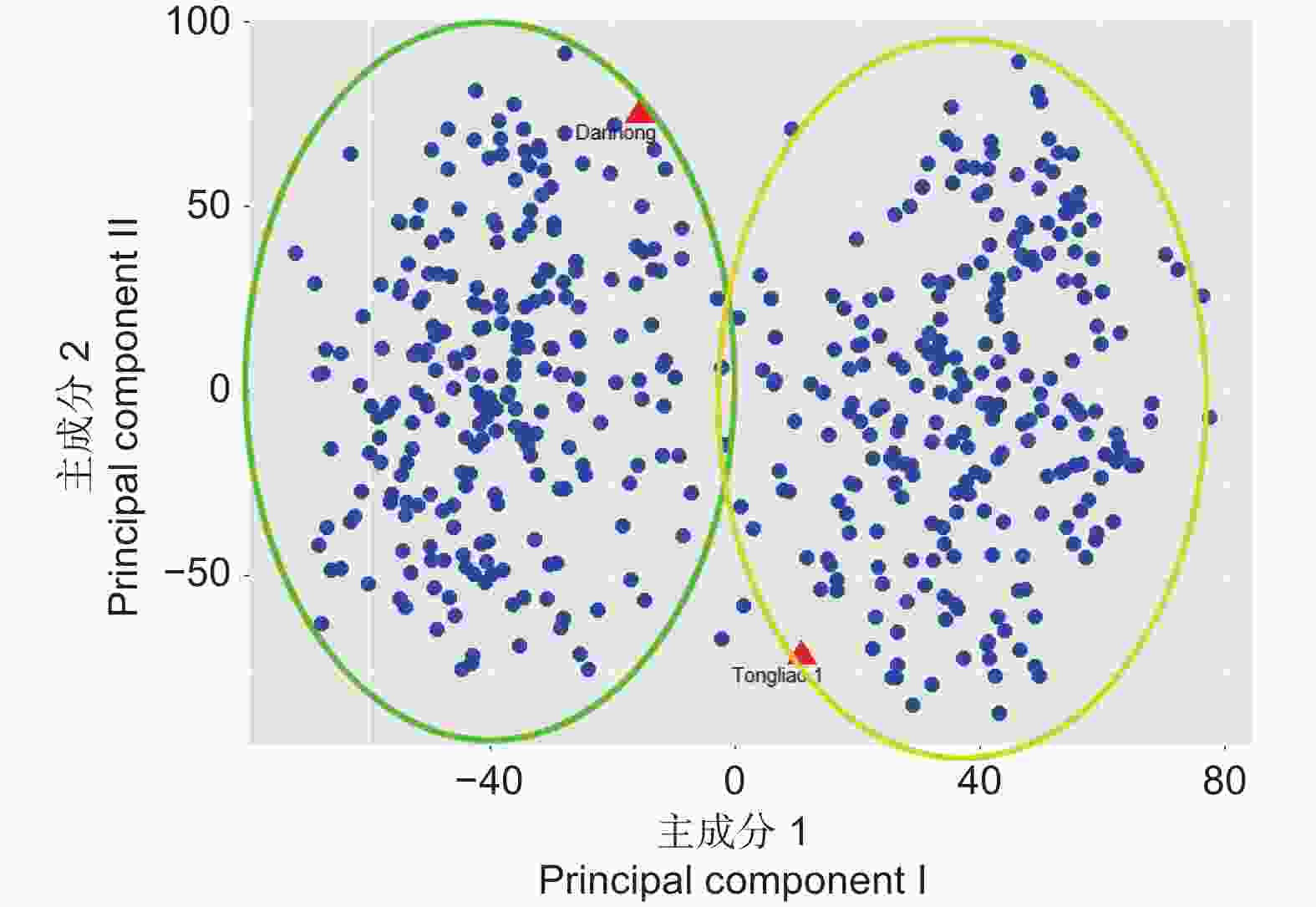
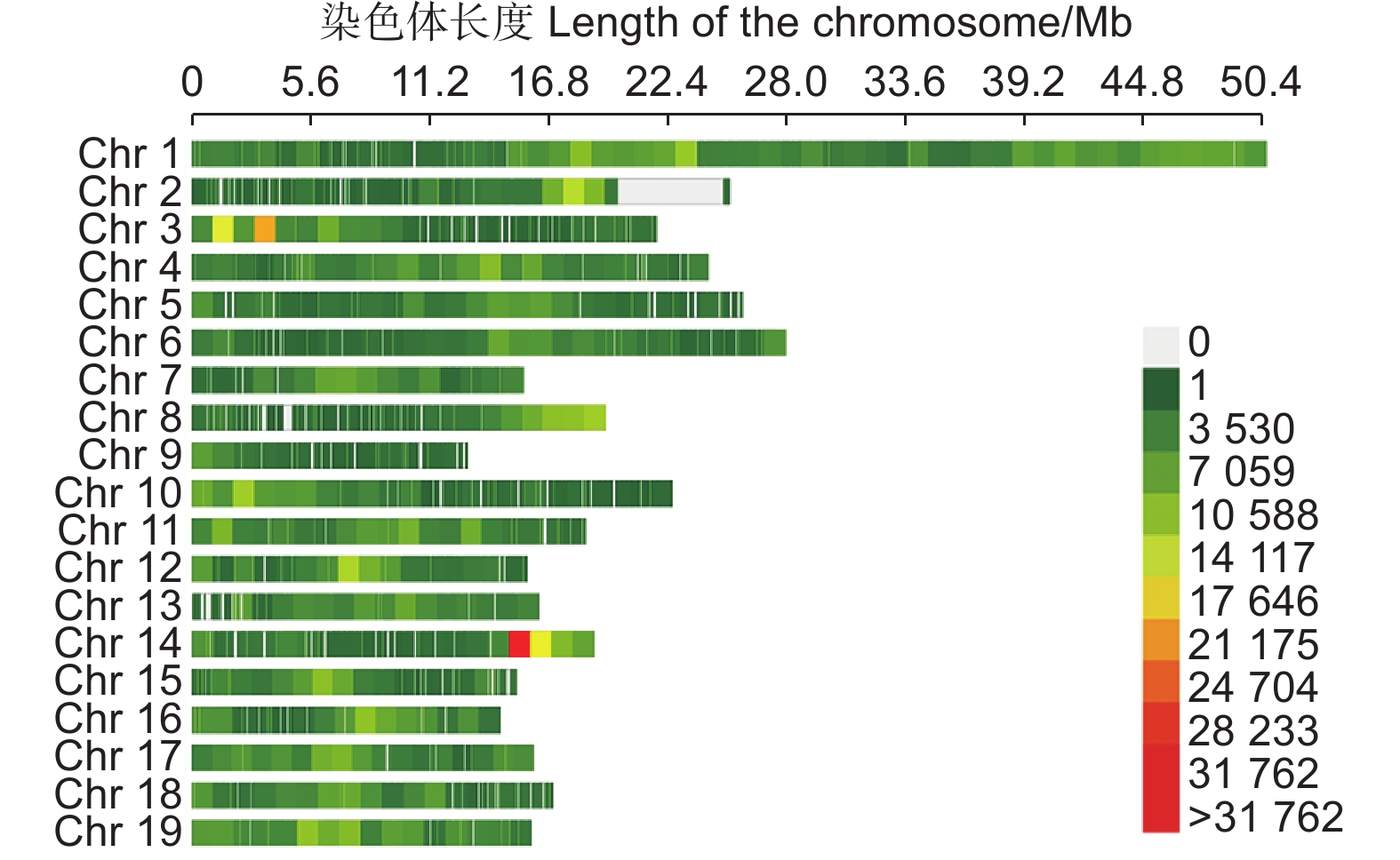
前期试验获得了2个亲本和500个杂交F1代的全基因组重测序数据[23]。重测序数据经过过滤后,获得了1 447 341个高质量的SNPs,均匀分布在19个染色体上(图1)。对500个杂交群体和2个父母的SNP数据进行PCA分析。结果可以看出丹红杨和通辽1号杨的差异较大,杂交群体可以分为2个亚群体。一个亚群偏向于丹红杨,一个亚群偏向于通辽1号杨(图2)。
-
丹红杨的地径在高氮和低氮条件下分别比通辽1号杨提高2.2倍和2.9倍。丹红杨的株高在高氮和低氮条件下分别比通辽1号杨提高了1.8倍和2.5倍。丹红杨的茎生物量在高氮和低氮条件下分别比通辽1号杨提高了20倍和33倍。结果说明丹红杨的生长表型在不同氮环境下显著高于通辽1号杨。在夏皮罗-威尔克检验中(表1),杂交群体中3个性状的W检验值范围为0.96~0.99,接近1,说明表型数据符合正态分布。在高氮和低氮环境下,杂交群体的地径、株高和茎生物量的变异系数在0.13~0.42之间。氮素利用率相关性状的变异系数表明F1群体具有丰富的遗传变异和选择潜力。地径、株高和茎生物量的遗传力分别为0.72、0.70和0.70(表1)。
表 1 杨树杂交群体表型性状观测值的统计分析
Table 1. Statistical analysis of phenotypic traits of poplar hybrid populations
性状
Traits处理条件
Condition亲本
Parents杂交后代
F1 populations丹红杨
Danhong通辽1号杨
Tongliao1最小值
min最大值
max均值
mean变异系数
Coefficient of variation正态性检验
norm test W遗传力
Heritability地径
Ground diameter/mm高氮 High N 25.2** 7.8 10.0 24.8 18.4 0.15 0.99 0.72 低氮 Low N 24.1** 6.2 6.7 22.7 15.6 0.18 0.99 株高
Plant height/cm高氮 High N 350.0** 126.7 148.0 365.0 293.6 0.13 0.96 0.70 低氮 Low N 335.6** 97.0 142.0 366.7 269.4 0.17 0.98 茎生物量
Stem biomass/g高氮 High N 288.3** 13.6 20.8 304.5 146.7 0.40 0.97 0.70 低氮 Low N 244.9** 7.3 17.8 253.9 108.9 0.42 0.97 注:*, ** 分别表示丹红杨和通辽1号杨之间的差异水平P < 0.05 和 P < 0.01
Notes: *, ** significant difference between two parents' levels of P < 0.05 and P < 0.01, respectively -
在502个基因型中包括具有田间测定表型值的364个基因型和没有测得表型的138个基因型。利用cBLUP、gBLUP、sBLUP模型对杂交群体在高氮条件下的地径、株高和茎生物量进行了全基因组预测育种值(图3)。结果表明cBLUP模型对地径、株高和茎生物量的预测的准确率分别为0.139、0.012、0.001。gBLUP模型对地径、株高和茎生物量的预测的准确率分别为0.990、0.987、0.990。sBLUP模型对地径、株高和茎生物量的预测的准确率分别为0.544、0.803、0.829。结果说明gBLUP预测结果最准确接近于1,而cBLUP预测结果的准确性最低。
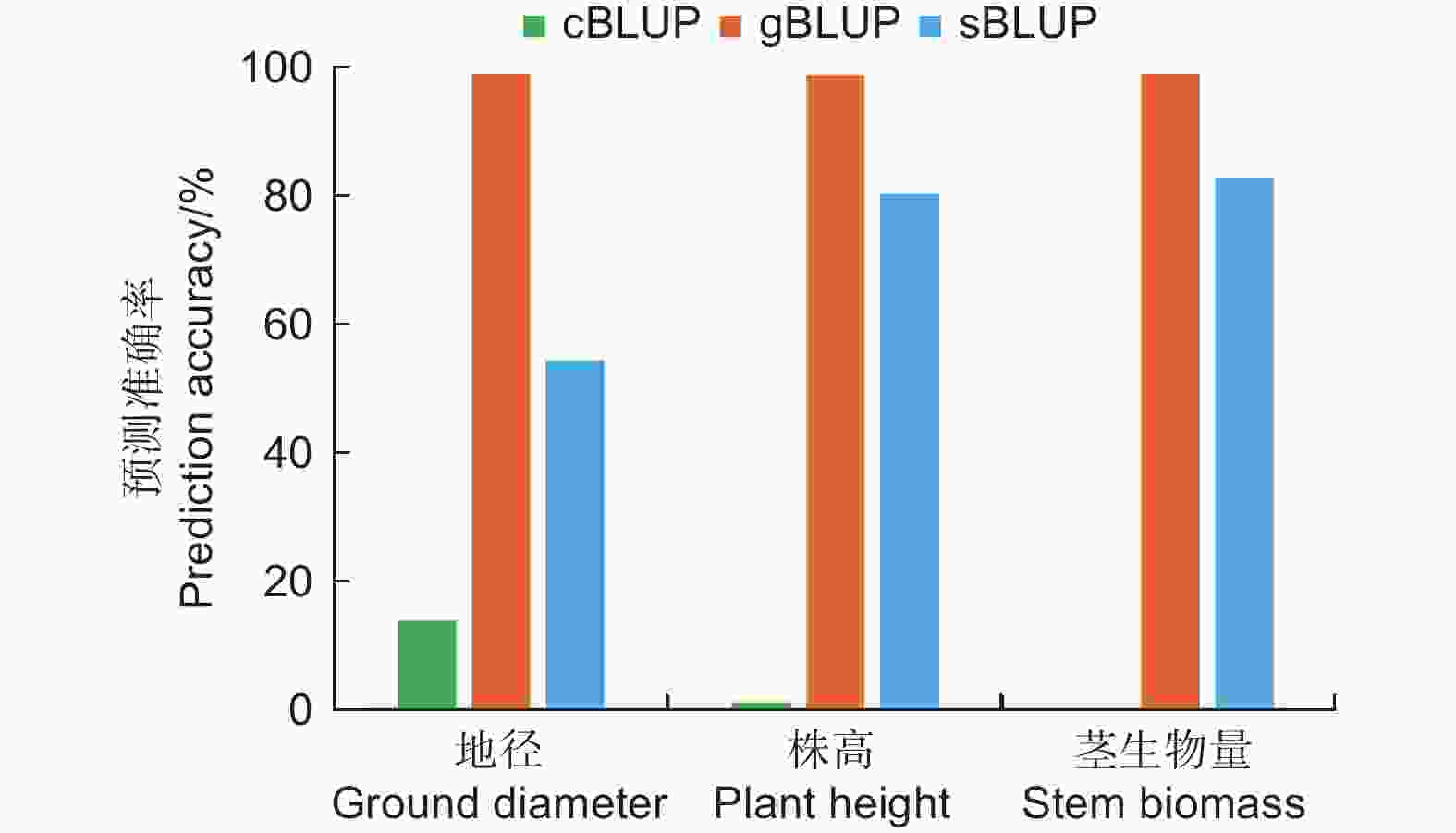
图 3 不同模型计算高氮环境下表型性状育种值的预测准确率比较分析
Figure 3. Comparative analysis of prediction accuracy of phenotypic traits breeding value under high nitrogen environment calculated by different models
364个基因型的观测值TBV和cBLUP、gBLUP、sBLUP计算的地径的均值分别为17.91、17.94、18.28、18.08;株高的均值分别为292.28、293.75、297.28、293.68;茎生物量的均值分别为144.61、144.61、144.61、144.61(表2)。结果说明3个模型计算的育种值的均值和观测值的均值差异较小。观测值TBV和cBLUP、gBLUP、sBLUP计算的地径的方差分别为2.96、0.42、2.90、1.76;株高方差分别为69.89、23.05、59.80、40.59;茎生物量的方差分别为58.45、19.49、58.45、42.89(表2)。通过方差的比较分析,可以看出cBLUP模型计算的方差值远小于观测值的方差值。
表 2 高氮环境下观测值和育种值的统计分析
Table 2. Statistical analysis of observed value and breeding value under a high nitrogen environment
性状
Traits模型
Model均值
Mean方差
SD最小值
Min最大值
Max偏度
Skew峰度
Kurtosis标准误
SE地径/mm
Ground diameterTBV 17.91 2.96 7.84 25.19 −0.13 −0.03 0.16 cBLUP 17.94 0.42 16.96 18.86 0.06 −0.55 0.03 gBLUP 18.28 2.90 7.84 25.19 −0.21 0.34 0.20 sBLUP 18.08 1.76 13.60 24.55 0.11 0.25 0.12 株高/cm
Plant heightTBV 292.28 69.89 126.67 1131.67 7.17 79.70 3.77 cBLUP 293.75 23.05 251.53 571.89 8.28 98.29 1.59 gBLUP 297.28 59.80 169.55 896.01 6.71 61.19 4.13 sBLUP 293.68 40.59 250.34 675.52 7.90 70.27 2.80 茎生物量/g
Stem biomessTBV 144.61 58.45 13.60 304.53 0.52 −0.09 4.03 cBLUP 144.61 19.49 91.55 203.39 0.45 0.00 1.35 gBLUP 144.61 58.45 13.60 304.53 0.52 −0.09 4.03 sBLUP 144.61 42.89 72.41 347.31 1.01 1.88 2.96 注:364个基因型的数据比较结果。TBV为观测值,cBLUP、gBLUP、sBLUP分别为3个模型计算的育种值
Notes: Data comparison results of 364 genotypes. TBV is the observed value, and cBLUP, gBLUP, and sBLUP are the breeding values calculated by the three models -
群体试验在单一环境下进行,受环境因素影响的表型数据不稳定,在不同的环境下鉴定表型性状的育种值更具有稳定性。图4所示,cBLUP模型对低氮条件下的地径、株高和茎生物量的预测的准确率分别为0.108、0.052、0.055;gBLUP模型预测的准确率分别为0.985、0.991、0.990;sBLUP模型准确率分别为0.574、0.590、0.777。
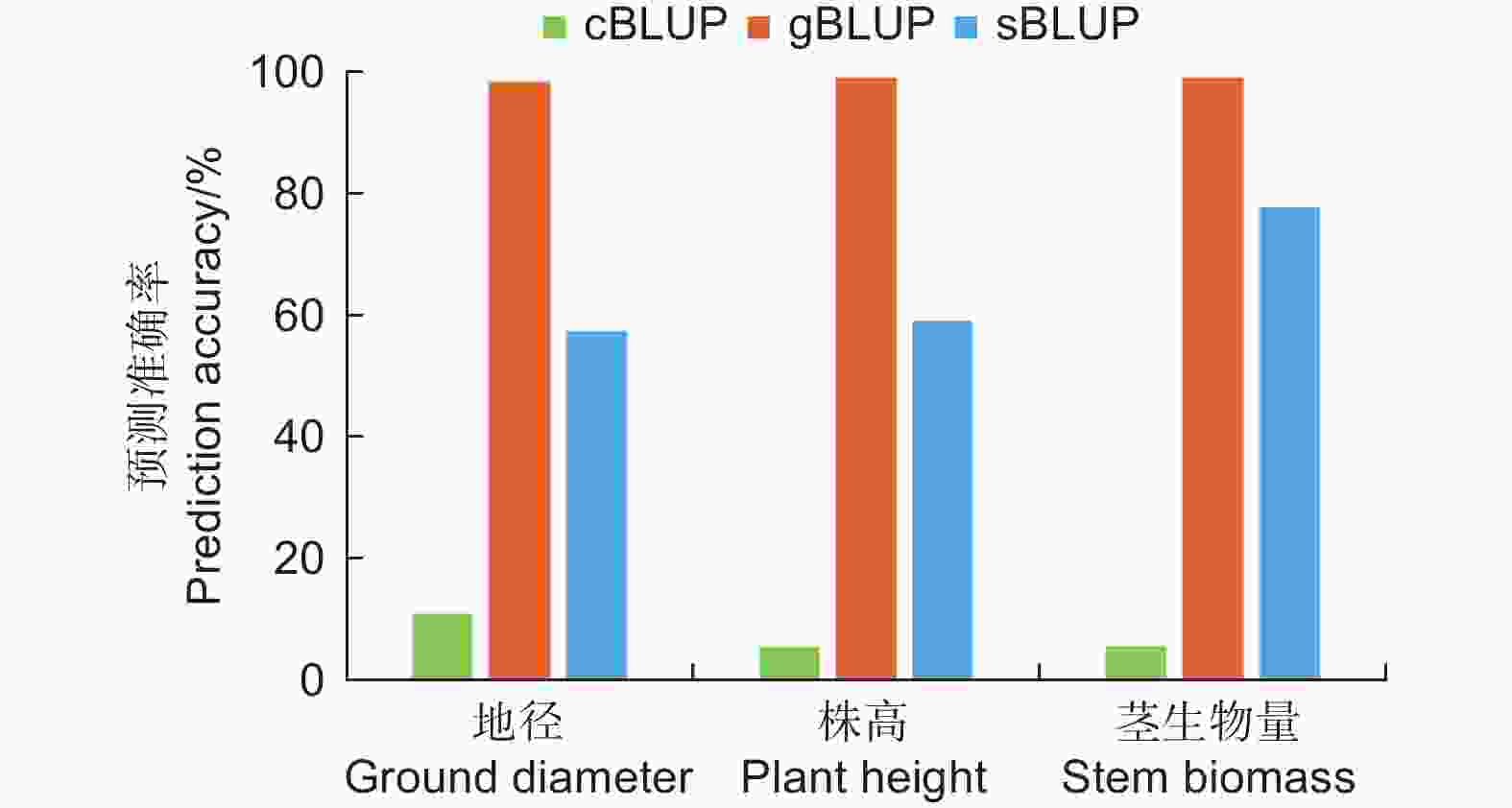
图 4 不同模型计算低氮环境下表型性状育种值的预测准确率比较分析
Figure 4. Comparative analysis of prediction accuracy of phenotypic traits breeding value under low nitrogen environment calculated by different models
364个基因型观测值TBV和cBLUP、gBLUP、sBLUP计算地径的均值分别为16.23、16.24、15.81、15.98;株高的均值分别为277.54、277.68、273.78、275.49;茎生物量的均值为109.01、109.01、109.01、109.01。结果说明低氮条件下地径、株高和茎生物量的育种值和观测值的均值比较分析发现差异较小(表3)。观测值TBV和cBLUP、gBLUP、sBLUP计算的地径的育种值的方差分别为3.31、0.35、1.92、2.14;株高的方差分别为48.32、2.50、22.32、29.42;茎生物量的方差分别为46.59、2.55、46.59、35.00(表3)。通过方差的比较分析可以看出cBLUP模型计算的方差值较小,gBLUP和sBLUP计算的育种值的方差与观测值的方差较为接近。
表 3 低氮环境下观测值和育种值的统计分析
Table 3. Statistical analysis of observed value and breeding value under a low nitrogen environment
性状
Traits模型
Model均值
Mean方差
SD最小值
Min最大值
Max偏度
Skew峰度
Kurtosis标准误
SE地径/mm
Ground diameterTBV 16.23 3.31 2.40 25.33 −0.48 0.91 0.18 cBLUP 16.24 0.35 15.38 17.07 0.01 −0.84 0.02 gBLUP 15.81 1.92 8.99 22.56 −0.35 1.28 0.13 sBLUP 15.98 2.14 8.52 20.15 −0.74 0.71 0.15 株高/cm
Plant heightTBV 277.54 48.32 97.00 376.67 −0.75 0.61 2.61 cBLUP 277.68 2.50 272.23 282.76 0.01 −1.05 0.17 gBLUP 273.78 22.32 184.10 330.00 −0.62 1.11 1.54 sBLUP 275.49 29.42 189.11 339.51 −0.56 0.03 2.03 茎生物量/g
Stem biomessTBV 109.01 46.59 7.33 253.87 0.56 0.35 3.22 cBLUP 109.01 2.55 104.25 113.60 −0.02 −1.44 0.18 gBLUP 109.01 46.59 7.33 253.87 0.56 0.35 3.22 sBLUP 109.01 35.00 54.38 253.55 0.77 0.93 2.42 注:364个基因型的数据比较结果。TBV为观测值,cBLUP、gBLUP、sBLUP分别为3个模型计算的育种值
Notes: Data comparison results of 364 genotypes. TBV is the observed value, and cBLUP, gBLUP, and sBLUP are the breeding values calculated by the three models -
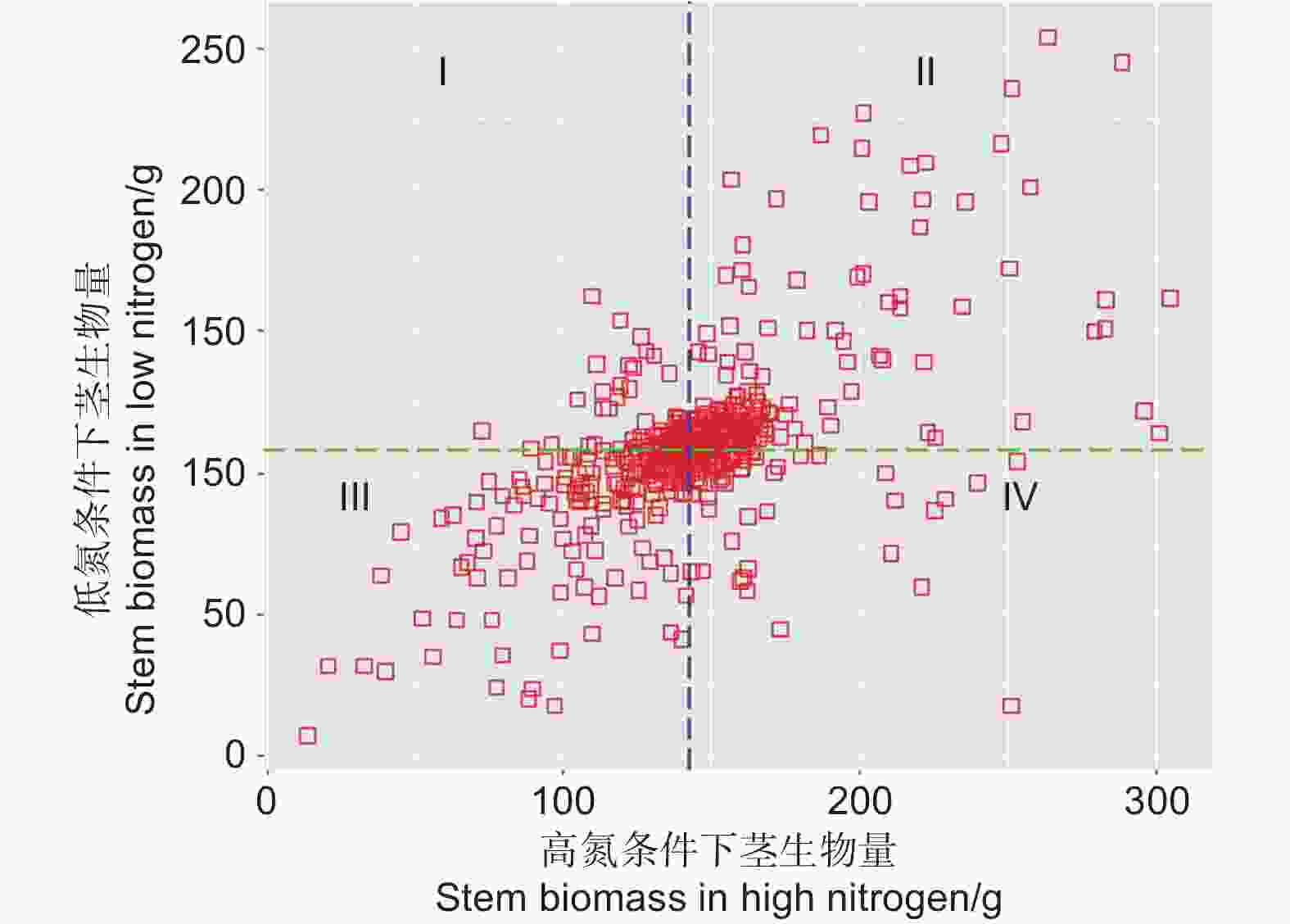
因为gBULP预测的育种值准确率较高,因此利用了杂交群体502个基因型的茎生物量的育种值进行了评价和筛选。根据高氮和低氮条件下茎生物量的均值把F1代群体划分为4种类型(图5)。低氮高效型(Ⅰ区域):本区域F1代的茎生物量在低氮条件下高于均值,在高氮条件下低于均值。双高效型(Ⅱ区域):本区域F1代的茎生物量在低氮和高氮条件下均高于平均值。高氮高效型(Ⅳ区域):本区域F1代的茎生物量在低氮条件下低于均值,在高氮条件下高于均值。低氮低效型(Ⅲ区域):本区域F1代的茎生物量在低氮和高氮条件下均低于平均值。双高效型(Ⅱ区域)包括191个基因型,均值(茎生物量在低氮条件和高氮条件下的均值)的前20名包括16-1-16、16-1-194、13-116、13-73、13-481、13-268、13-286、13-566、13-173、13-578、16-1-65、13-242、16-1-189、13-40、13-608、16-1-170、16-1-22、13-237、13-272、13-335。双高效型中前20名符合育种目标的要求,可作为优良遗传材料保存,进一步研究。
-
在林业生产中,为了避免与粮食生产竞争肥沃的土地,经常在贫瘠的土地上植树造林,而且人工林种植和管理较少施肥。因此杨树人工林的生产力取决于基因型的正确选择,需要研究高生物量生产的杨树品种,以便在边际土壤上种植。本研究利用丹红杨、通辽1号杨和杂交群体在田间进行了施氮肥试验,调查了364个基因型在低氮和高氮条件下的地径、株高和茎生物量。丹红杨的茎生物量在高氮和低氮条件下分别比通辽1号杨提高了20倍和33倍。结果说明丹红杨的生长表型在不同氮环境下显著高于通辽1号杨,具有优良的生长表型性状。田间试验更加贴合实际的木材生产情况,不同的氮肥处理条件下的生长表型性状的调查,可以帮助我们选择优良的高氮利用、耐低氮和高生物量生产的基因型,具有指导实际生产的意义。
基因组选择方法被迅速应用于动物育种[26]和植物育种的研究中[27]。基因组选择研究对多年生树木具有重要的应用价值,因为通过使用基因组标记来预测个体的遗传价值,可以在幼苗阶段选择个体,显著缩短选育周期,例如在林业树木中松树(Pinus pinaster Ait.)[28]、桉树(Eucalyptus spp.)[29]、油棕(Elaeis guineensis Jacq.)[30]。高通量测序技术的发展显著降低了分子标记的成本,覆盖全基因组的高密度分子标记使得复杂性状的基因组选择技术迅速发展。本研究中我们利用全基因组重测序数据,获得了1 447 341个SNPs位点,覆盖了整个基因组的遗传信息,保证了后续的基因组选择的需求。GS利用覆盖全基因组的高密度SNP标记,结合表型记录或系谱记录对个体育种值进行估计,其假定这些标记中至少有一个标记与所有控制性状的QTL(Quantitative trait locus)处于连锁不平衡状态,这样使得每个QTL的效应都可以通过SNP得到反映,将所有标记效应值累加,获得基因组估计育种值[31]。木本植物的选育大多基于田间表型选择,但是田间试验工作量大且繁琐和世代时间长,无法对大量杂交群体展开表型调查。本研究利用364个基因型的表型观测值和3个全基因组选择模型,对502个基因型(包括已知表型和未知表型的所有个体)进行育种值预测。对杨树杂交群体的地径、株高和茎生物量的观测值和3个GS模型计算的育种值的均值和方差进行了分析。群体育种值的均值差异较小,说明整体预测较差异较小;群体育种值的方差差异较大,说明个体预测3个模型差异较大。基因组预测研究结果可以帮助我们预测只有基因型数据没有观测表型值的杨树基因型个体,减少了田间测试的工作量和成本,提高了育种效率。对cBLUP、gBLUP、sBLUP三种预测模型的准确性结果进行了比较分析。gBLUP对生长表型性状预测结果最准确接近于1。sBLUP预测结果的准确性范围是0.5~0.9。cBLUP预测结果的准确性小于0.2。研究结果表明gBLUP模型预测的结果较为准确,cBLUP预测的结果最差。基因组最佳线性无偏预测(gBLUP)在计算速度上具有优势,而且在对极端复杂性状的预测精度上较高,因次适合大范围应用到林木的选育工作中。
我国杨树优良基因型资源的收集、筛选和鉴定工作做得相对较少,这是因为品种的选育需要耗费大量的人力与物力。优良的种质资源是通过大量种质资源筛选出来的,需要科学的评价方法,通过育种值进行评价筛选工作更加稳定和可靠,具有大范围推广的应用价值。由于gBLUP计算的育种值较为准确,因此本研究选择了gBLUP计算的502个基因型的育种值进行了后续的评价和筛选工作。杨树是以收获木材产量为主,因此本研究通过高氮和低氮条件下的茎生物量把F1代群体划分为4种类型,包括双高效型、高氮高效型、低氮高效型和低氮低效型。其中双高效型属于高生物量生产的类型,前20名可以作为优良基因型的备选,如16-1-16、16-1-194、13-116、13-73、13-481、13-268、13-286、13-566、13-173、13-578、16-1-65、13-242、16-1-189、13-40、13-608、16-1-170、16-1-22、13-237、13-272、13-335。育种值的预测和筛选帮助我们实现了早期选择,基因组选择的研究结果具有指导实际生产的意义。全基因组选择的育种应用虽然仍有一些瓶颈,但它必然是智能育种时代非常重要的一项技术,也是未来育种一个重要的方向,它将极大影响未来林木育种的方式和进程。
-
丹红杨和通辽1号杨的生长表型性状差异显著,杂交群体的生长表型性状具有丰富的遗传变异。基因组选择结果表明gBLUP模型预测的结果较为准确,cBLUP预测的结果最差。筛选出高生物量生产的优良基因型16-1-16、16-1-194、13-116、13-73、13-481、13-268、13-286、13-566、13-173、13-578、16-1-65、13-242、16-1-189、13-40、13-608、16-1-170、16-1-22、13-237、13-272、13-335。全基因组选择帮助杨树育种工作完成了早期选择,减少了表型测定成本,缩短了育种周期。
杨树杂交群体苗期生长性状的全基因组选择研究
Genomic Selection of Seedling Growth Traits in a Poplar Hybrid Population
-
摘要:
目的 对杨树生长表型性状进行全基因组选择研究并且实现早期选择具有重要的意义。 方法 以母本丹红杨(美洲黑杨)和父本通辽1号杨(小叶杨)及其杂交F1代为材料,在田间进行施肥和不施肥处理,测定了生长表型性状(地径、株高、茎生物量)。利用3个全基因组选择模型gBLUP、sBLUP、cBLUP和364个基因型的表型观测值对502个基因型进行预测育种值。 结果 丹红杨的茎生物量在高氮和低氮条件下分别比通辽1号杨提高了20倍和33倍。gBLUP对生长表型性状预测结果准确性接近于1,sBLUP预测结果的准确性范围是0.5~0.9,cBLUP预测结果的准确性小于0.2。研究结果表明gBLUP模型预测的结果最准确,cBLUP预测的结果最差。利用gBLUP模型计算的茎生物量育种值把杂交群体划分为双高效型、双低效型、低氮高效型、高氮高效型共4个类型。筛选出优良的基因型16-1-16、16-1-194、13-116、13-73、13-481、13-268、13-286、13-566、13-173、13-578、16-1-65、13-242、16-1-189、13-40、13-608、16-1-170、16-1-22、13-237、13-272、13-335。 结论 丹红杨和通辽1号杨的生长表型性状差异显著。全基因组选择研究结果帮助我们完成了杨树育种工作的早期选择,减少了表型测定成本,提高了育种效率。 Abstract:Objective To study the genomic selection of poplar growth traits and complete early selection. Method The female parent Populus deltoides ‘Danhong’, the male parent Populus simonii ‘Tongliao 1’, and 362 F1 generations were used to determine the growth traits (ground diameter, plant height, and stem biomass) under treatments with fertilization and no fertilization in the field. Three genome-wide selection models, gBLUP, sBLUP, cBLUP, and phenotypic observations of 364 clones were used to predict breeding values for 502 genotypes. Results The stem biomass of ‘Danhong’ increased by 20 times and 33 times compared to ‘Tongliao 1’ under high- and low-nitrogen conditions, respectively. The accuracy of gBLUP prediction for growth traits was close to 1, the accuracy of sBLUP prediction ranged from 0.5 to 0.9, and the accuracy of cBLUP prediction was less than 0.2. The results showed that the gBLUP model predicted the best and the cBLUP predicted the worst. The hybrid population based on the breeding values of stem biomass calculated from the gBLUP model could be classified into four types: double high-efficiency type, double low-efficiency type, low nitrogen high-efficiency type, and high nitrogen high-efficiency type. The excellent clones 16-1-16, 16-1-194, 13 - 116,13 - 73, 13-481, 13-268, 13-286, 13-566, 13-173, 13-578, 16-1-65, 13-242, 16-1-189, 13-40, 13-608, 16-1-170, 16-1-22, 13-237, 13-272, and 13-335 were identified. Conclusion There are significant differences in growth phenotypic traits between ‘Danhong’ and ‘Tongliao 1’. The results help us complete the early selection of poplar breeding work, reduce the cost of phenotype determination, and improve breeding efficiency. -
Key words:
- Poplar
- / growth trait
- / genomic selection
- / gBLUP
- / nitrogen fertilizer
-
表 1 杨树杂交群体表型性状观测值的统计分析
Table 1. Statistical analysis of phenotypic traits of poplar hybrid populations
性状
Traits处理条件
Condition亲本
Parents杂交后代
F1 populations丹红杨
Danhong通辽1号杨
Tongliao1最小值
min最大值
max均值
mean变异系数
Coefficient of variation正态性检验
norm test W遗传力
Heritability地径
Ground diameter/mm高氮 High N 25.2** 7.8 10.0 24.8 18.4 0.15 0.99 0.72 低氮 Low N 24.1** 6.2 6.7 22.7 15.6 0.18 0.99 株高
Plant height/cm高氮 High N 350.0** 126.7 148.0 365.0 293.6 0.13 0.96 0.70 低氮 Low N 335.6** 97.0 142.0 366.7 269.4 0.17 0.98 茎生物量
Stem biomass/g高氮 High N 288.3** 13.6 20.8 304.5 146.7 0.40 0.97 0.70 低氮 Low N 244.9** 7.3 17.8 253.9 108.9 0.42 0.97 注:*, ** 分别表示丹红杨和通辽1号杨之间的差异水平P < 0.05 和 P < 0.01
Notes: *, ** significant difference between two parents' levels of P < 0.05 and P < 0.01, respectively表 2 高氮环境下观测值和育种值的统计分析
Table 2. Statistical analysis of observed value and breeding value under a high nitrogen environment
性状
Traits模型
Model均值
Mean方差
SD最小值
Min最大值
Max偏度
Skew峰度
Kurtosis标准误
SE地径/mm
Ground diameterTBV 17.91 2.96 7.84 25.19 −0.13 −0.03 0.16 cBLUP 17.94 0.42 16.96 18.86 0.06 −0.55 0.03 gBLUP 18.28 2.90 7.84 25.19 −0.21 0.34 0.20 sBLUP 18.08 1.76 13.60 24.55 0.11 0.25 0.12 株高/cm
Plant heightTBV 292.28 69.89 126.67 1131.67 7.17 79.70 3.77 cBLUP 293.75 23.05 251.53 571.89 8.28 98.29 1.59 gBLUP 297.28 59.80 169.55 896.01 6.71 61.19 4.13 sBLUP 293.68 40.59 250.34 675.52 7.90 70.27 2.80 茎生物量/g
Stem biomessTBV 144.61 58.45 13.60 304.53 0.52 −0.09 4.03 cBLUP 144.61 19.49 91.55 203.39 0.45 0.00 1.35 gBLUP 144.61 58.45 13.60 304.53 0.52 −0.09 4.03 sBLUP 144.61 42.89 72.41 347.31 1.01 1.88 2.96 注:364个基因型的数据比较结果。TBV为观测值,cBLUP、gBLUP、sBLUP分别为3个模型计算的育种值
Notes: Data comparison results of 364 genotypes. TBV is the observed value, and cBLUP, gBLUP, and sBLUP are the breeding values calculated by the three models表 3 低氮环境下观测值和育种值的统计分析
Table 3. Statistical analysis of observed value and breeding value under a low nitrogen environment
性状
Traits模型
Model均值
Mean方差
SD最小值
Min最大值
Max偏度
Skew峰度
Kurtosis标准误
SE地径/mm
Ground diameterTBV 16.23 3.31 2.40 25.33 −0.48 0.91 0.18 cBLUP 16.24 0.35 15.38 17.07 0.01 −0.84 0.02 gBLUP 15.81 1.92 8.99 22.56 −0.35 1.28 0.13 sBLUP 15.98 2.14 8.52 20.15 −0.74 0.71 0.15 株高/cm
Plant heightTBV 277.54 48.32 97.00 376.67 −0.75 0.61 2.61 cBLUP 277.68 2.50 272.23 282.76 0.01 −1.05 0.17 gBLUP 273.78 22.32 184.10 330.00 −0.62 1.11 1.54 sBLUP 275.49 29.42 189.11 339.51 −0.56 0.03 2.03 茎生物量/g
Stem biomessTBV 109.01 46.59 7.33 253.87 0.56 0.35 3.22 cBLUP 109.01 2.55 104.25 113.60 −0.02 −1.44 0.18 gBLUP 109.01 46.59 7.33 253.87 0.56 0.35 3.22 sBLUP 109.01 35.00 54.38 253.55 0.77 0.93 2.42 注:364个基因型的数据比较结果。TBV为观测值,cBLUP、gBLUP、sBLUP分别为3个模型计算的育种值
Notes: Data comparison results of 364 genotypes. TBV is the observed value, and cBLUP, gBLUP, and sBLUP are the breeding values calculated by the three models -
[1] LV Z Y, LIU F, ZHANG Y B, et al. Ecologically adaptable Populus simonii is specific for recalcitrance‐reduced lignocellulose and largely-enhanced enzymatic saccharification among woody plants[J]. Global Change Biology Bioenergy, 2020, 13(1): 348-360. [2] CHEN C, CHU Y G, HUANG Q J, et al. Morphological, physiological, and transcriptional responses to low nitrogen stress in Populus deltoides Marsh. clones with contrasting nitrogen use efficiency[J]. BMC Genomics, 2021, 22(1): 697. doi: 10.1186/s12864-021-07991-7 [3] SONG H F, CAI Z Y, LIAO J, et al. Phosphoproteomic and metabolomic analyses reveal sexually differential regulatory mechanisms in poplar to nitrogen deficiency[J]. Journal of Proteome Research, 2020, 19(3): 1073-1084. doi: 10.1021/acs.jproteome.9b00600 [4] CHEN W, MENG C, JI J, et al. Exogenous GABA promotes adaptation and growth by altering the carbon and nitrogen metabolic flux in poplar seedlings under low nitrogen conditions[J]. Tree Physiology, 2020, 40(12): 1744-1761. doi: 10.1093/treephys/tpaa101 [5] HEFFNER E, JANNINK J L, SORRELLS M. Genomic selection accuracy using multifamily prediction models in a wheat breeding program[J]. Plant Genome, 2011, 4(1): 65-75. doi: 10.3835/plantgenome.2010.12.0029 [6] TEMPELMAN R J. Statistical and computational challenges in whole genome prediction and genome-wide association analyses for plant and animal breeding[J]. Journal of Agricultural, Biological, and Environmental Statistics, 2015, 20(4): 442-466. doi: 10.1007/s13253-015-0225-2 [7] YU X Q, LI X R, GUO T T, et al. Genomic prediction contributing to a promising global strategy to turbocharge gene banks[J]. Nature Plants, 2016, 2(10): 16150. doi: 10.1038/nplants.2016.150 [8] CROSSA J, PÉREZ-RODRÍGUEZ P, CUEVAS J, et al. Genomic selection in plant breeding: methods, models, and perspectives[J]. Trends Plant Science, 2017, 22(11): 961-975. doi: 10.1016/j.tplants.2017.08.011 [9] CAMPOS G, GIANOLA D, ALLISON D B. Predicting genetic predisposition in humans: the promise of whole-genome markers[J]. Nature Reviews Genetics, 2010, 11(12): 880-886. doi: 10.1038/nrg2898 [10] ASORO F, NEWELL M, BEAVIS W, et al. Accuracy and training population design for genomic selection on quantitative traits in elite north American oats[J]. Plant Genome, 2011, 4(2): 132-144. [11] 焦宇馨, 张宇翔, 杨文艳, 等. 结合辅助性状的玉米全基因组选择预测力评估[J]. 江苏农业学报, 2023, 39(2):313-320. doi: 10.3969/j.issn.1000-4440.2023.02.002 [12] SPINDEL J, BEGUM H, AKDEMIR D, et al. Correction: genomic selection and association mapping in rice (Oryza sativa): effect of trait genetic architecture, training population composition, marker number and statistical model on accuracy of rice genomic selection in elite, tropical rice breeding lines[J]. PLOS Genetics, 2015, 11(6): e1005350. doi: 10.1371/journal.pgen.1005350 [13] 曹 珂, 陈昌文, 杨选文, 等. 桃果实单果重及可溶性固形物含量的全基因组选择分析[J]. 中国农业科学, 2023, 56(5):951-963. doi: 10.3864/j.issn.0578-1752.2023.05.011 [14] VANRADEN P M. Efficient methods to compute genomic predictions[J]. Journal of Dairy Science, 2008, 91(11): 4414-4423. doi: 10.3168/jds.2007-0980 [15] WANG J B, ZHOU Z K, ZHANG Z, et al. Expanding the BLUP alphabet for genomic prediction adaptable to the genetic architectures of complex traits[J]. Heredity (Edinb), 2018, 121(6): 648-662. doi: 10.1038/s41437-018-0075-0 [16] KLÁPŠTĚ J, LSTIBŮREK M, KOBLIHA J. Initial evaluation of half-sib progenies of Norway spruce using the best linear unbiased prediction[J]. Journal of Forest Science, 2007, 53(2): 41-46. doi: 10.17221/2136-JFS [17] LI C R, WENG Q J, CHEN J B, et al. Genetic parameters for growth and wood mechanical properties in Eucalyptus cloeziana F. Muell[J]. New Forests, 2017, 48(1): 33-49. doi: 10.1007/s11056-016-9554-4 [18] PIEPHO H P, MÖHRING J, MELCHINGER A E, et al. BLUP for phenotypic selection in plant breeding and variety testing[J]. Euphytica, 2008, 161(1-2): 209-228. doi: 10.1007/s10681-007-9449-8 [19] MILLET E J, KRUIJER W, COUPEL-LEDRU A, et al. Genomic prediction of maize yield across European environmental conditions[J]. Nature Genetics, 2019, 51(6): 952-956. doi: 10.1038/s41588-019-0414-y [20] CROSSA J, PÉREZ P, HICKEY J, et al. Genomic prediction in CIMMYT maize and wheat breeding programs[J]. Heredity, 2014, 112(1): 48-60. doi: 10.1038/hdy.2013.16 [21] 张春玲, 李淑梅, 赵自成, 等. 杨树新品种‘丹红杨’[J]. 林业科学, 2008, 44(1):169-169. doi: 10.3321/j.issn:1001-7488.2008.01.027 [22] ZHANG J, SONG X Q, ZHANG L, et al. Agronomic performance of 27 Populus clones evaluated after two 3-year coppice rotations in Henan, China[J]. Global Change Biology Bioenergy, 2020, 12(2): 168-181. doi: 10.1111/gcbb.12662 [23] SUN P, JIA H X, CHENG X Q, et al. Genetic architecture of leaf morphological and physiological traits in a Populus deltoides ‘Danhong’ × P. simonii ‘Tongliao1’ pedigree revealed by quantitative trait locus analysis[J]. Tree Genetics & Genomes, 2020, 16(3): 45. [24] ZHANG P, SU Z Q, XU L, et al. Effects of fragment traits, burial orientation and nutrient supply on survival and growth in Populus deltoides × P. simonii[J]. Scientific Reports, 2016, 6: 21031. doi: 10.1038/srep21031 [25] WANG J B, ZHANG Z W. GAPIT version 3: boosting power and accuracy for genomic association and prediction[J]. Genomics, Proteomics & Bioinformatics, 2021, 19(4): 629-640. [26] HAYES B, GODDARD M. Genome-wide association and genomic selection in animal breeding[J]. Genome, 2010, 53(11): 876-883. doi: 10.1139/G10-076 [27] HEFFNER E, LORENZ A, JANNINK J L, et al. Plant breeding with genomic selection: gain per unit time and cost[J]. Crop Science, 2010, 50(5): 1681-1690. doi: 10.2135/cropsci2009.11.0662 [28] ISIK F, BARTHOLOMÉ J, FARJAT A, et al. Genomic selection in Maritime Pine[J]. Plant Science, 2015, 242: 108-119. [29] BOUVET J M, MAKOUANZI G, CROS D, et al. Modeling additive and non-additive effects in a hybrid population using genome-wide genotyping: prediction accuracy implications[J]. Heredity (Edinb), 2016, 116(2): 146-157. doi: 10.1038/hdy.2015.78 [30] CROS D, DENIS M, BOUVET J M, et al. Long-term genomic selection for heterosis without dominance in multiplicative traits: case study of bunch production in oil palm[J]. BMC Genomics, 2015, 16: 651. doi: 10.1186/s12864-015-1866-9 [31] KRISHNAPPA G, SAVADI S, TYAGI B S, et al. Integrated genomic selection for rapid improvement of crops[J]. Genomics, 2021, 113(3): 1070-1086. doi: 10.1016/j.ygeno.2021.02.007 -




 下载:
下载: