-
遗传多样性是生物多样性的基础和核心,一个物种的遗传多样性决定其稳定性和进化潜力[1]。一般来讲,遗传多样性的高低与物种的进化速度和适应能力呈正相关,但也受到生境片段化和栖息地丧失等严重威胁。生境片段化导致居群变小,结果因遗传漂变和近交衰退,使居群的遗传多样性降低、隐性有害等位基因纯合,进而使后代适合度降低。从进化角度讲,较之于天然居群,其子代群体的遗传多样性水平更能反映居群未来的生存状态和发展趋势[2]。对子代群体遗传多样性的研究不仅可揭示居群间的亲缘关系,而且还能为其进化潜能的深入研究提供重要的基础资料[3]。从已有的研究来看,对子代遗传多样性研究多集中于种子园自由授粉或控制授粉子代群体,而对不同天然居群或孤立木自由授粉子代群体的遗传多样性研究较少。
红豆树(Ormosia hosiei Hemsl. et Wils.)是我国特有的珍贵阔叶用材树种,自然分布于浙江、江西、福建、湖北、四川、重庆、陕西等省(市),其材质优良,有光泽,纹理美观,是制作工艺雕刻、装饰镶嵌等的珍贵用材。由于红豆树具有极高的经济价值,其原有天然资源遭到人为严重破坏,导致现有天然居群通常都很小,几株至数十株不等,片段化严重。红豆树天然居群内每年仅有少量植株开花结实,且具有开花结实间隔期很长,一般为3~5 a,个别植株甚至更长,也有植株开花后不结实或孤立木开花结实现象[4]。已有研究表明, 现有红豆树天然居群均具有较高的遗传多样性[5],且较大的天然居群的遗传多样性较高,但是其子代遗传多样性是否还维持较高水平、居群子代的遗传多样性是否与孤立木子代有较大差异等皆未予揭示。鉴于此,本研究以浙江、江西、福建和四川等有限分布区选择的26个红豆树优树自由授粉子代家系为试验材料,利用SSR分子标记对其遗传多样性进行研究,并比较同一地区天然居群子代与孤立木子代的遗传多样性水平差异,以了解子代遗传多样性的变化规律,阐明天然居群对遗传多样性维持的作用。同时通过相关分析研究子代遗传多样性对其苗木生长的影响,以期为红豆树优良种质的发掘利用提供理论依据。
-
2015年,采用优势木对比法进行材积评定并结合形质指标在浙江省龙泉市、江西省资溪县、福建省柘荣县和四川省内江市等地选择77株优树,并从中选择了当年开花并结实的26株代表性优树,具体情况见表 1。所选优树树龄均大于30 a,树高10~20 m,胸径19.4~95.3 cm。1、2、4号和9、10、11、12号优树分别来自江西资溪马头山(JXMTS)和浙江龙泉八都(ZJBD)的红豆树天然居群,3、25和36号为孤立木优树。当年10—11月,密切观察各优树种子成熟状况,于种子成熟期分别单株采种,并用网袋分装,带回实验室备用。
表 1 红豆树优树来源
Table 1. Sources of Ormosia hosiei plus tree
编号Code 来源Sources 1 江西省资溪县马头山镇 2 江西省资溪县马头山镇 3 江西省资溪县马头山镇排上村周家 4 江西省资溪县马头山镇 5 江西省广昌县尖峰乡 8 浙江省龙泉市锦溪镇岭根村 9 浙江省龙泉市八都镇青山村 10 浙江省龙泉市八都镇青山村 11 浙江省龙泉市八都镇青山村 12 浙江省龙泉市八都镇青山村 13 浙江省龙泉市锦溪镇吴林村 14 浙江省龙泉市八都镇青山村 25 浙江省龙泉市住龙镇住溪村 30 浙江省龙泉市 36 浙江省云和县西弄村 38 浙江省庆元县黄田镇 39 福建省泰宁县 40 福建省泰宁县 42 福建省蒲城县富岭镇 43 福建省松溪县 51 福建省柘荣县富溪镇富溪村 54 福建省柘荣县东源乡 58 福建省柘荣县富溪镇前宅村 75 四川省资阳市安岳县 76 四川省内江市太平镇 77 四川省内江市太平镇 -
每家系随机选取30粒种子,用电子游标卡尺分别测量种长、种宽和种厚,以平行于种胚方向为其长,以腹面横向为其宽,以腹面与背面距离为其厚,3个指标均在最长处测量,精确到0.01 mm,重复3次。然后再通过计算得到长/宽比。种子质量测定采用四分法取样,用1/100电子天平称量种子百粒质量(不足百粒的换算为百粒质量)。
-
2016年5月,将采集的优树种子经热水浸泡结合人工机械破皮后,及时播种于浙江省龙泉市林科院省级保障性苗圃(119.09° E,28.03° N,海拔245 m,年均气温17.6℃,年均降水量1 699.4mm);该圃地为黄壤土,其表层为混合均匀的黄土和泥炭,比例为7:3,排灌条件良好,土壤肥力中等。家系苗木测定采用完全随机区组设计,采用点播方式育苗,3次重复,30株小区,即每小区播种3行,每行10株,行距30 cm,株距15 cm。根据Sjogren等[6]的假设,要检测到稀有等位基因(频率小于5%),取样个体数至少达30。当苗高达15~20 cm时采集子代叶样,子代个体数如少于30株,则全部采集,如多于30株,则随机采集30株子代,每优树子代采集数量为21~30株,共计765株子代。每株幼苗取2~3片嫩叶(为避免叶片采集对幼苗生长造成明显影响,尽量采集中下部叶片),液氮速冻后,置于-80℃冰箱中备用。分别于移苗后1个月和生长季末,每重复试验小区选取中间一行的10株为调查株,用钢卷尺测量其苗高,精确至0.1 cm。第2次生长调查时,增测地径,精确至0.01 mm。
-
采用鼎国昌盛生物技术公司DNA提取试剂盒提取子代基因组DNA,利用NanoDrop-2000超微量分光光度计和0.8%琼脂糖凝胶电泳检测DNA质量及浓度。选用11对多态性好、特异性强的SSR引物进行PCR扩增(未发表数据)。参照胡磊等[7]和赵颖等[8]设定本试验的反应体系为25 μL:2×Taq Plus Master Mix(Vazyme)12.5 μL,Primer F & R(100 μmol)1 μL,DNA模板(20 ng·μL-1)2 μL,ddH2O 9.5 μL。PCR扩增反应在Takara PCR Thermal cycler上进行,其程序为:94℃预变性5 min;94℃变性30 s,72℃延伸90 s,46~57℃退火60 s,35个循环;72℃延伸10 min,4℃保存[7]。PCR扩增产物首先经2%的琼脂糖凝胶电泳检测,质检合格(特异性强、条带清晰)的PCR产物进行毛细管电泳分离(台湾BIOptic(光鼎)公司生产的Qsep100TM)。
-
采用GenALEx6.5软件[9](Peakall & Smouse, 2012)分别计算不同引物和不同家系的遗传多样性参数:等位基因数(NA)、有效等位基因数(NE)、观测杂合度(HO)、期望杂合度(HE)、Shannon多样性指数(I)、Nei’s多样性指数(h)、遗传分化系数Fst及基因流(Nm)等。运用FSTAT[10]进行1 000次模拟,检测居群子代和孤立木子代群体的遗传多样性差异。
-
为了衡量优树子代群体的遗传分化程度,在GenALEx6.5中进行F统计量指数的统计[11],并利用Arlequin3.0软件[12]对不同家系子代群体的遗传差异进行分子变异分析(AMOVA)。利用GenALEx6.5软件计算子代群体间的Nei’s遗传距离和遗传一致度。根据Nei’s遗传距离用NTSYS-pc 2.10e软件基于UMPGA法对26个红豆树优树子代群体构建聚类图。
-
用SPSS19.0软件采用Pearson法对家系遗传多样性参数与其种子及苗木生长性状进行相关性分析。
-
由于前期设计并筛选的12对SSR引物中有1对引物在子代群体中扩增效果不理想,因此,利用11对SSR引物对26个红豆树优树子代群体的765个个体进行扩增,共检测到184个等位基因(表 2)。每个位点的等位基因数(NA)变化范围为13~26个,平均为16.730个,其中,SSR3检测出的位点数最多,而SSR10和SSR13的最少。有效等位基因数(NE)从5.024(SSR5)到11.460(SSR1),平均为7.766。观测杂合度(HO)和期望杂合度(HE)变化范围分别为0.059~0.889和0.801~0.913,其平均值分别为0.469和0.865。除SSR8位点外,其余位点的观测杂合度均小于期望杂合度,说明这些位点处于杂合子缺失状态。由表 2还可以看出:不同位点的多样性参数存在一定差异,SSR1引物的Shannon多样性指数(I)最高,为2.555,SSR5最低,为1.860,平均为2.236,说明各位点对子代群体遗传多样性的贡献不同。
表 2 红豆树优树子代群体的遗传多样性分析
Table 2. Analysis of the genetic diversity of the progeny population of Ormosia hosiei plus tree
引物primer NA NE HO HE I h Fst Nm SSR1 16 11.460 0.828 0.913 2.555 0.913 0.199 1.008 SSR2 19 6.534 0.476 0.847 2.170 0.848 0.227 0.850 SSR3 26 9.069 0.243 0.890 2.463 0.890 0.286 0.625 SSR4 15 6.932 0.453 0.856 2.129 0.856 0.211 0.934 SSR5 13 5.024 0.259 0.801 1.860 0.801 0.285 0.628 SSR6 14 8.711 0.059 0.885 2.298 0.886 0.329 0.511 SSR7 18 8.897 0.505 0.888 2.430 0.888 0.246 0.766 SSR8 16 7.887 0.889 0.873 2.308 0.874 0.197 1.016 SSR9 21 8.310 0.416 0.880 2.416 0.880 0.258 0.718 SSR10 13 6.296 0.395 0.841 2.007 0.842 0.240 0.793 SSR13 13 6.309 0.631 0.841 1.961 0.842 0.094 2.397 均值Mean 16.730 7.766 0.469 0.865 2.236 0.866 0.234 0.931 注:NA:等位基因数; NE:有效等位基因数; HE:期望杂合度; Ho:观测杂合度; I:Shannon多样性指数; h:Nei’s遗传多样性指数; Fst:群体间遗传分化系数. Nm:基因流. NA: Number of alleles; NE: Number of effective alleles; HE: Expected heterozygosity; Ho: Observed heterozygosity; I: Shannon diversity index; h: Nei’s diversity index; Fst: The coefficient of genetic differentiation among populations; Nm: Gene flow. -
26个优树子代的遗传多样性见表 3。不同家系检测到的等位基因总数相差很大,从50个(5号家系)到85个(51号家系),平均为67.8个。优树子代的平均等位基因数(NA)和有效等位基因数(NE)分别为6.185和3.463,NA和NE最高的是51号家系(NA=7.727;NE=4.731),NA最低的为25号家系(4.273),NE最低的为8号家系(2.588)。
表 3 26个红豆树优树子代群体的SSR遗传多样性
Table 3. Genetic diversity of 16 progeny populations of Ormosia hosiei based on SSR
家系family N NA NE HO HE I h F 白化苗Albino seedling 1 52 5.364 3.660 0.442 0.674 1.359 0.690 0.34 No 2 68 6.182 3.430 0.399 0.657 1.359 0.669 0.39 No 3 55 5.000 3.135 0.333 0.629 1.214 0.640 0.47 No 4 76 6.909 4.073 0.410 0.702 1.502 0.714 0.42 No 5 50 4.545 3.026 0.449 0.610 1.150 0.620 0.26 No 8 63 5.727 2.588 0.285 0.539 1.090 0.548 0.47 No 9 64 5.818 3.502 0.482 0.668 1.341 0.679 0.28 No 10 57 5.182 3.116 0.479 0.634 1.258 0.644 0.24 No 11 62 5.636 3.402 0.518 0.660 1.320 0.672 0.22 No 12 83 7.545 4.503 0.543 0.760 1.643 0.773 0.29 No 13 61 5.545 3.031 0.461 0.607 1.210 0.617 0.24 No 14 81 7.364 3.487 0.462 0.682 1.440 0.693 0.32 No 25 47 4.273 2.822 0.339 0.630 1.147 0.640 0.46 Yes 30 76 6.909 4.184 0.448 0.752 1.585 0.766 0.40 No 36 62 5.636 3.163 0.448 0.619 1.224 0.63 0.28 No 38 72 6.545 3.543 0.479 0.651 1.349 0.662 0.26 No 39 58 5.273 2.856 0.439 0.621 1.205 0.632 0.29 No 40 53 4.818 2.880 0.480 0.628 1.191 0.638 0.24 No 42 60 5.455 2.769 0.406 0.589 1.160 0.599 0.31 No 43 82 7.455 3.446 0.439 0.667 1.449 0.679 0.34 No 51 85 7.727 4.731 0.563 0.741 1.640 0.754 0.24 Yes 54 82 7.455 4.398 0.633 0.746 1.608 0.759 0.15 Yes 58 76 6.909 3.857 0.442 0.725 1.535 0.737 0.39 Yes 75 78 7.091 3.539 0.594 0.701 1.434 0.713 0.15 No 76 75 6.818 3.455 0.511 0.657 1.355 0.668 0.22 No 77 84 7.636 3.439 0.687 0.673 1.430 0.684 -0.02 No 均值 67.8 6.185 3.463 0.468 0.662 1.354 0.674 0.29 标准差 1.056 0.556 0.088 0.054 0.164 0.055 注:N:等位基因总数; F:固定指数; 白化苗:Albino seedlings(有/无, Yes/No).N: Total number of alleles; F:fix index 在26个优树子代中,观测杂合度(HO)和期望杂合度(HE)分别为0.285~0.687和0.539~0.760,除个别家系外,绝大多数家系的观测杂合度(HO)小于其期望杂合度(HE),说明红豆树子代家系存在纯合子过剩现象。家系平均的Shannon多样性指数(I)为1.354,其中,12号家系最高(1.643),8号家系最低(1.090)。由此可见,各遗传多样性参数在26个家系中变幅较大,说明红豆树优树子代群体多态性水平差异较大。
以期望杂合度(HE)和Shannon多样性指数(I)这2个评价遗传多样性最常用的指标进行评价,12号家系的遗传多样性水平最高,最低的是8号家系。将孤立木优树子代(3、25、36号家系)和来自于不同居群子代(1、2、4家系来自于JXMTS居群,9、10、11、12家系来自于ZJBD居群)进行对比分析时发现,JXMTS居群(HE和I平均值分别为0.678和1.407)和ZJBD居群子代(HE和I平均值分别为0.680和1.391)遗传多样性水平较高,均显著(P=0.032)或极显著(P=0.002)高于孤立木子代的遗传多样性(HE和I平均值分别为0.626和1.195),而JXMTS和ZJBD两居群子代间的遗传多样性无显著差异(P=0.278)。由表 3中还可看出:子代中观测到有白化苗的家系也都保持了相对较高的遗传多样性。这表明,虽然这些具白化苗的家系中出现了隐性致死基因纯合体,但其存活下来的个体多为杂合体后代。
-
26个子代群体的遗传分化系数变化范围为0.094~0.329,平均值为0.234,其中,SSR6位点的遗传分化最高,分化最低的是SSR13,说明红豆树家系间具有较高的遗传分化水平(表 2)。子代间的基因流变化范围为0.511~2.397,平均为0.931,表明子代间的基因交流程度较低,这与26个红豆树优树多来自不同的天然居群有关。通过分子方差分析(AMOVA)表明:红豆树优树子代群体的遗传变异主要存在于家系内(77.12%),家系间的遗传分化相对较小,仅占22.88%(表 4),这与F统计分析结果一致。
表 4 优树子代群体的分子方差分析(AMOVA)
Table 4. Analysis of molecular variance (AMOVA) of offspring populations of plus trees
变异来源
Source of variation自由度
Df均方和
Sum of squares变异组分
Variance components估计方差
Estimation Variance变异百分率
Percentage variation/%家系间Among families 25 1 702.141 1.099 1.075 22.88 家系内Within families 1 504 5 546.753 3.703 1.156 77.12 总计Total 1 529 7 248.895 4.802 4.805 100.00 为了进一步揭示不同红豆树优树子代间的遗传关系,根据Nei(1987)方法计算了26个子代间的遗传距离(GD)及遗传一致度(GI),26个红豆树优树子代间的遗传距离为0.180~1.988,遗传相似系数为0.137~0.835,其中,8号和10号家系间的遗传距离最大(GD=1.988),说明这两个家系的亲缘关系较远,39号和40号家系的遗传距离最小,其亲缘关系最近。来自同一居群优树子代间的遗传距离也有一定差异,如JXMTS居群子代的遗传距离为0.269~0.764,ZJBD居群子代的遗传距离则在0.540~1.064,而来自江西资溪的4号优树子代和来自福建柘荣的58号优树子代,其地理距离虽较远但其遗传距离则较小,仅为0.415,这意味着子代间的遗传距离与其亲本间的地理距离不完全相关。优树子代间的遗传一致度则与遗传距离呈相反趋势。
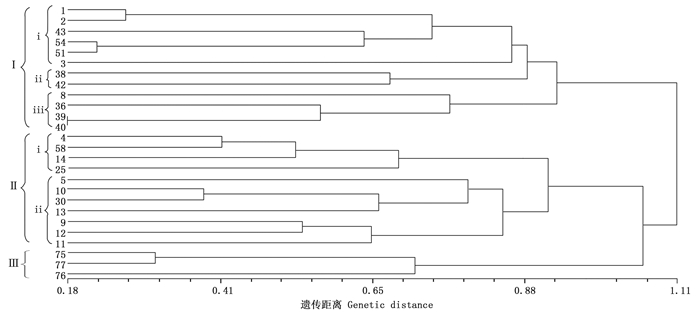
基于遗传距离,用UMPGA法构建了26个家系的聚类图(图 1)。聚类结果显示:在遗传距离为0.97时,26个红豆树优树子代可划分为3组,1、2、3、8、36、38、39、40、42、43、51、54号等12个家系聚为第Ⅰ组,包括江西、福建省及个别来自浙江省的家系;4、5、9、10、11、12、13、14、25、30、58号11个家系聚为第Ⅱ组,主要由浙江龙泉家系组成,四川省的3个家系(75、76、77)聚为第Ⅲ组。当遗传距离阈值取0.80时,第Ⅰ组和第Ⅱ组又可分别划分为3个和2个亚组。第Ⅰ组第ⅰ亚组(包括1、2、3、43、51和54)先与第ⅱ亚组(包括38、42)聚在一起,然后再与第ⅲ亚组(包括8、36、39和40)聚在一起;第Ⅱ组第ⅰ亚组包括4、14、25和58号家系,而第ii亚组包括5、9、10、11、12、13和30号计7个家系。
-
为解释子代遗传多样性是否与种子性状有关,以及是否影响子代的生长,对红豆树优树子代遗传多样性参数与种子、子代生长性状进行了相关性分析,结果(表 5)表明:遗传多样性参数与种子、生长性状存在一定的相关性。在子代遗传多样性参数中,NA和HO 2个遗传多样性参数与种子厚度、百粒质量呈显著正相关(r=0.385~0.527),而与种子长、种子宽及种子长宽比呈不显著正相关(r=0.153~0.272);其它遗传多样性参数与种子各性状间相关性不大,这意味着种子越饱满、百粒质量越大的优树家系,其遗传多样性就越高;NA、HO和I 3个遗传多样性参数均与年高生长量呈显著正相关,相关系数为0.378~0.512,但与移苗后1个月苗高和地径年生长量呈微弱正相关(r=0.049~0.130和r=0.131~0.242)。可见,子代遗传多样性与种子性状、子代年高生长量关系密切。
表 5 红豆树优树子代遗传多样性参数与种子性状、子代生长性状的相关性分析
Table 5. Analysis of genetic diversity parameters, seed traits and growth of Ormosia hosiei
SL SW ST SL/SW HSW 移苗后1个月
1 month after transplanting H年生长量
annual incrementH GD NA 0.272 0.153 0.393* 0.201 0.385* 0.065 0.394* 0.153 HO 0.260 0.160 0.527* 0.192 0.401* 0.130 0.521* 0.131 HE 0.329 0.098 0.285 0.333 0.242 -0.025 0.352 0.235 I 0.304 0.098 0.285 0.318 0.256 0.049 0.378* 0.242 注:SL:种子长; SW:种子宽; ST:种子厚度; SL/SW:种子长宽比; HSW:百粒质量; H:苗高; GD:地径; NA:等位基因数; HO:观测杂合度; HE:期望杂合度; I:Shannon多样性指数 *表示在0.05水平上差异显著。
Note:SL: Seed length; SW: Seed width; ST: Seed thickness; SL/SW: Seed length/Seed width; HSW: Hundred-seed weight; H: Height; GD: Ground diameter; NA: Number of alleles; HO: Observed heterozygosity; HE: Expected heterozygosity; I: Shannon diversity index * indicates significant level at P < 0.05. -
生境片段化减少了居群与外界发生基因交流的机会,遗传漂变和近交衰退使等位基因纯合度提高,同时也增加了有害等位基因纯合度,使后代的遗传多样性和适合度降低[13-14]。本研究利用11对SSR引物对26个红豆树优树子代的756个个体进行扩增,平均每个位点的等位基因数为16.73个,有效等位基因数7.766个,平均观测杂合度和期望杂合度分别为0.469和0.865,Shannon信息指数为2.236,说明红豆树优树子代群体仍保持较高的遗传多样性,但稍低于天然居群的遗传多样性(HE=0.870)(待发表数据),类似于王霞等[15]对观光木(Tsoongiodendron odorum Chun)和Mathiasen等[16]对智利火焰树(Embothrium coccineum J.R. Forst. & G. Forst)的研究结果。这可能与以下两方面有关:首先,红豆树现有天然居群较小,居群中仅有少数几株开花结实,甚至孤立木也能开花结实,从而造成授粉不充分或近交;其次,红豆树优树种子经过机械破皮后人工培育在苗圃,较之于自然更新的群体(如天然林),人为破除种皮阻力和苗圃中适宜的环境使部分纯合体得以生存[17],其遗传多样性或杂合体所占比例要低[18],也就是说天然林中多数自交或近交后代在未达到生殖年龄前死亡。同时,这也说明了片段化生境并没有明显增加红豆树子代的遗传侵蚀,可能是由于自交不亲和机制[16]或是早期表达的致死效应,从而使子代保持较高的遗传多样性,在子代中发现一定比例的白化苗就是很好的佐证。以期望杂合度(HE)作为参照指标,红豆树子代群体的遗传多样性水平亦高于其它濒危木本植物,如鹅掌楸(Liriodendron chinense(Hemsl.)Sargent)(0.68)[2]、厚朴(Magnolia officinalis Rehd. et Wils.)(0.64)[19]和观光木(0.499)[15]等。这与红豆树交配方式以异交为主有关(未发表数据)。
虽然红豆树子代群体保持了较高的遗传多样性,但从各个家系的遗传多样性水平来看,不同红豆树优树子代的遗传多样性水平差异较大,说明这些子代群体存在丰富的遗传变异,可以在子代群体中进行优异种质的选择,来弥补天然居群日渐缩小、遗传多样性水平降低对红豆树长期进化带来的不利影响。进一步分析发现,孤立木优树子代群体的遗传多样性显著低于居群子代,说明较大红豆树天然居群中,有多株植株同年开花,这对子代遗传多样性的维持发挥了重要作用。
-
F统计量(Fst)是指群体间变异占总变异的百分比,是衡量等位基因频率在不同群体间分化程度的指标。研究表明,红豆树优树子代群体的遗传分化系数为0.234,高于天然居群间的遗传分化程度(0.116),却与长寿命林木(0.25)及特有种(0.26)相接近[20],这与红豆树优树来源于不同居群有关。红豆树天然居群片段化现象严重,而优树又来自于不同的居群甚至是孤立木,个体间基因流受阻(Nm=0.931)。另外,AMOVA分析表明,77.12%的变异存在于红豆树优树家系内,说明家系内的变异较大,而优树子代间的遗传分化程度相对较小。
群体间的遗传距离与相应的地理距离之间的关系因物种而异。有研究认为,二者呈正相关[21],但也有研究认为不相关[22]。本文基于遗传距离进行UMPGA法聚类,结果显示,除四川省的3个家系外,其它23个红豆树优树家系可单独聚成一组;而来自浙江、福建和江西3个省的这23个家系在聚类时虽然并不完全按产地各呈一组,但基本呈现同一产地的优树家系优先聚在一起,这与红豆树天然居群的聚类结果大体一致(待发表数据)。但也发现,一些优树子代未能聚合在母树相应的居群中。值得一提的是,除几个母树位于同一居群的优先聚在一起外,母树分别位于江西省和福建省的4号和58号家系却先聚在一起(GD=0.415),其原因有待于进一步研究。总体上看,江西省和福建省的优树子代群体亲缘关系更近。浙江省和四川省优树的地理距离较远,但其亲缘关系却较近,可能与四川省优树家系数量较少有关,因此,后期应加强在各个居群(产地)皆选取一定数量的优树,以进一步鉴定其亲缘关系。
-
目前,关于遗传多样性与子代生长性状的相关性研究结论不一[23-27],且多数涉及居群遗传多样性与子代生长的相关性。在本文中,以NA和HO作为衡量遗传多样性指标,发现红豆树优树子代的遗传多样性与其种子厚度、百粒质量及全年苗高生长量呈显著正相关,这意味着种子越饱满、百粒质量越大的优树家系,其遗传多样性越高,子代苗木的高生长量也就越大。但子代的遗传多样性与其地径相关性较小,这与美洲山杨(Populus tremuloides Michaux)的研究结果一致[28],这可能与植物对竞争的反应是以牺牲地径和根系生长为代价而又利于高生长有关,这种现象在近交个体中尤为明显[29]。然而,在智利火焰树的研究中却表明,有效等位基因数与子代生长(苗高、地径和生物量)均呈显著正相关[16]。但也有研究表明,子代生长与遗传多样性不相关[26],但与母株的异交率呈正相关[13, 23]。依据本研究结果,在今后选择优树采种时,应选择粒大、饱满的种子,进而在后期以苗高作为优良家系的选择依据。由于红豆树并非连年开花,且居群内开花植株的数量和密度不固定,随交配系统的时空变化可能导致同一优树子代不同年份的遗传多样性可能不同,今后还应开展红豆树优树子代遗传多样性及生长性状相关性在多年份的变化规律研究,以验证本文所得出结论的正确性。
-
利用11对SSR引物对26个自由授粉家系的765个子代群体进行遗传多样性分析,共检测到184个等位基因(NA)。红豆树优树子代群体具有较高的遗传多样性,但绝大多数位点处于杂合子缺失状态,且不同家系的遗传多样性存在明显差异。子代群体的遗传变异主要存在于家系内,家系间的遗传分化较小。进一步比较发现,天然居群(JXMTS和ZJBD)子代的遗传多样性显著或极显著地高于孤立木子代,这说明较大红豆树天然居群中,有多株植株同年开花,保证了较为广泛的花粉供体,使得子代维持较宽的遗传基础,这对子代遗传多样性的维持发挥了重要作用。通过遗传多样性与子代生长性状的相关性分析发现,子代遗传多样性参数与种子性状、子代年高生长量呈显著正相关(r=0.378~0.527),但与其地径相关性较小(r=0.131~0.242),在子代苗期选择时,可以苗高作为主要指标。以上研究结果为红豆树遗传保育和优良家系选择提供了理论依据。
红豆树优树子代遗传多样性及与生长相关性分析
Analysis of Genetic Diversity Among Progenies from Plus Trees of Ormosia hosiei and Its Correlation with Growth
-
摘要:
目的 研究红豆树优树自由授粉子代遗传多样性及其对生长的影响,比较天然居群子代和孤立木子代遗传多样性差异,揭示子代遗传多样性的变化规律及天然居群在子代遗传多样性维持中的作用,为红豆树遗传保育和优异种质挖掘提供科学依据。 方法 以来自浙、闽、赣、川等26个红豆树优树自由授粉家系为研究对象,利用11对SSR引物对765个子代群体进行遗传多样性评价,同时分析子代遗传多样性参数与种子、生长性状的相关性。 结果 (1)红豆树优树子代群体具有较高的遗传多样性,有效等位基因数为7.766个,观测杂合度(HO)和期望杂合度(HE)分别为0.469和0.865。(2)除SSR8外,其余位点的观测杂合度均小于期望杂合度,表明子代群体绝大多数位点处于杂合子缺失状态。(3)红豆树不同家系的遗传多样性存在明显差异,12号家系的遗传多样性水平最高,8号家系则最低。(4)比较发现,天然居群子代的遗传多样性显著或极显著地高于孤立木子代。(5)F统计量和分子方差分析(AMOVA)均表明,红豆树优树子代群体的遗传变异主要存在于家系内,家系间的遗传分化相对较小。(6)相关性分析发现,子代遗传多样性参数与种子性状、子代年高生长量呈显著正相关(r=0.378~0.527)。 结论 较大的红豆树天然居群在维持其子代较高遗传多样性中发挥了重要作用,子代遗传多样性显著影响苗木生长,这为红豆树遗传保育和优良家系选择提供了理论依据。 Abstract:Objective To assess the genetic diversity of open-pollinated progenies of Ormosia hosiei and its effect on the growth, and compare the genetic differences of progenies derived from populations and isolate trees, to reveal the change of progenies of O. hosiei plus tree and the role of natural populations in maintenance of genetic diversity, so as to provide references for the genetic conservation and excavation of excellent germplasm resources. Method Eleven polymorphic SSR loci were used to evaluated the genetic diversity of 765 progenies of 26 plus trees coming from Zhejiang, Fujian, Jiangxi and Sichuan provinces and the correlation of genetic diversity parameters with seed and growth traits, meanwhile, the correlation of genetic diversity parameters with seed and growth traits was analyzed. Result (1) The genetic diversity of offspring was rich with the number of 7.766 of effective alleles, the values of observed heterozygosity (HO) and expected heterozygosity (HE) were 0.469 and 0.865 respectively. (2) The HO among loci were less than the HE except SSR8, indicating that the majority locus of offspring was in heterozygous deletion state. (3) The genetic diversity of different families was also different significantly, with the highest appeared in the family 12, and the lowest in the family 8. (4) The genetic diversity of population progeny was significantly higher than that of the isolates. (5) The genetic variation of offspring population mainly existed within family rather than among the families based on the F statistic and analysis of molecular variance (AMOVA). (6) The genetic diversity of offspring was positively correlated with the seed traits and annual increment of seedling height (r=0.378-0.527). Conclusion Higher genetic diversity in the offspring of natural populations of O. hosiei plus tree that played an important role in the maintenance of genetic diversity could significantly promote the growth of seedling. These findings could provide references for the selection of superior family. -
Key words:
- Ormosia hosiei
- / plus tree
- / family
- / genetic diversity
-
表 1 红豆树优树来源
Table 1. Sources of Ormosia hosiei plus tree
编号Code 来源Sources 1 江西省资溪县马头山镇 2 江西省资溪县马头山镇 3 江西省资溪县马头山镇排上村周家 4 江西省资溪县马头山镇 5 江西省广昌县尖峰乡 8 浙江省龙泉市锦溪镇岭根村 9 浙江省龙泉市八都镇青山村 10 浙江省龙泉市八都镇青山村 11 浙江省龙泉市八都镇青山村 12 浙江省龙泉市八都镇青山村 13 浙江省龙泉市锦溪镇吴林村 14 浙江省龙泉市八都镇青山村 25 浙江省龙泉市住龙镇住溪村 30 浙江省龙泉市 36 浙江省云和县西弄村 38 浙江省庆元县黄田镇 39 福建省泰宁县 40 福建省泰宁县 42 福建省蒲城县富岭镇 43 福建省松溪县 51 福建省柘荣县富溪镇富溪村 54 福建省柘荣县东源乡 58 福建省柘荣县富溪镇前宅村 75 四川省资阳市安岳县 76 四川省内江市太平镇 77 四川省内江市太平镇 表 2 红豆树优树子代群体的遗传多样性分析
Table 2. Analysis of the genetic diversity of the progeny population of Ormosia hosiei plus tree
引物primer NA NE HO HE I h Fst Nm SSR1 16 11.460 0.828 0.913 2.555 0.913 0.199 1.008 SSR2 19 6.534 0.476 0.847 2.170 0.848 0.227 0.850 SSR3 26 9.069 0.243 0.890 2.463 0.890 0.286 0.625 SSR4 15 6.932 0.453 0.856 2.129 0.856 0.211 0.934 SSR5 13 5.024 0.259 0.801 1.860 0.801 0.285 0.628 SSR6 14 8.711 0.059 0.885 2.298 0.886 0.329 0.511 SSR7 18 8.897 0.505 0.888 2.430 0.888 0.246 0.766 SSR8 16 7.887 0.889 0.873 2.308 0.874 0.197 1.016 SSR9 21 8.310 0.416 0.880 2.416 0.880 0.258 0.718 SSR10 13 6.296 0.395 0.841 2.007 0.842 0.240 0.793 SSR13 13 6.309 0.631 0.841 1.961 0.842 0.094 2.397 均值Mean 16.730 7.766 0.469 0.865 2.236 0.866 0.234 0.931 注:NA:等位基因数; NE:有效等位基因数; HE:期望杂合度; Ho:观测杂合度; I:Shannon多样性指数; h:Nei’s遗传多样性指数; Fst:群体间遗传分化系数. Nm:基因流. NA: Number of alleles; NE: Number of effective alleles; HE: Expected heterozygosity; Ho: Observed heterozygosity; I: Shannon diversity index; h: Nei’s diversity index; Fst: The coefficient of genetic differentiation among populations; Nm: Gene flow. 表 3 26个红豆树优树子代群体的SSR遗传多样性
Table 3. Genetic diversity of 16 progeny populations of Ormosia hosiei based on SSR
家系family N NA NE HO HE I h F 白化苗Albino seedling 1 52 5.364 3.660 0.442 0.674 1.359 0.690 0.34 No 2 68 6.182 3.430 0.399 0.657 1.359 0.669 0.39 No 3 55 5.000 3.135 0.333 0.629 1.214 0.640 0.47 No 4 76 6.909 4.073 0.410 0.702 1.502 0.714 0.42 No 5 50 4.545 3.026 0.449 0.610 1.150 0.620 0.26 No 8 63 5.727 2.588 0.285 0.539 1.090 0.548 0.47 No 9 64 5.818 3.502 0.482 0.668 1.341 0.679 0.28 No 10 57 5.182 3.116 0.479 0.634 1.258 0.644 0.24 No 11 62 5.636 3.402 0.518 0.660 1.320 0.672 0.22 No 12 83 7.545 4.503 0.543 0.760 1.643 0.773 0.29 No 13 61 5.545 3.031 0.461 0.607 1.210 0.617 0.24 No 14 81 7.364 3.487 0.462 0.682 1.440 0.693 0.32 No 25 47 4.273 2.822 0.339 0.630 1.147 0.640 0.46 Yes 30 76 6.909 4.184 0.448 0.752 1.585 0.766 0.40 No 36 62 5.636 3.163 0.448 0.619 1.224 0.63 0.28 No 38 72 6.545 3.543 0.479 0.651 1.349 0.662 0.26 No 39 58 5.273 2.856 0.439 0.621 1.205 0.632 0.29 No 40 53 4.818 2.880 0.480 0.628 1.191 0.638 0.24 No 42 60 5.455 2.769 0.406 0.589 1.160 0.599 0.31 No 43 82 7.455 3.446 0.439 0.667 1.449 0.679 0.34 No 51 85 7.727 4.731 0.563 0.741 1.640 0.754 0.24 Yes 54 82 7.455 4.398 0.633 0.746 1.608 0.759 0.15 Yes 58 76 6.909 3.857 0.442 0.725 1.535 0.737 0.39 Yes 75 78 7.091 3.539 0.594 0.701 1.434 0.713 0.15 No 76 75 6.818 3.455 0.511 0.657 1.355 0.668 0.22 No 77 84 7.636 3.439 0.687 0.673 1.430 0.684 -0.02 No 均值 67.8 6.185 3.463 0.468 0.662 1.354 0.674 0.29 标准差 1.056 0.556 0.088 0.054 0.164 0.055 注:N:等位基因总数; F:固定指数; 白化苗:Albino seedlings(有/无, Yes/No).N: Total number of alleles; F:fix index 表 4 优树子代群体的分子方差分析(AMOVA)
Table 4. Analysis of molecular variance (AMOVA) of offspring populations of plus trees
变异来源
Source of variation自由度
Df均方和
Sum of squares变异组分
Variance components估计方差
Estimation Variance变异百分率
Percentage variation/%家系间Among families 25 1 702.141 1.099 1.075 22.88 家系内Within families 1 504 5 546.753 3.703 1.156 77.12 总计Total 1 529 7 248.895 4.802 4.805 100.00 表 5 红豆树优树子代遗传多样性参数与种子性状、子代生长性状的相关性分析
Table 5. Analysis of genetic diversity parameters, seed traits and growth of Ormosia hosiei
SL SW ST SL/SW HSW 移苗后1个月
1 month after transplanting H年生长量
annual incrementH GD NA 0.272 0.153 0.393* 0.201 0.385* 0.065 0.394* 0.153 HO 0.260 0.160 0.527* 0.192 0.401* 0.130 0.521* 0.131 HE 0.329 0.098 0.285 0.333 0.242 -0.025 0.352 0.235 I 0.304 0.098 0.285 0.318 0.256 0.049 0.378* 0.242 注:SL:种子长; SW:种子宽; ST:种子厚度; SL/SW:种子长宽比; HSW:百粒质量; H:苗高; GD:地径; NA:等位基因数; HO:观测杂合度; HE:期望杂合度; I:Shannon多样性指数 *表示在0.05水平上差异显著。
Note:SL: Seed length; SW: Seed width; ST: Seed thickness; SL/SW: Seed length/Seed width; HSW: Hundred-seed weight; H: Height; GD: Ground diameter; NA: Number of alleles; HO: Observed heterozygosity; HE: Expected heterozygosity; I: Shannon diversity index * indicates significant level at P < 0.05. -
[1] 熊敏, 田双, 张志荣, 等.华木莲居群遗传结构与保护单元[J].生物多样性, 2014, 22(4):476-484. [2] 李康琴.鹅掌楸属群体遗传结构及分子系统地理学研究[D].南京: 南京林业大学, 2013. [3] Brauner S, Crawford D J, Stuessy T F. Ribosomal DNA and RAPD variation in the rare plant family Lactoridaceae[J]. American Journal of Botany, 1992, 79(12):1436-1439. doi: 10.1002/j.1537-2197.1992.tb13755.x [4] 郑天汉.红豆树生物生态学特征研究[D].福州: 福建农林大学, 2007. [5] Zhang R, Zhou Z, Du K. Genetic diversity of natural populations of endangered Ormosia hosiei, endemic to China[J].Biochemical Systematics and Ecology, 2012, 40(1):13-18. [6] Sjogren P, Wyoni P I. Conservation genetics and detection of rare alleles in finite populations[J].Conservation Biology, 1994, 8(1):267-270. doi: 10.1046/j.1523-1739.1994.08010267.x [7] 胡磊, 高丽, 杨波.利用磁珠富集法开发花榈木微卫星引物[J].华中农业大学学报, 2010, 29(5):629-633. [8] 赵颖, 何云芳, 周志春, 等.浙闽五个红豆树自然保留种群的遗传多样性[J].生态学杂志, 2008, 27(8):1279-1283. [9] Peakall R, Smouse P E. GenAlEx65:genetic analysis in Excel. Population genetic software for teaching and research-an update[J]. Bioinformatics, 2012, 28(19):2537-2539. doi: 10.1093/bioinformatics/bts460 [10] Goudet J. FSTAT, a program to estimate and test gene diversities and fixation indices[EB/OL]. http://www.unil.ch/izea/softwares/fstat.html. 2001. [11] Nei M. Analysis of gene diversity in subdivided populations[J]. Proceedings of the National Academy of Sciences, 1973, 70(12):3321-3323. doi: 10.1073/pnas.70.12.3321 [12] Excoffier L, Laval G, Schneider S. Arlequin (version 30):an integrated software package for population genetics data analysis[J]. Evolutionary Bioinformatics Online, 2005, 1(4A):47-50. [13] Gonzalez-Varo J P, Albaladejo R G, Aparicio A, et al. Linking genetic diversity, mating patterns and progeny performance in fragmented populations of Mediterranean shrub[J].Journal of Applied Ecology, 2010, 47(6):1242-1252. doi: 10.1111/jpe.2010.47.issue-6 [14] 阮咏梅, 张金菊, 姚小洪, 等.黄梅秤锤树孤立居群的遗传多样性及其小尺度空间遗传结构[J].生物多样性, 2012, 20(4):460-469. [15] 王霞, 王静, 蒋敬虎, 等.观光木片断化居群的遗传多样性和交配系统[J].生物多样性, 2012, 20(6):676-684. [16] Muona O, Harju A, Kärkkäinen K. Genetic comparison of natural and nursery grown seedlings of Pinus sylvestris using allozymes[J]. Scandinavian Journal of Forest Research, 1988, 3(1-4):37-46. doi: 10.1080/02827588809382493 [17] Mathiasen P, Rovere A E, Premoli A C. Genetic structure and early effects of inbreeding in fragmented temperate forests of a self-incompatible tree[J]. Embothrium Coccineum. Conservation Biology, 2007, 21(1):232-240. doi: 10.1111/cbi.2007.21.issue-1 [18] Gömöry D. Effect of stand origin on the genetic diversity of Norway spruce (Picea abies Karst.) populations[J]. Forest Ecology & Management, 1992, 54(1-4):215-223. [19] 麦静, 杨志玲, 杨旭, 等.基于SSR标记的厚朴亲本及其子代群体的遗传多样性分析[J].植物资源与环境学报, 2015, 24(3):10-17. doi: 10.3969/j.issn.1674-7895.2015.03.02 [20] Nybom H. Comparison of different nuclear DNA markers for estimating intraspecific genetic diversity in plants[J]. Molecular Ecology, 2004, 13(5):1143-1155. doi: 10.1111/mec.2004.13.issue-5 [21] 王玉山, 邢世岩, 唐海霞, 等.侧柏种源遗传多样性分析[J].林业科学, 2011, 47(7):91-96. [22] 陈娇, 王小蓉, 汤浩茹, 等.基于SSR标记的四川野生中国樱桃遗传多样性和居群遗传结构分析[J].园艺学报, 2013, 40(2):333-340. [23] Ouborg N J, Rvan T. Variation in fitness-related characters among small and large populations of Salvia pratensis[J]. Journal of Ecology, 1995, 83(3):369-380. doi: 10.2307/2261591 [24] Peterson A, Bartish I V, Peterson J. Effects of population size on genetic diversity, fitness and pollinator community composition in fragmented populations of Anthericum liliago L[J]. Plant Ecology, 2008, 198(1):101-110. doi: 10.1007/s11258-007-9388-4 [25] Leimu R, Mutikainen P, Koricheva J, et al.How general arepositive relationships between plant population size, fitnessand genetic variation[J].Journal of Ecology, 2006, 94(5):942-952. doi: 10.1111/jec.2006.94.issue-5 [26] Hensen I, Wesche K. Relationships between Population Size, Genetic Diversity and Fitness Components in the Rare Plant Dictamnus albus, in Central Germany[J]. Biodiversity and Conservation, 2006, 15(7):2249-2261. doi: 10.1007/s10531-004-7183-2 [27] 欧景莉, 郭俊杰, 赵志刚, 等.西南桦自由授粉子代适应性与斑块大小、遗传多样性间的相关性[J].种子, 2012, 31(5):15-18. [28] Latutrie M, Mérian P, Picq S, et al. The effects of genetic diversity, climate and defoliation events on trembling aspen growth performance across Canada[J]. Tree Genetics and Genomes, 2015, 11(5):1-14. [29] 怀特T L, 亚当斯W T, 尼尔D B.森林遗传学[M].李火根, 崔建国译.北京: 科学出版社, 2013: 83-85. -




 下载:
下载: