-
光是影响植物生长发育的最重要的环境因子之一。为了感知生长环境中的光信号并对各种光信号的变化及时做出响应,植物进化出了不同的光受体,通过光受体的调节使环境因子达到最适宜植物生长发育的状态[1]。先前的研究表明,植物共进化出四种光受体,分别包括感受红光和远红光的光敏色素(Phy)[2],感受蓝光的隐花色素(Cry)和向光蛋白(Phot)[3-4]以及感受紫外光-蓝光的UV-B受体(UV-B receptor)[4-5], 其中,感受红光和远红光的光受体-光敏色素是目前研究较清楚的光受体,参与了植物从种子萌发到成熟的整个生长发育过程[6-7]。光敏色素是一种可溶性色素蛋白,在植物体内存在两种状态,分别是具有生物活性的远红光吸收型(FR-absorbing form, Pfr)和不具有生物活性的红光吸收型(R-absorbing form, Pr),且这两种光敏色素可以在红光和远红光的照射下进行相互转换[8]。
在拟南芥基因组中共发现了5个光敏色素基因(PhyA-E)[9-10], 根据其在光下的稳定性不同,拟南芥光敏色素可以分为Ⅰ型光敏色素和Ⅱ型光敏色素。Ⅰ型光敏色素主要存在于黄化幼苗中,遇光极易降解,PhyA属于Ⅰ型光敏色素;Ⅱ型光敏色素主要存在于绿色组织中,遇光稳定,PhyB-E属于Ⅱ型光敏色素[8, 11]。在暗生长的黄化幼苗中,光敏色素以不具有生物活性的状态定位于细胞质中;感受到红光后,光敏色素转变为具有生物活性的远红光吸收型定位于细胞核中,并与细胞核内的其它蛋白质发生相互作用[12-14]。研究表明,光敏色素相互作用因子(PIF)能够直接与具有生物活性的光敏色素结合而启始光信号的传导[15-16]。
PIFs属于bHLH(basic helix-loop-helix)转录因子家族的一个亚家族,也称作PILs(phytochrome interacting factor-like),能够与具有生物活性型的光敏色素相互作用[17]。研究表明,大部分PIFs蛋白质在N端具有保守的APB结构域(active phytochrome-binding domain),APB结构域是PIFs与具有生物活性型光敏色素相结合的不可或缺的元件[18]。拟南芥中至少有7个PIFs成员,PIF1、PIF3、PIF4、PIF5、PIF6、PIF7和PIF8[18-24], 其中,PIF3是利用PHYB作为诱饵通过酵母双杂交筛选出来的最早的PIFs家族成员[20]。免疫共沉淀结果表明,PIF3能与具有生物活性型的PhyA和PhyB的C端相结合。拟南芥中只有PIF1和PIF3能够同时与PhyA和PhyB相互作用,而PIF4-PIF8只能与PhyB相互作用[25]。拟南芥PIF基因家族成员在下胚轴伸长和幼苗避荫反应等过程发挥了重要作用[26-29]。PIF3参与黄花幼苗的形成,在幼苗由黑暗至光照和由光照至黑暗过程中起着重要作用[30]。PIF1参与光诱导的种子萌发,抑制下胚轴伸长,负调控叶绿素合成[21]。PIF4正调控气孔的发育,PIF4、PIF5和PIF7受光周期的调控而影响下胚轴延伸[31]。此外,PIF类的转录因子还具有与靶基因启动子区顺式调控元件G-box (CACGTG)相结合的DNA结合结构域[21]。这些研究结果表明,PIF类转录因子在调控种子萌发、幼苗脱黄化以及开花时间等发育过程具有重要作用。
杨树是多年生木本植物,光是调控其生长发育的重要环境信号之一,光信号的变化使杨树需要具备适应不同发育状态的能力。PIF作为光敏色素相互作用因子,对光环境的变化做出相应的改变调整其达到最适宜树木生长的状态;但目前对于杨树的PtPIF基因家族成员在基因组中分布及其表达模式却鲜见报道。为此,本研究利用生物信息学对杨树PtPIF基因家族成员进行了系统进化分析,鉴定其基因结构、保守结构域,分析了不同家族成员的组织表达特性及其在不同非生物胁迫下的表达规律,研究结果为以后深入解析杨树PtPIF基因的功能奠定了基础。
-
实验所用材料毛果杨(Populus trichocarpa Torr. & Gray)由中国林业科学研究院林木遗传育种国家重点实验室保存。
-
RNA提取试剂盒RNeasy Plant;Mini与RNase-free DNase Ⅰ均购买于Qiagen公司;反转录试剂盒SuperscriptⅢ First-strand Kit以及PCR所用试剂均购买于Takara公司。采用Primer 5.0软件设计引物,实验所需引物在Invitrogen公司合成。
-
首先获取TAIR(http://www.arabidopsis.org/)数据库中已公布的拟南芥PIF基因及其编码的蛋白质序列,并用得到的拟南芥PIF蛋白序列作为诱饵,在杨树基因组数据库(http://www.phytozome.net/poplar.php)中通过blastp比对检索,获得PIF直系同源序列。使用MEGA5.0软件中的Neighbor-Joining(NJ)法构建系统发育进化树。杨树PtPIF蛋白的基本理化性质采用ProtParam(http://web.expasy.org/portparam/)分析完成。使用Gene Structure Display Server(http://gsds.cbi.pku.edu.cn/)软件分析杨树PtPIF基因的外显子及内含子结构。使用在线软件MEME(http://meme-suite.org/)分析杨树PtPIF蛋白的结构域。
-
使用RNA提取试剂盒(RNeasy Plant Kit)提取毛果杨的幼叶、成熟叶、初生茎、次生茎和根的总RNA,反转录合成cDNA第一链,并以cDNA为模板进行qRT-PCR。反应体系为:LightCycler®80 2×SYBR®Premix Ex TaqTM 10 μL,5′-引物(10 μmol·L-1)0.8 μL,3′-引物(10 μmol·L-1)0.8 μL,cDNA2 μL,超纯水6.4 μL;反应程序为:94℃30 s;94℃12 s,58℃30 s,72℃40 s,45个循环。引物序列见表 1。
表 1 实时荧光定量PCR所用引物序列
Table 1. The primer pair sequences used for quantitative real-time PCR
引物
Primer引物序列(5′-3′)
Primer sequence(5′-3′)PtPIF1-F TACCTGGTACTAAAGAGATCCGATC PtPIF1-R GAGGATATAGCAAATCGGCG PtPIF3a-F CAAGTAGTAGCAATCCGGCTG PtPIF3a-R GGAATTGTAGCCTCAGGTAGCT PtPIF3b-F AATAATCTATGTGCAGGATGCTGA PtPIF3b-R CTCATTGATCCTATCCCTTCG PtPIF4/5a-F CATAGAGCCAGACAATGAGCTAGTA PtPIF4/5a-R CTTGAATCAAATGACTTGAATTCAG PtPIF4/5b-F TGGAAGGAAAGTCGGCTACA PtPIF4/5b-R TGAGGTTTATTGTTGTTGAGTTGTC PtPIF8a-F AATGGGAATGGGAATGGG PtPIF8a-R GGCATTACCGTGGCTGAA PtPIF8b-F CAGTACTGGTTCGGACCATGA PtPIF8b-R GCTGGGCCGGAGTCTATC PtPIF9a-F ATCAACGTCTTCTTCCTCATTTATA PtPIF9a-R GTCTCGTTCTCACATTCCCAG PtPIF9b-F AGCTCAGAGCAAAATTCTCTTAATC PtPIF9b-R CGTGGACAGATTGAAAGGTG PtPIF10-F TTCAATGACCAACAATATCTAATGG PtPIF10-R ACCTCTTACTGGTAGTGGCATAGTT PtActin-F GTCCTTCTAACTTCCCAACAGTGC PtActin-R GACTACCAAAGTGTCTGACCACCA -
毛果杨组培苗培养1个月后,转移至人工气候箱中用Hogaland营养液水培。水培15 d后,分别进行4℃低温处理、干旱处理(10% PEG6000)及盐处理(150 mmol·L-1 NaCl),于处理6、12、24、36 h后取由顶端数的第4片叶片,每个处理设置3个重复,每次重复3株,以未处理的毛果杨作为对照。
-
使用RNeasy Plant Mini试剂盒与RNase-free DNase Ⅰ试剂盒提取处理后杨树叶片的RNA,使用反转录试剂盒SuperscriptⅢ First-strand Kit合成cDNA第一链。使用Primer 5.0在杨树PIF基因核苷酸序列的非保守区设计定量PCR扩增引物(表 1),引物序列的GC含量低于30%,Tm值为58~60℃,并由Invitrogen公司合成。实时荧光定量PCR根据SYBR®Premix Ex TaqTMⅡkit(TaKaRa Dalian, China)和Roche480实时荧光定量PCR仪的使用说明进行操作。反应体系为:LightCycler®480 2×SYBR®Premix Ex TaqTM 10 μL,上下游引物各0.8 μL,cDNA 2 μL,ddH2O 6.4 μL。选取杨树Actin(基因序列号:EF418792.1)作为内参基因。每个cDNA模板设置四个重复,所有实验重复三次,采用2-ΔΔCt计算方法进行数据分析。
-
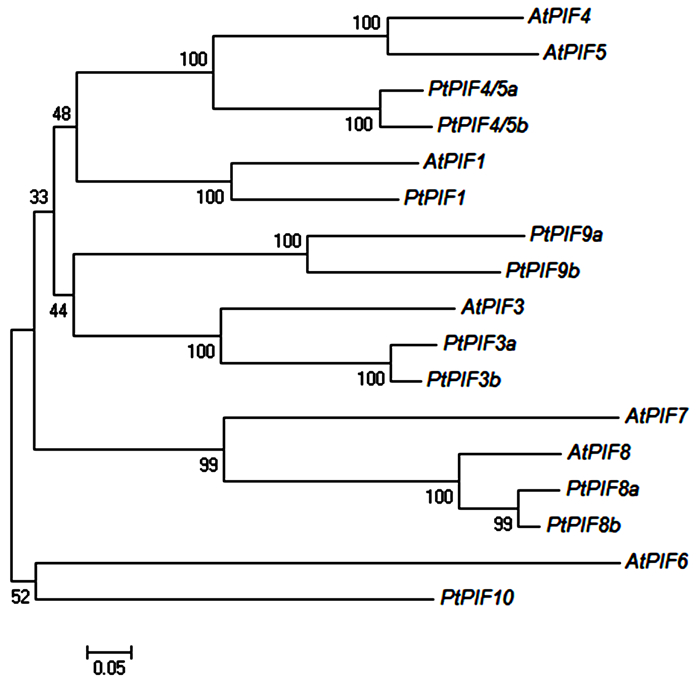
通过序列比对分析,在杨树基因组数据库中共筛选得到10个PtPIF基因成员(表 2)。这10个PIF基因长度为2 810~6 064 bp,所编码的蛋白序列长度为310~742 aa,蛋白质分子质量介于33~79 kDa,等电点为5.42~9.17,既有酸性蛋白,又有碱性蛋白。使用NJ法构建基于杨树和拟南芥PIF成员序列的系统发育进化树(图 1),根据聚类结果,将杨树10个PIF家族成员基因分别命名为PtPIF1、PtPIF3a、PtPIF3b、PtPIF4/5a、PtPIF4/5b、PtPIF8a、PtPIF8b、PtPIF9a、PtPIF9b和PtPIF10。
表 2 PtPIFs基因成员的基本信息
Table 2. Basic information of PtPIF family members in Populus
基因名
Gene基因号
Gene number基因在染色体上的位置
Location基因长度
Length of gene/bp蛋白质长度
Length of protein/aa等电点/分子量
pI/(MW/kDa)PtPIF1 Potri.002G252800 Chr02(-) 24187022-24193085 6 064 615 7.29/(67.366 00) PtPIF3a Potri.005G001800 Chr05 (-) 107969-111992 4 024 742 5.89/(79.784 41) PtPIF3b Potri.013G001300 Chr13 (-) 86122-91428 5 307 719 5.61/(77.620 79) PtPIF4/5a Potri.002G055400 Chr02 (-) 3705303-3709545 4 243 583 6.13/(64.164 79) PtPIF4/5b Potri.005G207200 Chr05 (+) 22153104-22156744 3 641 478 9.17/(52.311 36) PtPIF8a Potri.002G143300 Chr02 (+) 10651063-10655266 4 204 484 8.80/(52.715 58) PtPIF8b Potri.014G066500 Chr14 (-) 5402159-5406848 4 690 471 8.43/(51.139 76) PtPIF9a Potri.005G139700 Chr05 (+) 11648940-11653997 5 058 327 6.13/(35.941 34) PtPIF9b Potri.014G025800 Chr14 (+) 2209016-2211913 2 898 310 5.42/(33.741 64) PtPIF10 Potri.014G111400 Chr14 (-) 8714454-8717263 2 810 561 6.97/(61.476 18) 
图 1 杨树和拟南芥PIF基因家族成员系统进化分析
Figure 1. Phylogenetic relationship of PIF family members between Populus and Arabidopsis
通过对杨树PIF家族各成员的基因结构(外显子/内含子)分析,发现其中PtPIF10由5个外显子和4个内含子组成,PtPIF4/5b、PtPIF8a、PtPIF8b和PtPIF9b均由6个外显子和5个内含子组成,PtPIF3a、PtPIF3b和PtPIF9a均由7个外显子和6个内含子组成,PtPIF4/5a由8个外显子和7个内含子组成,而PtPIF1由10个外显子和9个内含子组成(图 2)。
-
使用软件对杨树PIF蛋白进行保守结构域分析。结果表明:杨树PIF蛋白与拟南芥PIF蛋白在功能结构域上具有较高的保守性(图 3),都具有bHLH DNA结合结构域,推测它们可能具有相似的功能。
-
为了探究杨树中PIF基因成员的组织表达特性,利用qRT-PCR方法分析了PtPIF在幼叶、成熟叶、初生茎、次生茎及根等不同组织中的表达模式, 结果(图 4)表明:PtPIF1、PtPIF8a与PtPIF9b在各组织中普遍表达,且PtPIF1与PtPIF8a均在成熟叶的表达相对较高,而PtPIF9b在幼叶的表达相对较高。PtPIF3a与PtPIF8b在根中检测不到表达,但在其他部位均有表达,且在幼叶和成熟叶中高表达。PtPIF4/5b只在成熟叶中表达,在其它部位检测不到表达。PtPIF3b、PtPIF4/5a、PtPIF9a和PtPIF10在各组织中的表达水平较低。杨树PIF基因家族成员在各组织中的表达差异,说明不同成员在杨树发育过程中可能具有不同的功能。
-
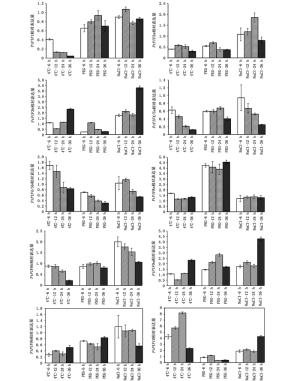
为揭示杨树PIF基因在非生物胁迫下的响应机制,利用qRT-PCR方法检测其在低温、干旱和NaCl胁迫下的表达模式(图 5)。在盐(150 mmol ·L-1 NaCl)、干旱(10% PEG)及4℃低温处理下,杨树PtPIF基因家族各成员间在响应时间、程度上各有不同。低温处理后,PtPIF1、PtPIF3a、PtPIF4/5a、PtPIF4/5b、PtPIF9b和PtPIF10响应时间较早,表达量均在6 h较对照显著改变,其中, PtPIF9b和PtPIF10分别是对照的0.3和4.3倍。之后PtPIF9b的表达量基本维持这个水平,而PtPIF10的表达量则呈先上升后下降的趋势,在24 h达到对照的8.2倍,36 h仅为对照的2.3倍。PtPIF1、PtPIF4/5a和PtPIF8b的表达量均随着处理时间的延伸而降低,且在36 h达到最低值,分别是对照的0.12、0.04、0.23倍。PtPIF3b和PtPIF9a的表达量呈现先下降后上升的趋势,在36 h均到达对照的2.5倍。干旱处理下,PtPIF1、PtPIF3a、PtPIF3b、PtPIF4/5a、PtPIF4/5b、PtPIF9b和PtPIF10的表达量较对照组均下调,PtPIF8a和PtPIF9a的表达量上调。盐处理下,PtPIF3a、PtPIF3b、PtPIF8b、PtPIF9a和PtPIF10的表达量均上调,PtPIF3b、PtPIF9a和PtPIF10的表达量在36 h达到对照的4.3倍。PtPIF4/5a的表达量随着处理时间的延伸而降低,在36 h为对照的0.25倍。
-
光是调控杨树生长发育的重要环境信号,PIF作为bHLH转录因子家族的一个亚家族广泛参与光信号传导途径。因此,利用杨树基因组数据进行PIF基因识别以及表达模式分析对揭示其在杨树生长发育过程中的作用具有重要意义。
基因的表达特性分析是进一步探究其功能的前提。本研究中杨树PtPIF在不同组织中表达模式存在明显差异,表明它们可能参与了不同的生物学过程。研究发现, 拟南芥PIF4和PIF5通过促进与衰老相关基因的表达而促使叶片衰老,而光敏色素B通过抑制PIF4和PIF5的表达而延缓衰老[32]。本研究发现, 杨树中拟南芥PIF4和PIF5的同源基因PtPIF4/5a和PtPIF4/5b在成熟叶片中高丰度表达,推测这2个基因可能参与杨树叶片衰老过程。GUS染色发现, 水稻光敏色素相互作用因子OsPIL1/OsPIL13在茎的节间处高表达,超表达OsPIL1/OsPIL13促进了水稻节间的伸长,表明OsPIL1/OsPIL13参与了水稻细胞的次生生长[33]。本研究发现, PtPIF3b、PtPIF4/5a、PtPIF9a和PtPIF10在成熟茎中确有高丰度表达,推测这些基因可能参与了杨树茎的次生生长。
PIF不仅具有参与光信号转导的能力,而且在响应低温、干旱和盐等非生物胁迫方面发挥着重要作用[34-35]。本研究中,干旱和盐胁迫下, PtPIF1的表达量变化不显著,而在低温胁迫下PtPIF1的表达量急剧下降,在36 h的表达量为对照的0.04倍,表明PtPIF1在响应低温胁迫上具有重要作用。PtPIF3a、PtPIF4/5a、PtPIF9b、PtPIF8b与PtPIF1类似,在低温胁迫下的表达量显著降低。PtPIF3b与PtPIF9a在低温和盐胁迫处理下具有相同的表达模式,低温胁迫下均呈先下降后上升的趋势,盐胁迫下均呈随着胁迫时间的延长而逐渐上升;但在干旱处理下却有着不同的表达模式,PtPIF3b的表达量下降,而PtPIF9a的表达量则呈现上升,表明在响应干旱胁迫时这2个基因具有功能互补机制。低温及盐胁迫下PtPIF10高表达,而干旱胁迫下低表达,表明PtPIF10在胁迫环境下发挥着重要作用。
-
本研究通过对杨树PtPIF基因家族的结构特点和组织表达特异性的分析,在杨树基因组中检测到10个PtPIF基因成员,且均含有bHLH转录因子典型的结构域;对PtPIF基因家族成员在顶端、幼叶、成熟叶、幼茎、成熟茎和根中的表达量进行分析,发现杨树PtPIF基因家族成员组织表达各异,表明PIF基因参与了不同的生物学过程。低温胁迫下,PtPIF10的表达量显著升高,最高时达到对照的8.2倍;干旱处理下,PtPIF8a的表达量较对照上升了4倍以上;盐胁迫导致PtPIF3b、PtPIF9a和PtPIF10的表达量在36 h达到对照的4.3倍,表明杨树PtPIF基因在低温、干旱和盐等非生物胁迫中发挥着重要作用。本研究为揭示PtPIF基因在杨树生长发育过程中的作用奠定了基础。
杨树PIF基因家族成员表达模式研究
Analysis of Expression Pattern of PIF Family Members in Populus
-
摘要:
目的 为探究光敏色素相互作用因子(PIF)基因在杨树生长发育中的功能。 方法 利用生物信息学方法预测了杨树基因组中的PIF基因家族成员并对各成员的基因结构、保守基序进行了系统分析; 在此基础上, 采用实时荧光定量PCR技术检测了PIF成员在不同组织及低温、干旱和NaCl胁迫下的表达模式。 结果 表明:杨树基因组中至少含有10个PtPIF成员, 这些成员在进化上相对保守, 均具有典型的bHLH结构域。基因表达分析显示:PtPIF基因在杨树不同组织及不同非生物胁迫条件下存在明显的表达差异。 结论 杨树PtPIF基因家族包含10个成员参与不同的生物学过程, 研究结果将为深入解析杨树PtPIF基因的功能奠定了基础。 -
关键词:
- 杨树
- / 光敏色素相互作用因子
- / 表达分析
Abstract:Objective To study the function of phytochrome interacting factor (PIF) in Populus. Method The molecular characteristics analysis of PIF was conducted, including gene family members, gene structure and conserved motifs. The expression profiles of PIF family members in different tissues were tested under cold, drought and NaCl stresses with real-time quantitative PCR. Result The results of bioinformatics analysis showed that there were at least ten PtPIF members in Populus and the PtPIF genes were conserved during evolution of species, all of them contained bHLH domain. Conclusion The result of qRT-PCR revealed that PtPIF has distinct expression pattern across different tissues and under different abiotic stress conditions. Present study provides references for further decipher the function of PtPIF in Populus. -
Key words:
- Populus
- / phytochrome-interacting factor
- / expression analysis
-
表 1 实时荧光定量PCR所用引物序列
Table 1. The primer pair sequences used for quantitative real-time PCR
引物
Primer引物序列(5′-3′)
Primer sequence(5′-3′)PtPIF1-F TACCTGGTACTAAAGAGATCCGATC PtPIF1-R GAGGATATAGCAAATCGGCG PtPIF3a-F CAAGTAGTAGCAATCCGGCTG PtPIF3a-R GGAATTGTAGCCTCAGGTAGCT PtPIF3b-F AATAATCTATGTGCAGGATGCTGA PtPIF3b-R CTCATTGATCCTATCCCTTCG PtPIF4/5a-F CATAGAGCCAGACAATGAGCTAGTA PtPIF4/5a-R CTTGAATCAAATGACTTGAATTCAG PtPIF4/5b-F TGGAAGGAAAGTCGGCTACA PtPIF4/5b-R TGAGGTTTATTGTTGTTGAGTTGTC PtPIF8a-F AATGGGAATGGGAATGGG PtPIF8a-R GGCATTACCGTGGCTGAA PtPIF8b-F CAGTACTGGTTCGGACCATGA PtPIF8b-R GCTGGGCCGGAGTCTATC PtPIF9a-F ATCAACGTCTTCTTCCTCATTTATA PtPIF9a-R GTCTCGTTCTCACATTCCCAG PtPIF9b-F AGCTCAGAGCAAAATTCTCTTAATC PtPIF9b-R CGTGGACAGATTGAAAGGTG PtPIF10-F TTCAATGACCAACAATATCTAATGG PtPIF10-R ACCTCTTACTGGTAGTGGCATAGTT PtActin-F GTCCTTCTAACTTCCCAACAGTGC PtActin-R GACTACCAAAGTGTCTGACCACCA 表 2 PtPIFs基因成员的基本信息
Table 2. Basic information of PtPIF family members in Populus
基因名
Gene基因号
Gene number基因在染色体上的位置
Location基因长度
Length of gene/bp蛋白质长度
Length of protein/aa等电点/分子量
pI/(MW/kDa)PtPIF1 Potri.002G252800 Chr02(-) 24187022-24193085 6 064 615 7.29/(67.366 00) PtPIF3a Potri.005G001800 Chr05 (-) 107969-111992 4 024 742 5.89/(79.784 41) PtPIF3b Potri.013G001300 Chr13 (-) 86122-91428 5 307 719 5.61/(77.620 79) PtPIF4/5a Potri.002G055400 Chr02 (-) 3705303-3709545 4 243 583 6.13/(64.164 79) PtPIF4/5b Potri.005G207200 Chr05 (+) 22153104-22156744 3 641 478 9.17/(52.311 36) PtPIF8a Potri.002G143300 Chr02 (+) 10651063-10655266 4 204 484 8.80/(52.715 58) PtPIF8b Potri.014G066500 Chr14 (-) 5402159-5406848 4 690 471 8.43/(51.139 76) PtPIF9a Potri.005G139700 Chr05 (+) 11648940-11653997 5 058 327 6.13/(35.941 34) PtPIF9b Potri.014G025800 Chr14 (+) 2209016-2211913 2 898 310 5.42/(33.741 64) PtPIF10 Potri.014G111400 Chr14 (-) 8714454-8717263 2 810 561 6.97/(61.476 18) -
[1] Jiao Y, Lau O S, Deng X W. Light-regulated transcriptional networks in higher plants[J]. Nature Reviews Genetics, 2007, 8(3):217-230. doi: 10.1038/nrg2049 [2] Nagy F, Schäfer E. Phytochromes control photomorphogenesis by differentially regulated, interacting signaling pathways in higher plants[J]. Annual Review of Plant Biology, 2002, 53:329-355. doi: 10.1146/annurev.arplant.53.100301.135302 [3] Casal J J. Phytochromes, cryptochromes, phototropin:photoreceptor interactions in plants[J]. Photochemistry and Photobiology, 2000, 71(1):1-11. [4] Ulm R, Nagy F. Signaling and gene regulation in response to ultraviolet light[J]. Current Opinion in Plant Biology, 2005, 8(5):477-482. doi: 10.1016/j.pbi.2005.07.004 [5] Quail P H. Photosensory preception and signaling in plant cells:new paradigms[J]. Current opinion of Cell Biology, 2002, 14(2):180-188. doi: 10.1016/S0955-0674(02)00309-5 [6] Parks B M, Quail P H. hy8, a new class of arabidopsis long hypocotyl mutants deficient in functional phytochrome A[J]. Plant Cell, 1993, 5(1):39-48. doi: 10.1105/tpc.5.1.39 [7] Nagatani A, Reed J W, Chory J. Isolation and initial characterization of Arabidopsis mutants that are deficient in phytochrome A[J]. Plant Physiology, 1993, 102(1):269-277. doi: 10.1104/pp.102.1.269 [8] Sharrock R A, Quail P H. Novel phytochrome sequences in Arabidopsis thaliana: structure, evolution, and differential expression of a plant regulatory photoreceptor family[J]. Genes and Development, 1989, 3(11):1745-1757. doi: 10.1101/gad.3.11.1745 [9] Clack T, Mathews S, Sharrock R A. The phytochrome apoprotein family in Arabidopsis is encoded by five genes:the sequences and expression of PHYD and PHYE[J]. Plant Molecular Biology, 1994, 25(3):413-427. doi: 10.1007/BF00043870 [10] Clough R C, Vierstra R D. Phytochrome degradation[J]. Plant, Cell and Environment, 1997, 20(6):713-721. doi: 10.1046/j.1365-3040.1997.d01-107.x [11] Sharrock R A, Clack T. Patterns of expression and normalized levels of the five Arabidopsis phytochromes[J]. Plant Physiology, 2002, 130(1):442-456. doi: 10.1104/pp.005389 [12] Rockwell N C, Su Y S, Lagarias J C. Phytochrome structure signaling mechanisms[J]. Annual Review of Plant Biology, 2006, 57(1):837-858. doi: 10.1146/annurev.arplant.56.032604.144208 [13] Kevei E, Schäfer E, Nagy F. Light-regulated nucleo-cytoplasmic partitioning of phytochromes[J]. Journal of Experimental Botany, 2007, 58(12):3113-3124. doi: 10.1093/jxb/erm145 [14] Franklin K A, Quail P H. Phytochrome functions in Arabidopsis development[J]. Journal of Experimental Botany, 2010, 61(1):11-24. doi: 10.1093/jxb/erp304 [15] Quail, P H. Phytochrome-interacting factors[J]. Seminars in Cell & Developmental Biology, 2000, 11(6):457-466. [16] Zhang Y, Mayba O, Pfeiffer A, et al. A quartet of PIF bHLH factors provides a transcriptionally centered signaling hub that regulates seeding morphogenesis through differential expression-patterning of shard target genes in Arabidopsis[J]. PLOS Genetics, 2013, 9(1):e1003244. doi: 10.1371/journal.pgen.1003244 [17] Toledo-Ortiz G, Huq E, Quail P H. The Arabidopsis basic/helix-loop-helix transcription factor family[J]. Plant Cell, 2003, 15(8):1749-1770. doi: 10.1105/tpc.013839 [18] Khanna R, Huq E, Kikis E A, et al. A novel molecular recognition motif necessary for targeting photoactivated phytochrome signaling to specific basic helix-loop-helix transcription factors[J]. Plant Cell, 2004, 16(11):3033-3044. doi: 10.1105/tpc.104.025643 [19] Ni M, Tepperman J M, Quail P H. Binding of phytochrome B to its nuclear signaling partner PIF3 is reversibly induced by light[J]. Nature, 1999, 400:781-784. doi: 10.1038/23500 [20] Huq E, Quail P H. PIF4, a phytochrome-interacting bHLH factor, functions as a negative regulator of phytochrome B signaling in Arabidopsis[J]. The EMBO Journal, 2002, 21(10):2441-2450. doi: 10.1093/emboj/21.10.2441 [21] Huq E, AI-Sady B, Hudson M, et al. Phytochrome-interacting factor 1 is a critical bHLH regulator of chlorophyⅡ biosynthesis[J]. Science, 2004, 305:1937-1941. doi: 10.1126/science.1099728 [22] Oh E, Kim J, Park E, et al. PIL5, a phytochrome-interacting basic helix-loop-helix protein, is a key negative regulator of seed germination in Arabidopsis thaliana[J]. Plant Cell, 2004, 16(11):3045-3058. doi: 10.1105/tpc.104.025163 [23] Leivar P, Monte E, AI-Sady B, et al. The Arabidopsis phytochrome-interacting factor PIF7, together with PIF3 and PIF4, regulates responses to prolonged red light by modulating phyB levels[J]. Plant Cell, 2008, 20(2):337-352. doi: 10.1105/tpc.107.052142 [24] Leivar P, Quail P H. PIFs:Pivotal components in a cellular signaling hub[J]. Trends in Plant Science, 2011, 16(1):19-28. doi: 10.1016/j.tplants.2010.08.003 [25] Wang H, Wang HY. Phytochrome Signaling:Time to Tight up the Loose Ends[J]. Molecular Plant, 2015, 8(4):540-551. doi: 10.1016/j.molp.2014.11.021 [26] Goyal A, Karayekov E, Galvão V C, et al. Shade Promotes Phototropism through Phytochrome B-Controlled Auxin Production.[J]. Current Biology Cb, 2016, 26(24):3280. doi: 10.1016/j.cub.2016.10.001 [27] Ma D, Li X, Guo Y, et al. Cryptochrome 1 interacts with PIF4 to regulate high temperature-mediated hypocotyl elongation in response to blue light.[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(1):224-229. doi: 10.1073/pnas.1511437113 [28] Miyazaki Y, Jikumaru Y, Takase T, et al. Enhance-229ment of hypocotyl elongation by LOV KELCH PROTEIN2 production is mediated by auxin and phytochrome-interacting factors in Arabidopsis thaliana[J]. Plant Cell Reports, 2016, 35(2):455-467. doi: 10.1007/s00299-015-1896-4 [29] Shor E, Paik I, Kangisser S, et al. PHYTOCHROME INTERACTING FACTORS mediate metabolic control of the circadian system in Arabidopsis[J]. New Phytologist, 2017, 215(1):217-228. doi: 10.1111/nph.14579 [30] Monte E, Tepperman J M, AI-Sady B, et al. The phytochrome-interacting transcription factor, PIF3, acts early, selectively, and positively in light-induced chloroplast development[J]. Proceedings of the National Academy of Science of the United States of America, 2004, 101(46):16091-16098. doi: 10.1073/pnas.0407107101 [31] Niwa Y, Yamashino T, Mizuno T. The circadian clock regulates the photoperiodic response of hypocotyl elongation through a coincidence mechanism in Arabidopsis thaliana[J]. Plant & Cell physiology, 2009, 50(4):838-854. [32] Y Sakuraba, J Jeong, MY Kang, et al. Phytochrome-interacting transcription factors PIF4 and PIF5 induce leaf senescence in Arabidopsis[J]. Nature Communications, 2014, 5:4636. doi: 10.1038/ncomms5636 [33] D Todaka, K Nakashima, K Maruyama, et al. Rice phytochrome-interacting factor-like protein OsPIL1 function as a key regulator of internode elongation and induces a morphological response to drought stress[J]. Proceeding of the National Academy of Science of the United States of America, 2012, 109(39):15947-15952. doi: 10.1073/pnas.1207324109 [34] He Y, Li Y, Cui L, et al. Phytochrome B Negatively Affects Cold Tolerance by Regulating OsDREB1 Gene Expression through Phytochrome Interacting Factor-Like Protein OsPIL16 in Rice[J]. Frontiers in Plant Science, 2016, 7:1963. [35] M Kudo, S Kidokoro, T Yoshida, et al. Double overexpression of DREB and PIF transcription factors improves drought stress tolerance and cell elongation in transgenic plants[J]. Plant Biotechnology Journal, 2017, 15(4):458. doi: 10.1111/pbi.2017.15.issue-4 -




 下载:
下载: