-
沃尔巴克氏体(Wolbachia)属于变形菌门(Proteobacteria),α变形菌纲(Alphaproteobacteria),立克次氏体目(Rickettsiales),立克次氏体科(Rickettsiaceae),是广泛存在于节肢动物和线虫体内的一种胞内共生菌,在昆虫中的感染率约为66%[1]。Wolbachia只有1个种Wolbachia pipientis,但存在着不同的株系,目前已发现14个超群(supergroup),分别命名为supergroup A-F和H-O[2-4]。Wolbachia的多样性主要基于wsp、16S rRNA、ftsZ、gltA和groEL等基因序列的特征,而株系的基因型是基于多位点序列分型(MLST:multilocus sequence typing)和wsp蛋白的4个高变区的氨基酸系列来确定[5-6]。Wolbachia主要存在于宿主的生殖器官或生殖细胞中,其主要功能是调节宿主的生殖功能,包括雌性化、孤雌生殖、杀雄和胞质不亲和[7-11],使宿主种群倾向于产出感染共生菌的雌性个体,有助于自身在宿主间的扩散与传播。除生殖方面以外,Wolbachia还能以多种方式改变宿主的生物学特性,如:为昆虫宿主提供营养物质、保护宿主免受RNA病毒侵染、调控宿主种群的年龄结构和提高宿主干细胞的增殖能力等[7,12-16]。由于Wolbachia在节肢动物中的广泛分布及其对宿主的生殖调控功能,并参与了物种的快速形成与进化,因而可以用于宿主的生物学、生态与进化、发育生物学等的研究,如开发基于内共生菌的害虫和病菌控制方法[17-18],因此Wolbachia成为目前昆虫学研究的热点。
蚜虫是重要的农林业害虫,但目前关于蚜虫Wolbachia的研究较少且起步较晚,现有的研究多集中在麦蚜(Sitobion miscanthi (Takahashi))、雪松长足大蚜(Cinara cedri Mimeur)和豆蚜(Aphis craccivora Koch)等自由生活的蚜虫中[2],对瘿蚜的Wolbachia研究很少。瘿蚜是蚜虫中的一个重要类群,目前报道的有400多种,约占已知蚜虫总数的10%,它们能够刺激寄主植物形成虫瘿并在封闭的瘿内生活,具有高湿度的特殊小生境,有些种类还同时具有高单宁和高CO2浓度等[19]。瘿蚜通常对植物有害,但倍蚜虫可以形成一种特殊的虫瘿即五倍子,五倍子富含单宁,广泛应用于医药、化工、矿冶、电子和食品等行业,是重要的资源昆虫产品[19]。
本文以五倍子蚜、杨树瘿蚜和黄连木瘿蚜等9种瘿蚜为实验材料并提取总DNA,选取16S rRNA、wsp和ftsZ等作为目的基因进行基因测序及系统发育分析,探讨瘿蚜Wolbachia与其它自由生活蚜虫携带的Wolbachia的株系差异及亲缘关系,为进一步研究瘿蚜Wolbachia的株系种类及功能提供理论依据。
-
本实验所用的9种瘿蚜14个种群分别采自云南、四川、湖北和陕西的7个县市,包括肚倍蚜5个种群,角倍蚜2个种群,其它7种各1个种群,每个种群采集的虫瘿数为3、6或9个不等(表 1)。从寄主树上采集接近成熟但尚未爆裂的虫瘿,带回室内剖开,取出蚜虫,置于100%的乙醇中,-20℃保存备用。由于虫瘿内的干雌一般均来自单头干母的孤雌生殖,因此每个虫瘿内的干雌可视为1个克隆。
表 1 蚜虫样品信息和Wolbachia检测结果
Table 1. Aphid samples and their Wolbachia infections
蚜虫种类
Species寄主植物
Host plant采集地点
Collection location经度
(E)纬度
(N)海拔
/m采集时间(年-月)
Collection date检测蚜虫数
Number of tested aphid感染数
Number of Wolbachia infection肚倍蚜
Kaburagia rhusicola青麸杨
Rhus potaninii Maxim.云南昆明 102°35′ 25°02′ 2 220 2016-06 6 4 陕西城固 107°20′ 33°06′ 602 2016-06 6 0 陕西宁强 106°15′ 32°47′ 1 012 2016-07 6 0 云南盐津 104°22′ 28°06′ 860 2016-07 6 0 四川峨眉 103°26′ 29°38′ 690 2016-07 6 0 铁倍花蚜
Floraphis meitanensis红麸杨
Rhus punjabensis var. sinica四川峨眉 103°26′ 29°38′ 690 2016-09 3 0 倍蛋蚜
Schlechtendalia peitan盐肤木
Rhus chinensis Mill.四川峨眉 103°26′ 29°38′ 690 2016-08 3 0 倍花蚜
Floraphis meitanensis盐肤木
Rhus chinensis Mill.四川峨眉 103°26′ 29°38′ 690 2016-09 3 0 角倍蚜
Schlechtendalia chinensis盐肤木
Rhus chinensis Mill.四川峨眉 103°26′ 29°38′ 690 2016-10 9 1 湖北五峰 110°52′ 30°10′ 980 2016-08 9 0 滇枝瘿绵蚜
Pemphigus yangcola滇杨
Populus yunnanensis Dode云南昆明 102°45′ 25°03′ 1 920 2017-05 6 0 滇叶瘿绵蚜
Pemphigus yunnanensis滇杨
Populus yunnanensis Dode云南昆明 102°45′ 25°03′ 1 920 2017-05 9 1 杨横瘿绵蚜
Pemphigus populitransversus滇杨
Populus yunnanensis Dode云南昆明 102°45′ 25°03′ 1 930 2017-07 9 0 黄连木梳齿毛根蚜
Chaetogeoica folidentata黄连木
Pistacia chinensis Bunge云南楚雄 101°30′ 25°03′ 1 780 2016-09 3 0 -
采用Dzup基因组DNA快速抽提试剂盒(上海生工,中国),按照说明书提供的步骤提取蚜虫总DNA。取20头蚜虫,用75%酒精漂洗2~3次,清除表面污染。用去离子水浸泡1 h,去除蚜虫表面酒精。将蚜虫转移至1.5 mL离心管,用灭菌枪头破碎虫体,加入1 mL裂解液(Dzup)裂解10 min,10 000 r·min-1离心10 min,取上清液(约900 μL)至1.5 mL离心管,加入500 μL无水乙醇,混匀,8 000 r·min-1离心5 min,收集沉淀。加入1 000 μL 75%乙醇漂洗DNA 1 min(注意要让沉淀浮起),12 000 r·min-1离心2 min,弃上清液。加入Elution Buffer或去离子水稀释至200 μL,充分溶解,12 000 r·min-1离心2 min,将上清液转移至1.5 mL离心管。取2 μL DNA提取液,用1.5%的琼脂糖凝胶和核酸定量仪对DNA进行检测,经检测合格的样本置于-20℃保存备用。
-
选择16Sr RNA、wspA、groEL、ftsZ和glt作为目的基因进行扩增,引物序列及PCR反应条件见表 2,PCR反应体系为25 μL,其中模板DNA 1 μL,上下游引物各1 μL(10 nM),Taq Master Mix 12.5 μL,ddH2O 9.5 μL。将PCR扩增产物置于1.5%的琼脂糖凝胶电泳,检测扩增产物的质量。
表 2 Wolbachia检测引物及PCR反应条件
Table 2. The detection primers of Wolbachia and PCR reaction conditions
次级共生菌
Secondary symbiont基因名称
Gene name引物名称和序列(5’-3’)
Primer name and sequence(5’-3’)PCR反应条件
PCR reaction condition参考文献
ReferencesWolbachia 16S rRNA 16S281F: CTA TAG CTG ATC TGA GAG GAT 94℃3 min; 94℃ 45 s, 55℃ 60 s, 72℃ [3] 16S1372R: YGC TTC GAG TGA AAC CAA TTC 90 s, 35 circles; 72℃ 10 min 16S rRNA WspecF: CAT ACC TAT TCG AAG GGA TAG 94℃ 3 min; 94℃ 45 s, 52℃ 60 s, 72℃ [20] WspecR: AGC TTC GAG TGA AAC CAA TTC 90 s, 35 circles; 72℃ 10 min Wsp WSP81F: TGG TCC AAT AAG TGA TGA AGA AAC 94℃ 3 min; 94℃ 45 s, 52℃ 60 s, 72℃ [21] WSP691R: AAA AAT TAA ACG CTA CTC CA 90 s, 35 circles; 72℃ 10 min groEL GroELF: GGT GAG CAG TTR CAR SAA GC 94℃ 3 min; 94℃ 45 s, 55℃ 60 s, 72℃ [3] GroELR: TAR CCR CGR TCA AAY TGC ATR CCA 90 s, 35 circles;72℃ 10 min ftsZ FtsZF: GTT GGY AAA GGT GCA GCA GAA GA 94℃ 3 min; 94℃ 45 s,53℃ 60 s, 72℃ [3] FtsZR: CGY ACY CAT TTK GCT GCA GMA TCA A 90 s, 35 circles; 72℃ 10 min gltA WgltAF1: TAC GAT CCA GGG TTT GTT TCT AC 94℃ 3 min; 94℃ 45 s, 56℃ 60 s, 72℃ [3] WgltAR1: CTC ATT AGC TCC ACC GTG TG 90 s, 35 circles; 72℃ 10 min -
对基因扩增产物进行双向序列测定(上海生工),采用DNAStar Lasergene v7.1-SeqMan对测序的原始序列进行拼接,拼接序列在NCBI中BLAST检索,评估和验证测序结果的可信度。联合NCBI中的Wolbachia相关基因序列,采用MEGA 6.0和MrBayes 3.1.2进行序列比对和系统发育分析,ML树选取Kmura-Z-Parameter模型,bootstrap置信度用1 000次自导复制进行评价;BI分析采用TPM3+G模型,Lest设置替代模型nst = 6,位点速率变异模型为rates = gamma,MCMC参数如下:起始树为随机树,代数 = 3 000 000,链数 = 4,取样频率 = 100,temperature = 0.5,去除老化树burnin = 7 500(25%),最终构建50%的最大合意树(consensus),通过后验概率来检验各分支置信度。采用MEGA 6.0计算遗传距离。
-
根据瘿蚜样本的数量,本实验共提取瘿蚜DNA样品87份,经1.5%琼脂糖凝胶电泳和核酸定量仪检测后,舍弃不合格的DNA样本3份,实际检测DNA样品84份(表 1)。
-
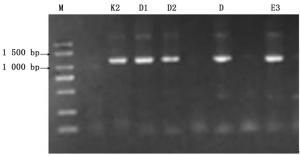
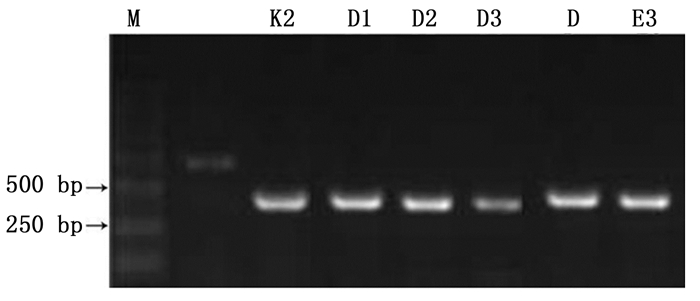
采用Wolbachia特定基因(16S rRNA、wsp、groEL、ftsZ和gltA)引物分别对9种瘿蚜的感染情况进行检测,结果仅有16S rRNA的2对引物16S 281F/16S 1372R、wspecF/wspecR分别在3种瘿蚜中扩增出清晰的DNA条带(图 1、2),分子量约为1 050 bp和440 bp,而其余4个基因引物扩增无条带。表明在检测的14个地理种群共9种瘿蚜中,有3种瘿蚜共3个种群感染有Wolbachia,分别为肚倍蚜(Kaburagia rhusicola Takagi)昆明种群、角倍蚜(Schlechtendalia chinensis (Bell))峨眉种群和滇叶瘿绵蚜(Pemphigus yunnanensis (Zhang))昆明种群,其它种类没有检测到有Wolbachia感染,综合感染率约为21.4%(表 1)。可见次级共生菌Wolbachia在这9种瘿蚜中并不普遍,其中在肚倍蚜(K. rhusicola)昆明种群的感染率为66.7%(共检测6个克隆,其中4个携带有Wolbachia)、角倍蚜(S. chinensis)峨眉种群和滇叶瘿绵蚜(P. yunnanensis)昆明种群的感染率分别为11.1%(分别检测9个克隆,各有1个携带有Wolbachia)。
-
采用16S 281F/1372R引物分别对携带有Wolbachia的肚倍蚜昆明种群、角倍蚜峨眉种群和滇叶瘿绵蚜昆明种群的感染个体进行扩增,分别选择3个扩增产物样品进行双向测序。测序结果的峰形图分布均匀,没有重叠峰,表明感染的个体均为Wolbachia单独感染。对序列拼接后,获得16S rRNA基因片段,其中肚倍蚜昆明种群1 077 bp、角倍蚜峨眉种群1 061 bp和滇叶瘿绵蚜昆明种群1 054 bp,经过统一比对和裁剪,获得的一致序列长度为1 053 bp。用同样的方法采用WspecF/WspecR引物的对上述3个蚜虫种群进行扩增,测序后获得的16S rRNA基因序列为:肚倍蚜昆明种群440 bp、角倍蚜峨眉种群440 bp和滇叶瘿绵蚜昆明种群442 bp。对上述2对引物扩增获得的序列进行比对分析,发现16S 281F/1372R的扩增序列包含了WspecF/WspecR引物的扩增序列,因此仅选择16S 281F/1372R的扩增序列进行系统发育分析。
序列比较分析表明,肚倍蚜(K. rhusicola)昆明种群和角倍蚜(S. chinensis)峨眉种群的16S rRNA序列的相似性很高,而与滇叶瘿绵蚜(P. yunnanensis)昆明种群序列有较大差异。将测定的序列在NCBI中的Blastn的比对结果,3种瘿蚜中的Wolbachia株系与多种蚜虫、蚧壳虫和螨虫中检测到的Wolbachia株系具有高度一致性(表 3)。
表 3 瘿蚜Wolbachia 16S rRNA基因序列的Blastn部分比对结果
Table 3. Blastn results of 16S rRNA gene of Wolbachia from gall aphids
宿主昆虫
Host insect最大序列一致性
Maximum sequence identity/%比对覆盖度
Query cover/%E值
E valueGenBank登录号
GenBank accession no.烟粉虱Bemisia tabaci 99 99 0.0 KF454771.1 雪松长足大蚜Cinara cedri 98 99 0.0 JN384079.1 蔷薇斑大蚜Maculolachnus submacula 98 99 0.0 JN384077.1 香蕉蚜Pentalonia nigronervosa 98 99 0.0 KJ786950.1 褐色桔蚜Toxoptera citricidus 98 98 0.0 JN384073.1 柳树大蚜Tuberolachnus salignus 97 99 0.0 JN384085.1 南美谷蚜Sipha maydis 98 98 0.0 JN384068.1 夹竹桃蚜Aphis nerii 98 97 0.0 JN384083.1 羽毛螨Torotrogla cardueli 97 99 0.0 KP114101.1 桧柏蚜Cinara fresai 98 97 0.0 JN384074.1 -
从NCBI中选取48条Wolbachia株系的16S rRNA基因序列(分别为14个超群的代表序列),与本文中获得的5条Wolbachia株系16S rRNA基因序列构建系统发育树(图 3),以烟盲蝽(Nesidiocoris tenuis (Reuter))和烟粉虱(Bemisia tabaci (Gennadius))的次级共生菌Rickettsia株系为外类群[22-23]。结果显示滇叶瘿绵蚜(P. yunnanensis)昆明种群感染的Wolbachia株系与B超群的Wolbachia株系聚成一支,ML支持率和贝叶斯后验概率较高,分别为53%和91%,且2者的遗传距离为0.003~0.019(表 4),均小于0.02,表明其感染的Wolbachia株系属于B超群。而肚倍蚜(K. rhusicola)昆明种群和角倍蚜(S. chinensis)峨眉种群感染的Wolbachia株系与O超群的Wolbachia株系(wBt_10)(寄主为烟粉虱中国种群,Asia II1)聚成一支[4],ML支持率和贝叶斯后验概率很高,分别为99%和90%,表明这两个种群感染的Wolbachia株系与wBt_10可能同属于新发现的O超群,遗传距离分析也支持上述结果。有研究认为,确定一个新超群,16S rRNA基因的遗传距离需要大于2%[24-25]。上述肚倍蚜和角倍蚜感染的4个Wolbachia株系与其他13个超群代表株系的最小遗传距离为0.034(M超群),最大遗传距离为0.102(I超群),而与O超群wBt_10株系的遗传距离仅为0.003(表 4),进一步支持这4个Wolbachia株系wBt_10同属于O超群。
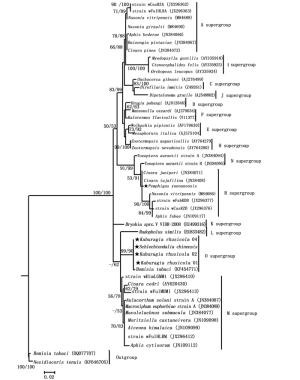
图 3 采用最大似然法(ML)和贝叶斯推论(BI)对Wolbachia株系间16S rRNA基因序列的系统发育分析
Figure 3. Phylogenetic analysis inferred from Wolbachia 16S rRNA gene sequences using Maximum Likelihood (ML) and Bayesian Inference (BI)
表 4 瘿蚜Wolbachia 16S rRNA基因序列与各超群代表序列的遗传距离比较结果
Table 4. Comparison of genetic distances between the sequences of Wolbachia 16S rRNA gene from gall aphids and the representative sequences of other supergroups
A_M84690 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 A_JN384072 0.003 B_M84686 0.048 0.051 B_JN384071 0.048 0.051 0.017 B_JN38408 0.048 0.051 0.017 0.000 C_AJ276499 0.025 0.028 0.069 0.069 0.069 D_AJ012646 0.019 0.022 0.057 0.057 0.057 0.034 E_AF179630 0.014 0.017 0.057 0.051 0.051 0.028 0.022 F_Y11377 0.014 0.017 0.051 0.051 0.051 0.028 0.011 0.017 H_AY764279 0.019 0.022 0.057 0.057 0.057 0.039 0.022 0.017 0.017 I_AY335918 0.046 0.049 0.096 0.096 0.096 0.070 0.064 0.058 0.058 0.064 I_AY335923 0.043 0.046 0.096 0.096 0.096 0.070 0.064 0.058 0.058 0.064 0.003 J_AJ548802 0.042 0.039 0.075 0.075 0.075 0.034 0.045 0.045 0.039 0.051 0.089 0.088 K_EU499316 0.042 0.045 0.067 0.067 0.067 0.057 0.045 0.045 0.040 0.045 0.089 0.089 0.063 L_EU833482 0.060 0.063 0.094 0.095 0.095 0.075 0.069 0.075 0.069 0.069 0.109 0.105 0.084 0.063 M_AY620430 0.046 0.048 0.055 0.055 0.055 0.066 0.054 0.054 0.048 0.054 0.090 0.089 0.063 0.042 0.042 M_JN384077 0.040 0.037 0.061 0.061 0.061 0.060 0.048 0.048 0.043 0.048 0.086 0.086 0.057 0.037 0.042 0.011 N_JN384094 0.054 0.057 0.037 0.020 0.020 0.075 0.063 0.051 0.057 0.057 0.102 0.102 0.075 0.060 0.085 0.046 0.052 O_KF454771 0.057 0.060 0.073 0.067 0.067 0.078 0.054 0.066 0.060 0.066 0.106 0.105 0.075 0.060 0.063 0.042 0.037 0.057 K.rhusicola 0.054 0.057 0.070 0.064 0.064 0.075 0.051 0.063 0.057 0.063 0.102 0.102 0.072 0.057 0.060 0.039 0.034 0.054 0.003 S.chinensis 0.054 0.057 0.070 0.064 0.064 0.075 0.051 0.063 0.057 0.063 0.102 0.102 0.072 0.057 0.060 0.039 0.034 0.054 0.003 0.000 P.immunis 0.048 0.051 0.019 0.003 0.003 0.069 0.057 0.051 0.051 0.057 0.096 0.096 0.075 0.067 0.095 0.055 0.061 0.020 0.067 0.064 0.064 注:各超群的代表序列用超群名称和Genbank登录号表示,新获得序列用寄主蚜虫的种名和属名表示。
Note: The representative sequence of supergroups is expressed by the supergroup name and the Genbank accession number,the newly acquired sequence is expressed by the name of the host aphid. -
Wolbachia是昆虫中广泛存在的次级共生菌,但在蚜虫中的感染率很低[26-31],其主要原因是感染的浓度较低或不同株系间的基因变异较大,加大了Wolbachia的检测难度[2],因此关于蚜虫Wolbachia的研究起步较晚,已有的研究主要集中在自由生活的蚜虫中,对瘿蚜的研究很少,在已经研究过的300多种蚜虫中,仅有2种瘿蚜[3]。本研究对14个地理种群的9种瘿蚜的Wolbachia进行检测分析,发现有3种瘿蚜各有1个种群感染有Wolbachia,综合感染率为21.4%,与自由生活的蚜虫相比,其感染率偏低[3],其中肚倍蚜(K. rhusicola)昆明种群中Wolbachia感染率为66.7%,而角倍蚜(S. chinensis)峨眉种群和滇叶瘿绵蚜(P. yunnanensis)的感染率分别为11.1%。不同种类或种群间的感染率差异很大,但基本符合已有报道的Wolbachia在蚜虫中“most or few”的感染模式[1]。
Wolbachia的株系间存在丰富的多样性,目前已发现了14个超群(supergroup),其中蚜虫中的Wolbachia属于4个超群(A、B、M和N),且M和N超群仅存在于蚜虫中[3-4]。Wang Z等[3]检测了73种分布于中国的蚜虫的Wolbachia感染情况,发现M超群占主导地位。本研究所发现的滇叶瘿绵蚜Wolbachia株系属于B超群,而肚倍蚜昆明种群和角倍蚜峨眉种群的Wolbachia株系可能属于新发现的O超群。表明瘿蚜携带的Wolbachia株系与自由生活的蚜虫有较大差别,不仅株系不同,且株系间的差异较大。肚倍蚜和角倍蚜对原生寄主植物具有高度专一性,且大部分生活阶段位于完全封闭的虫瘿内,因此通过取食共同的寄主植物或与其他蚜虫或昆虫接触而发生共生菌水平转移的机会极低,这可能是其携带的Wolbachia株系特殊的原因,其Wolbachia株系的具体归属和形成原因值得进一步研究。
目前Wolbachia的检测引物较多,但通用性不好[2,6,32],其中16S rRNA基因最为保守,扩增效果稳定,但缺点是特异性不好,有时会出现非目标条带[4];wsp基因进化速度快,适用于Wolbachia株系的分群,其缺点是扩增效果不稳定。蚜虫中的Wolbachia感染浓度低,且种系变异大,给检测分析带来很大困难,对于寄主专化性高、生活在封闭小环境的瘿蚜更是如此,因为与其他自由生活的蚜虫相比,瘿蚜携带的Wolbachia株系具有更大的变异。这可能是本研究中的4种蛋白编码基因(wsp、groEL、ftsZ和gltA基因)引物在瘿蚜中扩增没有条带的主要原因。因此,开发更加高效且特异性好的Wolbachia基因引物,将是今后蚜虫,尤其是瘿蚜Wolbachia研究的重点之一。
-
对9种瘿蚜14个种群的检测结果,有3种瘿蚜共3个种群感染有次级共生菌Wolbachia,感染率为21.4%,均为单株系感染。其中滇叶瘿绵蚜昆明种群感染的Wolbachia属于B超群,肚倍蚜昆明种群和角倍蚜峨眉种群感染的Wolbachia属于新发现的O超群。与其他自由生活的蚜虫相比,瘿蚜的Wolbachia感染率偏低,而且株系不同,且株系间的差异较大。
9种瘿蚜的Wolbachia分子检测和系统发育分析
Molecular Detection and Phylogenetic Analysis of Wolbachia in Nine Gall Aphid Species
-
摘要:
目的 以五倍子蚜、杨树瘿蚜和黄连木瘿蚜等9种瘿蚜为实验材料,探讨瘿蚜Wolbachia与其它自由生活蚜虫携带的Wolbachia的株系差异及亲缘关系,明确五倍子蚜、杨树瘿蚜和黄连木瘿蚜等9种瘿蚜的Wolbachia感染情况、株系及系统发育关系。 方法 从田间采集新鲜虫瘿,将蚜虫转移到离心管内,100%乙醇-20℃保存。采用Wolbachia的16S rRNA、wsp、ftsZ、groE和gltA基因引物,以瘿蚜的总DNA为模板进行扩增、测序和分析,联合NCBI中Wolbachia 16S rRNA相关基因序列进行系统发育分析。 结果 在检测的9种瘿蚜中,有3种瘿蚜共3个种群感染有Wolbachia,其中滇叶瘿绵蚜昆明种群的Wolbachia属于B超群,肚倍蚜昆明种群和角倍蚜峨眉种群的Wolbachia可能属于新发现的O超群。 结论 与自由生活的蚜虫相比,瘿蚜的Wolbachia感染率偏低,且株系间的差异较大。 Abstract:Objective To determine the infection and phylogenetic relationships of Wolbachia in nine gall aphids hosted on Rhus spp., Populus yunnanensis and Pistacia chinensis. Method Fresh aphid galls were collected in the field and transferred the aphids to an Eppendorf tube, and placed in 100% ethanol then deposited in -20℃. The genomic DNA was amplified and sequenced using the primers of 16S rRNA, wsp, ftsZ, groE and gltA genes. The phylogenetic relationships were analysis using Wolbachia 16S rRNA gene sequences and relevant gene sequences from NCBI. Result Three species among the nine gall aphids species were infected with Wolbachia. The strain from Pemphigus yunnanensis of Kunming population belongs to B supergroup, and strain from Kaburagia rhusicola of Kunming population and Schlechtendalia chinensis of Emei population belong to O supergroup. Conclusion Compared with the free-living aphids, Wolbachia infected in gall aphids have low infection rates and significant differences among strains. -
Key words:
- gall aphid
- / facultative symbiont
- / Wolbachia
- / phylogenetic analysis
- / supergroup
-
表 1 蚜虫样品信息和Wolbachia检测结果
Table 1. Aphid samples and their Wolbachia infections
蚜虫种类
Species寄主植物
Host plant采集地点
Collection location经度
(E)纬度
(N)海拔
/m采集时间(年-月)
Collection date检测蚜虫数
Number of tested aphid感染数
Number of Wolbachia infection肚倍蚜
Kaburagia rhusicola青麸杨
Rhus potaninii Maxim.云南昆明 102°35′ 25°02′ 2 220 2016-06 6 4 陕西城固 107°20′ 33°06′ 602 2016-06 6 0 陕西宁强 106°15′ 32°47′ 1 012 2016-07 6 0 云南盐津 104°22′ 28°06′ 860 2016-07 6 0 四川峨眉 103°26′ 29°38′ 690 2016-07 6 0 铁倍花蚜
Floraphis meitanensis红麸杨
Rhus punjabensis var. sinica四川峨眉 103°26′ 29°38′ 690 2016-09 3 0 倍蛋蚜
Schlechtendalia peitan盐肤木
Rhus chinensis Mill.四川峨眉 103°26′ 29°38′ 690 2016-08 3 0 倍花蚜
Floraphis meitanensis盐肤木
Rhus chinensis Mill.四川峨眉 103°26′ 29°38′ 690 2016-09 3 0 角倍蚜
Schlechtendalia chinensis盐肤木
Rhus chinensis Mill.四川峨眉 103°26′ 29°38′ 690 2016-10 9 1 湖北五峰 110°52′ 30°10′ 980 2016-08 9 0 滇枝瘿绵蚜
Pemphigus yangcola滇杨
Populus yunnanensis Dode云南昆明 102°45′ 25°03′ 1 920 2017-05 6 0 滇叶瘿绵蚜
Pemphigus yunnanensis滇杨
Populus yunnanensis Dode云南昆明 102°45′ 25°03′ 1 920 2017-05 9 1 杨横瘿绵蚜
Pemphigus populitransversus滇杨
Populus yunnanensis Dode云南昆明 102°45′ 25°03′ 1 930 2017-07 9 0 黄连木梳齿毛根蚜
Chaetogeoica folidentata黄连木
Pistacia chinensis Bunge云南楚雄 101°30′ 25°03′ 1 780 2016-09 3 0 表 2 Wolbachia检测引物及PCR反应条件
Table 2. The detection primers of Wolbachia and PCR reaction conditions
次级共生菌
Secondary symbiont基因名称
Gene name引物名称和序列(5’-3’)
Primer name and sequence(5’-3’)PCR反应条件
PCR reaction condition参考文献
ReferencesWolbachia 16S rRNA 16S281F: CTA TAG CTG ATC TGA GAG GAT 94℃3 min; 94℃ 45 s, 55℃ 60 s, 72℃ [3] 16S1372R: YGC TTC GAG TGA AAC CAA TTC 90 s, 35 circles; 72℃ 10 min 16S rRNA WspecF: CAT ACC TAT TCG AAG GGA TAG 94℃ 3 min; 94℃ 45 s, 52℃ 60 s, 72℃ [20] WspecR: AGC TTC GAG TGA AAC CAA TTC 90 s, 35 circles; 72℃ 10 min Wsp WSP81F: TGG TCC AAT AAG TGA TGA AGA AAC 94℃ 3 min; 94℃ 45 s, 52℃ 60 s, 72℃ [21] WSP691R: AAA AAT TAA ACG CTA CTC CA 90 s, 35 circles; 72℃ 10 min groEL GroELF: GGT GAG CAG TTR CAR SAA GC 94℃ 3 min; 94℃ 45 s, 55℃ 60 s, 72℃ [3] GroELR: TAR CCR CGR TCA AAY TGC ATR CCA 90 s, 35 circles;72℃ 10 min ftsZ FtsZF: GTT GGY AAA GGT GCA GCA GAA GA 94℃ 3 min; 94℃ 45 s,53℃ 60 s, 72℃ [3] FtsZR: CGY ACY CAT TTK GCT GCA GMA TCA A 90 s, 35 circles; 72℃ 10 min gltA WgltAF1: TAC GAT CCA GGG TTT GTT TCT AC 94℃ 3 min; 94℃ 45 s, 56℃ 60 s, 72℃ [3] WgltAR1: CTC ATT AGC TCC ACC GTG TG 90 s, 35 circles; 72℃ 10 min 表 3 瘿蚜Wolbachia 16S rRNA基因序列的Blastn部分比对结果
Table 3. Blastn results of 16S rRNA gene of Wolbachia from gall aphids
宿主昆虫
Host insect最大序列一致性
Maximum sequence identity/%比对覆盖度
Query cover/%E值
E valueGenBank登录号
GenBank accession no.烟粉虱Bemisia tabaci 99 99 0.0 KF454771.1 雪松长足大蚜Cinara cedri 98 99 0.0 JN384079.1 蔷薇斑大蚜Maculolachnus submacula 98 99 0.0 JN384077.1 香蕉蚜Pentalonia nigronervosa 98 99 0.0 KJ786950.1 褐色桔蚜Toxoptera citricidus 98 98 0.0 JN384073.1 柳树大蚜Tuberolachnus salignus 97 99 0.0 JN384085.1 南美谷蚜Sipha maydis 98 98 0.0 JN384068.1 夹竹桃蚜Aphis nerii 98 97 0.0 JN384083.1 羽毛螨Torotrogla cardueli 97 99 0.0 KP114101.1 桧柏蚜Cinara fresai 98 97 0.0 JN384074.1 表 4 瘿蚜Wolbachia 16S rRNA基因序列与各超群代表序列的遗传距离比较结果
Table 4. Comparison of genetic distances between the sequences of Wolbachia 16S rRNA gene from gall aphids and the representative sequences of other supergroups
A_M84690 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 A_JN384072 0.003 B_M84686 0.048 0.051 B_JN384071 0.048 0.051 0.017 B_JN38408 0.048 0.051 0.017 0.000 C_AJ276499 0.025 0.028 0.069 0.069 0.069 D_AJ012646 0.019 0.022 0.057 0.057 0.057 0.034 E_AF179630 0.014 0.017 0.057 0.051 0.051 0.028 0.022 F_Y11377 0.014 0.017 0.051 0.051 0.051 0.028 0.011 0.017 H_AY764279 0.019 0.022 0.057 0.057 0.057 0.039 0.022 0.017 0.017 I_AY335918 0.046 0.049 0.096 0.096 0.096 0.070 0.064 0.058 0.058 0.064 I_AY335923 0.043 0.046 0.096 0.096 0.096 0.070 0.064 0.058 0.058 0.064 0.003 J_AJ548802 0.042 0.039 0.075 0.075 0.075 0.034 0.045 0.045 0.039 0.051 0.089 0.088 K_EU499316 0.042 0.045 0.067 0.067 0.067 0.057 0.045 0.045 0.040 0.045 0.089 0.089 0.063 L_EU833482 0.060 0.063 0.094 0.095 0.095 0.075 0.069 0.075 0.069 0.069 0.109 0.105 0.084 0.063 M_AY620430 0.046 0.048 0.055 0.055 0.055 0.066 0.054 0.054 0.048 0.054 0.090 0.089 0.063 0.042 0.042 M_JN384077 0.040 0.037 0.061 0.061 0.061 0.060 0.048 0.048 0.043 0.048 0.086 0.086 0.057 0.037 0.042 0.011 N_JN384094 0.054 0.057 0.037 0.020 0.020 0.075 0.063 0.051 0.057 0.057 0.102 0.102 0.075 0.060 0.085 0.046 0.052 O_KF454771 0.057 0.060 0.073 0.067 0.067 0.078 0.054 0.066 0.060 0.066 0.106 0.105 0.075 0.060 0.063 0.042 0.037 0.057 K.rhusicola 0.054 0.057 0.070 0.064 0.064 0.075 0.051 0.063 0.057 0.063 0.102 0.102 0.072 0.057 0.060 0.039 0.034 0.054 0.003 S.chinensis 0.054 0.057 0.070 0.064 0.064 0.075 0.051 0.063 0.057 0.063 0.102 0.102 0.072 0.057 0.060 0.039 0.034 0.054 0.003 0.000 P.immunis 0.048 0.051 0.019 0.003 0.003 0.069 0.057 0.051 0.051 0.057 0.096 0.096 0.075 0.067 0.095 0.055 0.061 0.020 0.067 0.064 0.064 注:各超群的代表序列用超群名称和Genbank登录号表示,新获得序列用寄主蚜虫的种名和属名表示。
Note: The representative sequence of supergroups is expressed by the supergroup name and the Genbank accession number,the newly acquired sequence is expressed by the name of the host aphid. -
[1] Hilgenboecker K, Hammerstein P, Schlattmann P, et al. How many species are infected with Wolbachia?-a statistical analysis of current data[J]. FEMS Microbiology Letters, 2008,281(2):215-220. doi: 10.1111/fml.2008.281.issue-2 [2] Augustinos A A, Santosgarcia D, Dionyssopoulou E, et al. Detection and characterization of Wolbachia infections in natural populations of aphids is the hidden diversity fully unraveled[J]. PLoS ONE, 2011, 6(12):e28695. doi: 10.1371/journal.pone.0028695 [3] Wang Z, Su X M, Wen J, et al. Widespread infection and diverse infection patterns of Wolbachia in Chinese aphids[J]. Insect Science, 2014, 21(3):313-325. doi: 10.1111/ins.2014.21.issue-3 [4] Bing X L, Xia W Q, Gui J D, et al. Diversity and evolution of the Wolbachia endosymbionts of Bemisia (Hemiptera:Aleyrodidae) whiteflies[J]. Ecology and Evolution, 2014, 13(4):2714-2737. [5] Baldo L, Dunning Hotopp J C, Jolley K A, et al. Multilocus sequence typing system for the endosymbiont Wolbachia pipientis[J]. Applied and Environmental Microbiology, 2006, 72(11):7098-7110. doi: 10.1128/AEM.00731-06 [6] Paraskevopoulos C, Bordenstein S R, Wernegreen J J, et al. Toward a Wolbachia multilocus sequence typing system:discrimination of Wolbachia strains present in drosophila species[J]. Current Microbiology, 2006, 53(5):388-395. doi: 10.1007/s00284-006-0054-1 [7] Werren J H, Baldo L, Clark M E. Wolbachia: master manipulators of invertebrate biology[J]. Nature Reviews Microbiology, 2008, 6(10):741-751. doi: 10.1038/nrmicro1969 [8] Stouthamer R, Breeuwert J A, Luck R F, et al. Molecular identification of microorganisms associated with parthenogenesis[J]. Nature, 1993,361(6407):66-68. doi: 10.1038/361066a0 [9] Yen J H, Barr A R. New hypothesis of the cause of cytoplasmic incompatibility in Culex pipiens L[J]. Nature, 1971,232(5313):657-658. doi: 10.1038/232657a0 [10] Gdd H, Men M, Walker L E. The importance of cytoplasmic male killing elements in natural populations of the two spot ladybird, Adalia bipunctata (Linnaeus) (Coleoptera:Coccinellidae)[J]. Biological Journal of the Linnean Society, 1993, 49(2):195-202. doi: 10.1111/bij.1993.49.issue-2 [11] Rigaud T, Souty-Grosset C, Raimond R, et al. Feminizing endocytobiosis in the terrestrial crustacean Armadillidium vulgare Latr. (Isopoda):Recent acquisitions[J]. Endocytobiosis and Cell Research, 1991, 7(3):259-273. [12] Moreira L A, Iturbe-Ormaetxe I, Jeffery J A, et al. Wolbachia symbiont in Aedes aegypti limits infection with dengue, Chikungunya, and Plasmodium[J]. Cell, 2009, 7(139):1268-1278. [13] McMeniman C J, Lane R V, Cass B N, et al. Stable introduction of a life-shortening Wolbachia infection into the mosquito Aedes aegypti[J]. Science, 2009,323(5910):141-144. doi: 10.1126/science.1165326 [14] Hedges L M, Brownlie J C, O'Neill S L, et al. Wolbachia and virus protection in insects[J]. Science, 2008,322(5902):702. doi: 10.1126/science.1162418 [15] Hosokawa T, Koga R, Kikuchi Y, et al. Wolbachia as a bacteriocyte-associated nutritional mutualist[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 2(107):769-774. [16] Fast E M, Toomey M E, Panaram K, et al. Wolbachia enhance Drosophila stem cell proliferation and target the germline stem cell niche[J]. Science, 2011,334(6058):990-992. doi: 10.1126/science.1209609 [17] Bourtzis K. Wolbachia-based technologies for insect pest population control[J]. Advances in Experimental Medicine and Biology, 2008,627(1):104-113. [18] Saridaki A, Bourtzis K. Wolbachia: more than just a bug in insects genitals[J]. Current Opinion in Microbiology, 2010, 13(1):67-72. doi: 10.1016/j.mib.2009.11.005 [19] 杨子祥.五倍子高产培育技术[M].北京:中国林业出版社, 2011. [20] Werren J H, Windsor D M. Wolbachia infection frequencies in insects:evidence of a global equilibrium[J]. Proceeding of the Royal Society London B:Biological Sciences, 2000,267(1450):1277-1285. doi: 10.1098/rspb.2000.1139 [21] Zhou W, Rousset F, O'Neill S. Phylogeny and PCR-based classification of Wolbachia strains using wsp gene sequences[J]. Proceeding of the Royal Society London B:Biological Sciences, 1998,265(1395):509-515. doi: 10.1098/rspb.1998.0324 [22] Gottlieb Y, Ghanim M, Chiel E, et al. Identification and localization of a Rickettsia sp. in Bemisia tabaci (Homoptera:Aleyrodidae)[J]. Applied & Environmental Microbiology, 2006, 72(5):3646-3652. [23] Caspi F A, Inbar M, Steinberg S, et al. Characterization of the symbiont Rickettsia in the mirid bug Nesidiocoris tenuis (Reuter) (Heteroptera:Miridae)[J]. Bulletin of Entomological Research, 2014,104(6):681-688. doi: 10.1017/S0007485314000492 [24] Stouthamer R, Breeuwert J A, Luck R F, et al. Molecular identification of microorganisms associated with parthenogenesis[J]. Nature, 1993,361(6407):66-68. doi: 10.1038/361066a0 [25] Breeuwer J A, Stouthamer R, Barns S M, et al. Phylogeny of cytoplasmic incompatibility microorganisms in the parasitoid wasp genus Nasonia (Hymenoptera:Pteromalidae) based on 16S ribosomal DNA sequences[J]. Insect Molecular Biology, 2010, 1(1):25-36. [26] Sintupachee S, Milne J R, Poonchaisri S, et al. Closely related Wolbachia strains within the pumpkin arthropod community and the potential for horizontal transmission via the plant[J]. Microbial Ecology, 2006, 51(3):294-301. doi: 10.1007/s00248-006-9036-x [27] Wang Z, Shen Z R, Song Y D, et al. Distribution and diversity of Wolbachia in different populations of the wheat aphid Sitobion miscanthi (Hemiptera:Aphididae) in China[J]. European Journal of Entomology, 2009,106(1):49-55. doi: 10.14411/eje.2009.007 [28] Jeyaprakash A, Hoy M A. Long PCR improves Wolbachia DNA amplification:wsp sequences found in 76% of sixty-three arthropod species[J]. Insect Molecular Biology, 2000, 9(4):393-405. doi: 10.1046/j.1365-2583.2000.00203.x [29] 江幸福, 王蕾, 张蕾, 等.蔬菜蚜虫感染沃尔巴克氏体(Wolbachia)的分子检测[J].植物保护, 2009, 35(4):63-65. doi: 10.3969/j.issn.0529-1542.2009.04.013 [30] Duron O, Bouchon D, Boutin S, et al. The diversity of reproductive parasites among arthropods:Wolbachia do not walk alone[J]. BMC Biology, 2008, 6(1):1-12. doi: 10.1186/1741-7007-6-1 [31] West S A, Cook J M, Werren J H, et al. Wolbachia in two insect host-parasitoid communities[J]. Molecular Ecology, 1998, 7(11):1457-1465. doi: 10.1046/j.1365-294x.1998.00467.x [32] Casiraghi M, Bordenstein S R, Baldo L, et al. Phylogeny of Wolbachia pipientis based on gltA, groEL and ftsZ gene sequences:clustering of arthropod and nematode symbionts in the F supergroup, and evidence for further diversity in the Wolbachia tree[J]. Microbiology, 2005,151(12):4015-4022. doi: 10.1099/mic.0.28313-0 -




 下载:
下载: