-
MYB蛋白是植物体内最大的转录因子家族之一,几乎存在于所有植物中,参与调控植物体内众多基因的转录[1]。依据MYB基序重复种类和数目的不同,将植物MYB转录因子家族分为四类:4R-MYB、3R-MYB、1R-MYB/MYB-related、R2R3-MYB。4R-MYB由4个R1/R2重复组成,它是植物MYB转录因子中最小的一类,仅在拟南芥(Arabidopsis thaliana)、杨树(Populus trichocarpa)等模式植物中有所发现,且编码的蛋白质很少;3R-MYB,由R1、R2、R3组成,普遍存在于大多数的真核生物中;1R-MYB/MYB-related,含有单个重复的或不完整的MYB基序[1-3]。在植物MYB家族中,R2R3-MYB是最重要的亚家族,其成员数最多。在模式植物拟南芥中,R2R3-MYB类转录因子数目约占整个MYB转录因子家族的64.3%。基于DNA结合区域和转录调控区域的特异性和多样性,R2R3-MYB类转录因子的功能具有多样化[4],成为当前研究最广泛的MYB蛋白。
近年来,R2R3-MYB在植物发育、代谢以及抗逆等方面的作用引起了广泛关注,如细胞形态的建成和分化、植物生长发育的信号转导、次级代谢的调控、生物和非生物胁迫的应答以及细胞周期的调控等[1, 4-9],还参与植物合成和储存花青素的过程[5, 10]。AtMYB2基因可以被脱水素和盐胁迫诱导表达[11],AtMYB62基因参与磷胁迫应答[12],AtMYB96基因通过ABA信号途径参与干旱胁迫应答[13],AtMYB41、102基因参与机械损伤和渗透胁迫应答[14-15],水稻(Oryza sativa)OsMYB3R-2转基因植株可以提高对低温、干旱和高盐的耐受性[16-17],大豆(Glycine max)GmMYB72、96、117基因在ABA、盐、干旱和低温胁迫下被诱导表达[18],麻风树(Jatropha curcas)JcMYB2基因在低温、盐和茉莉酸胁迫条件下可以被明显地诱导表达,转基因烟草(Nitotiana tobacum)的低温和干旱耐受性明显提高[9],52个棉花(Gossypium raimondii)GrMYB基因在干旱和盐胁迫条件下在叶子和根中差异表达明显[7],大白菜(Brassica rapa ssp. pekinensis)BrMYB210、137、88、154、222基因在低温和渗透胁迫条件下明显上调表达[19],AtMYB12、111基因参与黄酮醇的生物合成[20],AtMYB3、4、7、32基因的表达使拟南芥中花青素的合成受到抑制[21]。AtMYB123、21、24基因表达可促进拟南芥中花青素的积累[22],且在蔷薇(Rosa)、百合(Lilium brownii)、西红柿(Lycopersicon esculentum)等植物中都发现了MYB基因参与次生代谢调控[23]。
关于兰科植物R2R3-MYB转录因子对兰花花色的调控研究已经开展[10, 24-25]。许传俊等发现,白色蝴蝶兰花(Phalaenopsis)品种‘空港枫叶’和黄色花品种‘富乐夕阳’的花中花色素苷含量较低,MYB基因表达量也较低[26]。Wu等从杂交石斛兰(Dendrobium)‘Woo Leng’花组织的cDNA文库中,筛选到6个DwMYBs全长cDNA克隆,其中,4个是典型的R2R3-MYB类转录因子(DwMYB1、2、8、10)。DwMYB4基因仅在花中特异性表达,DwMYB9基因在成熟花和花序中高表达,在发育的花芽中表达量较低[27]。
全基因组分析是鉴定基因家族成员、阐明其生物学作用的首要步骤和有效途径,借助于该分析手段,已在多种植物中开展基因组范围内MYB转录因子家族预测与潜在功能分析[4-5],但关于蝴蝶兰MYB转录因子家族的全基因组分析尚未见报道。蝴蝶兰属植物小兰屿蝴蝶兰(P. equestris)基因组测序的完成[11]为该物种MYB类转录因子家族(PeMYBs)的全基因组预测和生物信息学分析提供了宝贵资源。鉴于R2R3-MYB转录因子在植物中生命调控功能的多样性及其研究的广泛性,本研究在小兰屿蝴蝶兰全基因组范围内开展MYB转录因子家族预测的基础上重点对R2R3-MYB转录因子亚家族成员进行生物信息学及表达分析,以期为蝴蝶兰属R2R3-MYB类转录因子家族功能验证提供数据基础,将有助于蝴蝶兰的分子育种和种质创新。
-
小兰屿蝴蝶兰基因组数据库:https://www.ncbi.nlm.nih.gov/genome/?term=Phalaenopsis+equestris;拟南芥基因组数据库:http://arabidopsis.org/;Pfam数据库:http://pfam.xfam.org/。
-
氨基酸序列分析软件:BLASTP,clustalW;系统进化分析软件:MEGA 5.0;基因表达热图绘制软件:HemI(Heatmap Illustrator)1.0;图像处理软件:PhotoShop。
-
下载小兰屿蝴蝶兰全基因组蛋白编码序列、已报道的16条蝴蝶兰R2R3-MYB基因家族蛋白序列(PeMYB1-16)[6]和拟南芥AtMYB家族成员[2]的蛋白序列。利用BLASTP工具进行比对搜索(缺省参数设置),手动去除错配序列。
-
将BLASTP比对获得的潜在PeMYBs蛋白序列文件保存为FASTA的格式,利用Pfam数据库验证PeMYBs蛋白序列的保守结构域MYB(缺省参数设置)及其个数。
-
利用软件ClustalW 2.1对R2R3-PeMYBs氨基酸序列MYB结构域进行多重序列比对分析(缺省参数设置),利用在线软件weblogo(http://weblogo.berkeley.edu/logo.cgi)绘制保守结构域R2-domain和R3-domain的多重序列logos,手动标记结构域特征位点;从小兰屿蝴蝶兰全基因组数据库[11]获取PeMYBs基因注释信息, 统计分析PeMYBs基因“外显子-内含子”结构。
-
首先,用ClustalW2.1软件对预测获得的R2R3-PeMYBs和拟南芥AtMYBs蛋白序列进行比对(缺省参数设置),利用MEGA5.0软件采用邻接法(Neighbor-joining Method)对上述文件进行进化树分析。Bootstrap设置为1 000,最后生成无根树。
-
PeMYBs基因表达数据来自小兰屿蝴蝶兰根、茎、叶和花4个组织转录组数据库(http://www.nature.com/ng/journal/v47/n3/full/ng0315-304a.html)[11]。将RPKM (Reads per kilobase per million mapped reads)值取log2对数输入软件HemI1.0绘制PeMYBs基因表达热图。
-
分别以已报道的蝴蝶兰PeMYB1-16(GenBank登录号KF769466- KF769481)和拟南芥AtMYBs氨基酸序列为query序列,利用BLASTP软件从小兰屿蝴蝶兰的蛋白库中搜索相似度较高的PeMYBs序列(E-value<0.001)。利用Pfam数据库进行保守结构域检测,综合已报道的16条PeMYBs基因共获得125条含有MYB保守结构域的PeMYBs氨基酸序列,其中,27条含有R1-MYB保守结构域,96条含有R2R3-MYB保守结构域,2条含有R1R2R3-MYB保守结构域,未发现含有4R-MYB保守结构域的PeMYBs氨基酸序列。已知拟南芥含有196条AtMYBs氨基酸序列[2],水稻含有201条OsMYBs [12],毛果杨含有207条PtMYBs[4],大约是小兰屿蝴蝶兰PeMYBs数量的1.5倍;葡萄含有114条VvMYBs序列[5],和小兰屿蝴蝶兰PeMYBs数量相近;而在小立碗藓(Physcomitrella patens)中检测到95条PpMYBs序列,略少于小兰屿蝴蝶兰PeMYBs数量(表 1)。
表 1 植物MYB转录因子亚家族成员统计
Table 1. MYB transcription factors in six plant species
Species物种 MYB protein classes MYB蛋白亚家族 1R-MYB R2R3-MYB 3R-MYB 4R-MYB Eudicot Arabidopsis thaliana[2] 64 126 5 1 Populus trichocarpa[3-4] n.d 202 5 - Vitis vinifera[5] n.d 108 5 1 Monocot Oryza sativa[4] 70 125 5 1 Phalaenopsis equestris 27 96 2 - Moss Physcomitrella patens# 31 61 2 1 注:n.d未检测到(not determined)。
#:the Moss Transcription Factor Database (http://planttfdb.cbi.pku.edu.cn). -
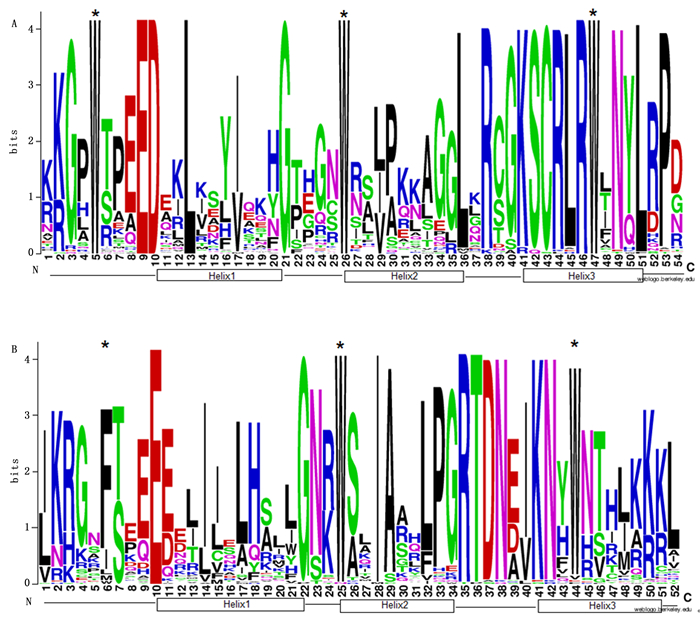
本研究重点对小兰屿蝴蝶兰R2R3-MYB类转录因子进行生物信息学分析。利用ClustalW 2.1软件将预测获得的小兰屿蝴蝶兰R2R3-MYB类转录因子编码氨基酸序列保守结构域进行多重序列比对,结果显示:预测获得的小兰屿蝴蝶兰R2R3-MYB类转录因子均含有保守的色氨酸残基和2个MYB基序R2和R3,每个MYB基序分别形成三段螺旋结构的MYB转录因子特有的结构特征(图 1),与所报道的植物R2R3-MYB类转录因子保守结构域特点相一致。有研究认为,R2和R3是MYB识别DNA序列所必需的,其中,R3亚基C端上的螺旋(Helix)能与顺式作用元件中的核心序列特异结合[1-3]。植物MYB基序的Helix2和Helix3形成“螺旋-转角-螺旋”(helix-turn-helix,HTH)结构,与Helix1形成一个具有疏水核心的三维HTH结构域,其中,Helix3能精确地与特定DNA序列结合,从而体现出较高的特异性[1-3]。小兰屿蝴蝶兰R2R3-MYB类转录因子R3基序中保守的Helix3也可能精确地与特定DNA序列结合,从而体现出较高的调控特异性。
图 1 小兰屿蝴蝶兰R2R3-MYB家族蛋白序列保守结构域
Figure 1. R2-and R3-MYB repeats are highly conserved across all R2R3-MYB proteins in the P. equestris
对小兰屿蝴蝶兰R2R3-MYB类转录因子基因编码的氨基酸序列长度进行了统计,结果详见表 2。编码氨基酸序列长度由129 aa (PeMYB85)-563 aa (PeMYB 42),平均长度为275 aa。在基因家族扩张的过程中,复制事件的发生及其平常而有效。综合分析小兰屿蝴蝶兰基因组注释结果和ClustalW 2.1软件对R2R3-MYB类转录因子编码的氨基酸序列进行比对结果发现:小兰屿蝴蝶兰R2R3-MYB类转录因子间存在基因片段复制现象,例如PeMYB81与PeMYB87序列分别位于不同的scaffolds序列,但序列之间存在重复片段,类似的片段复制现象还发生在PeMYB62与PeMYB94,PeMYB64与PeMYB97,PeMYB77、78与PeMYB79,PeMYB17与PeMYB95等序列间。
表 2 小兰屿蝴蝶兰R2R3-MYB类转录因子信息统计
Table 2. R2R3-MYB sequence characteristics of Phalaenopsis equestris
基因名称
Gene name基因ID
Gene ID长度
Length/aa内含子数量
IntronsPeMYB01 KF769466 269 - PeMYB02 PEQU_10971 303 2 PeMYB03 PEQU_30611 294 2 PeMYB04 PEQU_23598 211 1 PeMYB05 KF769470 169 - PeMYB06 PEQU_22029 262 1 PeMYB07* PEQU_03393 249 1 PeMYB08 PEQU_10762 219 2 PeMYB09 PEQU_20333 409 2 PeMYB10 PEQU_05119 353 2 PeMYB11 PEQU_09551 238 2 PeMYB12 PEQU_19346 245 3 PeMYB13 PEQU_14300 220 2 PeMYB14 KF769479 220 - PeMYB16 PEQU_09064 377 3 PeMYB17 PEQU_00368 324 2 PeMYB18 PEQU_00728 234 2 PeMYB19 PEQU_02109 198 1 PeMYB20 PEQU_02591 303 2 PeMYB21 PEQU_03213 267 2 PeMYB22 PEQU_04692 322 2 PeMYB23 PEQU_04858 288 2 PeMYB24 PEQU_05034 325 1 PeMYB25 PEQU_05643 295 2 PeMYB26 PEQU_05683 292 2 PeMYB27 PEQU_05903 288 2 PeMYB28 PEQU_05918 312 1 PeMYB29* PEQU_06231 344 1 PeMYB30 PEQU_06488 201 2 PeMYB 31 PEQU_06600 286 2 PeMYB32 PEQU_06922 241 3 PeMYB33 PEQU_07074 313 2 PeMYB34 PEQU_07150 303 2 PeMYB35 PEQU_07382 338 2 PeMYB36 PEQU_07503 326 1 PeMYB37 PEQU_07637 347 2 PeMYB38 PEQU_08801 287 2 PeMYB39 PEQU_09074 229 1 PeMYB40* PEQU_09152 172 1 PeMYB41 PEQU_09363 304 2 PeMYB42 PEQU_09491 563 2 PeMYB43* PEQU_09503 289 11 PeMYB44 PEQU_10362 247 2 PeMYB45* PEQU_10550 287 0 PeMYB46* PEQU_10664 251 2 PeMYB47 PEQU_10683 225 2 PeMYB48* PEQU_10866 237 1 PeMYB49 PEQU_11292 315 3 PeMYB50 PEQU_12592* 271 0 PeMYB51 PEQU_12801* 204 2 PeMYB52 PEQU_13544* 239 2 PeMYB53 PEQU_14547 294 1 PeMYB54 PEQU_14880 245 2 PeMYB55 PEQU_14919 221 2 PeMYB56 PEQU_15257 233 0 PeMYB57 PEQU_15899 180 2 PeMYB58 PEQU_16171 258 1 PeMYB59 PEQU_16389* 231 2 PeMYB60 PEQU_17021* 207 1 PeMYB61 PEQU_17178* 227 1 PeMYB62 PEQU_17258* 239 2 PeMYB63 PEQU_18431 233 2 PeMYB64 PEQU_18783 232 2 PeMYB65 PEQU_18879 358 2 PeMYB66 PEQU_19266 345 2 PeMYB67 PEQU_20514 320 2 PeMYB68 PEQU_21248* 309 2 PeMYB69 PEQU_21694 320 1 PeMYB70 PEQU_23041* 333 1 PeMYB71 PEQU_23940* 312 1 PeMYB72 PEQU_24421 244 2 PeMYB73 PEQU_25920 260 1 PeMYB74 PEQU_29058 310 2 PeMYB75 PEQU_29143 244 1 PeMYB76 PEQU_29475 250 2 PeMYB77 PEQU_30835 302 2 PeMYB78 PEQU_30836 307 2 PeMYB79 PEQU_30837 313 2 PeMYB80 PEQU_30955 322 2 PeMYB81 PEQU_31568 340 2 PeMYB82 PEQU_32696 380 1 PeMYB83 PEQU_32963 250 2 PeMYB84 PEQU_33555* 188 2 PeMYB85 PEQU_33827 129 2 PeMYB86 PEQU_33908* 310 2 PeMYB87 PEQU_34202 340 2 PeMYB88 PEQU_34697 255 2 PeMYB89 PEQU_36816 290 2 PeMYB90 PEQU_36904 276 1 PeMYB91 PEQU_37625 278 1 PeMYB92 PEQU_39789 270 2 PeMYB93 PEQU_40332 260 1 PeMYB94 PEQU_40608 209 1 PeMYB95 PEQU_40802 214 1 PeMYB96 PEQU_41555 239 2 PeMYB97 PEQU_42161 260 2 注:*氨基酸序列中包含保守的EAR-motif. * There is conserved EAR-motif in the amino acid sequence. 本文还对其内含子的数量进行了统计和简要的分析。由于PeMYB02-04、06-13、16基因序列及其详细的基因结构注释信息在小兰屿蝴蝶兰基因组数据库中均可检索到,在表 2中对其内含子数量进行了标注,但PeMYB01、05、14基因序列在小兰屿蝴蝶兰基因组数据库中暂未检索到,只在NCBI中检索到其基因序列,所以,在表 2中未对其内含子数量进行统计。表 2表明:3个R2R3-MYB转录因子不含内含子,26个含有1个内含子,59个含有2个内含子,4个含有3个内含子,而PeMYB43(PEQU_09503)基因含有11个内含子,数量最多,结构最复杂。
-
利用邻接法分析小兰屿蝴蝶兰R2R3-MYB转录因子之间的系统发育,并按类别将其分别与拟南芥R2R3-MYB转录因子进行聚类,依据拟南芥的分类标准[2]将小兰屿蝴蝶兰R2R3-MYB类转录因子划分为20个亚群,命名为S1-S20(图 2),分别对应拟南芥中相应类群,其中,S5、S11、S12、S13、S19亚群成员数较多,分别为9、13、10、10、12个。除S14外,其它19个亚群同时包含小兰屿蝴蝶兰与拟南芥R2R3-MYB类转录因子,表明这些亚群的基本特征在小兰屿蝴蝶兰与拟南芥分离之前就已经形成,每个亚群成员数目不同也许是由于基因复制或基因丢失的结果。未被划分亚群的拟南芥R2R3-MYB类转录因子有16个,这些AtMYBs在杨树中能检测到划分为同一亚群的同源基因[4],这可能是这些基因亚群在小兰屿蝴蝶兰中发生了基因丢失现象。大部分小兰屿蝴蝶兰R2R3-MYB类转录因子亚群分类与拟南芥的R2R3-MYB相应亚群分类情况吻合,有几个亚群把拟南芥中的2或多个亚群合在了1个小兰屿蝴蝶兰亚群里(如S13),这表明在拟南芥与小兰屿蝴蝶兰R2R3-MYB类转录因子之间有较高的保守性,也稍有差异。在其他植物R2R3-MYB类转录因子家族中也检测到类似现象[3-4]。

图 2 小兰屿蝴蝶兰R2R3-MYB家族成员系统进化分析
Figure 2. Phylogenetic relationships and subgroup designations of PeMYBs and AtMYB
基因系统进化树上位于同一分支的序列往往具有较近的亲缘关系。小兰屿蝴蝶兰R2R3-MYB转录因子内部存在同源性较高的旁系同源序列(自展值>95%),如PeMYB01/PeMYB13/PeMYB14,PeMYB25/ PeMYB26,PeMYB81/PeMYB87,PeMYB56/PeMYB18,PeMYB62/PeMYB94和PeMYB01/PeMYB91等。小兰屿蝴蝶兰基因组中也存在一些R2R3-MYB类转录因子与拟南芥同源性较高的直系同源序列(自展值>95%),如PeMYB56/AtMYB16/AtMYB106,PeMYB38/AtMYB35,PeMYB29/AtMYB67,PeMYB68/AtMYB91和PeMYB42/AtMYB88/AtMYB124等。不同兰科植物之间同源性较高的序列有PeMYB16/PeMYB65/DwMYB1,PeMYB11/OgMYB1/PsMYB(P.schilleriana MYB),PeMYB08/DwMYB8,等。研究发现,同源性较高的基因具有很高的序列相似性,并可能具有类似的功能[4, 10],本文通过对PeMYBs亚家族内及与AtMYBs亚家族间序列同源性的分析可以为预测PeMYBs的潜在功能提供潜在数据基础。
-
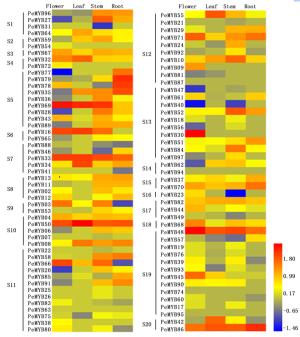
为了解小兰屿蝴蝶兰R2R3-MYB家族基因在不同器官中的表达情况,笔者利用小兰屿蝴蝶兰4种器官(花、叶、根、茎)的转录组数据标准化值进行比较分析,检测到88个PeMYBs基因在4种器官中表达,图 3表明:各基因在不同器官中的表达情况各异。各器官中表达量相对较高的基因有:PeMYB30、48、69、66、50、86等基因在花中表达量相对较高,PeMYB16、33、34、42、48、50、69、86等基因在叶中表达量相对较高,PeMYB08、10、33、48、50、52、69、84、86等基因在茎中表达量相对较高,PeMYB33、35、48、50、70、71、78、79、86等基因在根中表达量相对较高,推测它们可能在小兰屿蝴蝶兰的生长发育过程中有着某些重要作用。39个PeMYBs基因在所有4种器官中均表达,其它48个基因在不同器官中有特异性不表达现象,其中,20个基因只在叶组织中未检测到表达,2个基因(PeMYB53、57)只在花组织中未检测到表达,7个PeMYBs基因(PeMYB16-19、24、90、93)只在根组织中未检测到表达,茎中未出现PeMYB基因特异性不表达现象;还有一些基因呈现组织器官特异性表达特点,如PeMYB09、30、38、54、72五个基因只在花中检测到表达,而且PeMYB09、30两个基因在花中特异性高表达,PeMYB07、17-19、21、22、25、26、31基因只在茎组织中检测到低量表达,PeMYB41只在根组织中检测到低量表达,在其它3种组织中均未检测到表达。研究发现,在某些组织中高表达或者特异性表达的基因可能参与相应组织特定发育时期的调控[4-6, 10]。在各亚群中均检测到相对高或低或未表达的PeMYBs基因,且未呈现亚群特异性表达现象。
-
通过生物信息学分析,本文对小兰屿蝴蝶兰MYB转录因子超家族进行全基因组范围的预测分析,综合NCBI中已登录的PeMYB转录因子,共获得125个PeMYBs转录因子。研究报道的拟南芥、水稻、毛果杨、葡萄、小立碗藓基因组分别有196、201、207、114和95个MYB转录因子成员[2-3, 5, 29]。目前从小兰屿蝴蝶兰基因组预测获得PeMYBs基因的数量远少于拟南芥、水稻和毛果杨,与葡萄MYBs的数量相近,大于小立碗藓MYBs的数量。对比分析NCBI数据库的基因组数据,拟南芥、水稻、毛果杨、葡萄和小立碗藓分别有33 583、30 534、45 942、41 208和35 934个蛋白编码基因,小兰屿蝴蝶兰基因组29 431个蛋白编码基因,说明该基因家族成员的数量可能不与物种基因组编码蛋白基因数量成正相关。研究发现,全基因组复制和R2R3-MYB转录因子的选择性扩增可能是导致杨树MYB转录因子家族扩张的主要原因[3-4, 30]。目前,R2R3-MYB转录因子对模式植物拟南芥等花色与逆境胁迫应答调控机理的研究已较为深入,但对蝴蝶兰MYB转录因子的研究则刚起步。在拟南芥、葡萄和苹果上研究表明,R2R3-MYB的调控功能存在多样性和冗余性[4-5]。可见,鉴定出与花青素合成、逆境胁迫应答有关新的R2R3-MYB转录因子,加强R2R3-MYB表达的研究,并验证其功能,阐明其分子作用机制,将有助于蝴蝶兰的分子育种和种质创新。
系统进化树中在同一分支同源性较高的基因一般具有很高的序列相似性,并可能具有类似的功能[4, 10, 31-32]。小兰屿蝴蝶兰PeMYB01、02、11、12、13、14基因与参与花色调控的OgMYB1和PsMYB基因在系统进化树中聚在同一亚群S8中,具有较高的同源性。研究发现,文心兰中分离到R2R3-MYB转录因子OgMYB1基因可以诱导花青素苷的合成,对于花器官中红色表型具有关键作用[33]。PsMYB基因在西蕾丽蝴蝶兰紫色花朵和P.everspring蝴蝶兰紫色斑点花瓣中表达,但在白色花P.amabilis蝴蝶兰中不表达[24]。Hsu等研究发现,PeMYB2、11、12基因与OgMYB1和PsMYB基因功能类似,也参与花色调控[10]。石斛兰中克隆获得的DwMYB1、2、8、10基因参与花发育与花色调控[27]。这些花色调控基因氨基酸序列R3结构域均含有保守的“[D/E]Lx2[R/K]x3Lx6Lx3R”基序,可以与R/B类bHLH蛋白协同作用,激活花青素编码基因的启动子,从而调控花青素的生物合成[24]。在系统发育树中,这些DwMYBs基因分别在S10、S12亚群和PeMYB6-8、PeMYB9、10聚类在同一分支,这些基因氨基酸序列中均具有保守的“[D/E]Lx2[R/K]x3Lx6Lx3R”基序,说明PeMYB6-10基因也可通过与R/B类bHLH蛋白协同作用调控花青素的生物合成。PeMYB71与AtMYB26基因聚类在一起(位于S12亚群),AtMYB26表达产物可以显著激活花粉裂开过程中花药次级细胞壁增厚[14],在水稻、杨树、玉米等植物中均发现存在AtMYB26基因的直系同源物,说明AtMYB26基因参与生殖调控的功能可能在进化过程中非常保守[4]。在小兰屿蝴蝶兰(花、叶、茎、根)4种组织中,PeMYB71在花中的表达量相对最高,在其它3种营养器官中表达量相对较低,可能与其同源基因AtMYB26的生殖调控的功能相一致。
EAR (ethylene-responsive elementbinding factor-associated amphiphilic repression)转录抑制子在调节植物防御和逆境胁迫反应的过程中发挥重要作用[34]。EAR基序(LxLxL)是非常保守的氨基酸序列,含有EAR基序的转录因子通过负调控生长发育和胁迫应答相关基因的表达,从而使植物在不同环境下保持正常的生理状态[35]。AtMYB3、4、6、7、32基因和金鱼草(Antirrhinum majus)AmMYB308和330、番茄LeMYB27、大麦(Hordeum vulgare)HvMYB1和5基因编码的氨基酸中均包含有“LxLxL” motif[36],马铃薯MYB家族24个成员包含有“LxLxL” EAR-motif[8]。AtMYB3、4、6、7、32基因参与胁迫应答调控[9]。另外,AtMYB4和32基因还参与拟南芥花粉正常发育调控[36],水稻和拟南芥MYBH基因C端EAR基序的表达促进植物衰老,缺失EAR-mitof的突变体与野生型相比在相同的生长发育阶段未出现衰老症状[37]。本研究通过手动搜索发现21个小兰屿蝴蝶兰R2R3-MYB氨基酸序列包含“LxLxL”基序,其中,PeMYB07与AtMYB3、4、6、7、32基因编码的氨基酸序列在系统进化树中聚在S10亚群同一分枝。
研究发现,在氧化条件下,2个Cys残基在分子内部能够形成二硫键,二硫键的形成可以防止DNA结合域被氧化,保证其正常的生物活性[1]。本文中,S9亚群中PeMYB03、53与AtMYB11、12、111基因编码的氨基酸序列高度相似,在系统进化树中聚在同一分枝。在这5个基因编码的氨基酸序列的R2基序中均有一段含有4个半胱氨酸残基(Cys)的保守序列,可能和氧化胁迫响应调控相关,保证在胁迫条件下维持正常的生命活动。PeMYB54、59、PeMYB04-08、50基因分别与AtMYB13、15和AtMYB3、4、8基因聚类为S2和S10亚群,表明其同源关系较近。研究发现,拟南芥的这些基因可以受多种胁迫诱导其表达,参与胁迫应答调控[3, 38-39];与这些拟南芥基因同源关系较近的小兰屿蝴蝶兰基因也可能参与相应胁迫应答调控。
-
本研究在全基因组范围内预测获得125条含有MYB保守结构域的PeMYBs蛋白序列,其中,R2R3-MYB转录因子96条,对其基因结构、系统进化及在不同器官中的表达情况进行分析,并对部分PeMYB转录因子可能的调控功能进行了预测。小兰屿蝴蝶兰R2R3-MYB家族的生物信息学分析将为细致研究蝴蝶兰MYB转录因子调控植物生长发育和逆境胁迫响应的分子机理提供一定的数据基础。
小兰屿蝴蝶兰R2R3-MYB转录因子分析
Genome-wide Analysis of R2R3-MYB Transcription Factors in Phalaenopsis equestris
-
摘要:
目的 本研究为了探讨在植物发育和抗逆过程扮演着重要角色的MYB转录因子的潜在功能。 方法 利用拟南芥MYB转录因子家族蛋白序列(AtMYBs)和已报道的蝴蝶兰R2R3-MYB转录因子家族蛋白序列(PeMYBs),采用本地化软件BLASTP对小兰屿蝴蝶兰全基因组数据库进行搜索,并利用Pfam数据库验证MYB结构域,获得小兰屿蝴蝶兰MYB转录因子家族编码序列(PeMYBs)125条,包含1R-MYB结构域的PeMYBs蛋白序列27条,R2R3-MYB结构域96条,R1R2R3-MYB结构域2条。重点对96条R2R3-MYB结构PeMYBs蛋白序列特点进行生物信息学分析。 结果 依据拟南芥的分类标准将小兰屿蝴蝶兰R2R3-MYB类转录因子划分为20个亚群,预测获得同源性较高的直系和旁系同源基因;各基因在四种器官(花、叶、根、茎)中的表达情况各异,39个PeMYBs基因在4种器官中均表达,48个基因在不同器官中有特异性不表达现象,一些基因呈现器官特异性表达特点,推测其可能参与相应组织特定发育时期的调控。 结论 预测获得125条PeMYBs蛋白序列,并对部分PeMYB转录因子可能的调控功能进行了预测,将为细致研究蝴蝶兰MYB转录因子调控植物生长发育和逆境胁迫响应的分子机理提供一定的数据基础。 -
关键词:
- 小兰屿蝴蝶兰
- / R2R3-MYB转录因子家族
- / 全基因组分析
Abstract:Objective To study the sequence characteristics of Phalaenopsis equestris. Method BLASTP was used to search the MYB transcription factors of P. equestris (PeMYBs) in the database of P. equestris whole genome, and then the conserved MYB domain and structural characteristics of PeMYBs were predicted. The sequence alignment, phylogenetic analysis and expression profiles of PeMYBs were conducted by using ClustalW, MEGA and HemI 1.0 softwares. Result 96 PeMYBs have conserved R2R3-MYB domains, 27 have R1-MYB domains, and 2 have R1R2R3-MYB domains. According to the classification criterion, the 96 R2R3-PeMYB members could be divided into 20 groups, and among them, some PeMYB genes were orthologs and PeMYB/AtMYB paralogs. They had different profiles in four organs, among them 39 PeMYBs were expressed in all the four organs, and 48 PeMYBs were specific expression or no expression in different organs. Conclusion The results of this study could provide references for further functional analysis of MYB genes in P. equestris. -
Key words:
- Phalaenopsis equestris
- / R2R3-MYB gene family
- / genome-wide analysis
-
表 1 植物MYB转录因子亚家族成员统计
Table 1. MYB transcription factors in six plant species
Species物种 MYB protein classes MYB蛋白亚家族 1R-MYB R2R3-MYB 3R-MYB 4R-MYB Eudicot Arabidopsis thaliana[2] 64 126 5 1 Populus trichocarpa[3-4] n.d 202 5 - Vitis vinifera[5] n.d 108 5 1 Monocot Oryza sativa[4] 70 125 5 1 Phalaenopsis equestris 27 96 2 - Moss Physcomitrella patens# 31 61 2 1 注:n.d未检测到(not determined)。
#:the Moss Transcription Factor Database (http://planttfdb.cbi.pku.edu.cn).表 2 小兰屿蝴蝶兰R2R3-MYB类转录因子信息统计
Table 2. R2R3-MYB sequence characteristics of Phalaenopsis equestris
基因名称
Gene name基因ID
Gene ID长度
Length/aa内含子数量
IntronsPeMYB01 KF769466 269 - PeMYB02 PEQU_10971 303 2 PeMYB03 PEQU_30611 294 2 PeMYB04 PEQU_23598 211 1 PeMYB05 KF769470 169 - PeMYB06 PEQU_22029 262 1 PeMYB07* PEQU_03393 249 1 PeMYB08 PEQU_10762 219 2 PeMYB09 PEQU_20333 409 2 PeMYB10 PEQU_05119 353 2 PeMYB11 PEQU_09551 238 2 PeMYB12 PEQU_19346 245 3 PeMYB13 PEQU_14300 220 2 PeMYB14 KF769479 220 - PeMYB16 PEQU_09064 377 3 PeMYB17 PEQU_00368 324 2 PeMYB18 PEQU_00728 234 2 PeMYB19 PEQU_02109 198 1 PeMYB20 PEQU_02591 303 2 PeMYB21 PEQU_03213 267 2 PeMYB22 PEQU_04692 322 2 PeMYB23 PEQU_04858 288 2 PeMYB24 PEQU_05034 325 1 PeMYB25 PEQU_05643 295 2 PeMYB26 PEQU_05683 292 2 PeMYB27 PEQU_05903 288 2 PeMYB28 PEQU_05918 312 1 PeMYB29* PEQU_06231 344 1 PeMYB30 PEQU_06488 201 2 PeMYB 31 PEQU_06600 286 2 PeMYB32 PEQU_06922 241 3 PeMYB33 PEQU_07074 313 2 PeMYB34 PEQU_07150 303 2 PeMYB35 PEQU_07382 338 2 PeMYB36 PEQU_07503 326 1 PeMYB37 PEQU_07637 347 2 PeMYB38 PEQU_08801 287 2 PeMYB39 PEQU_09074 229 1 PeMYB40* PEQU_09152 172 1 PeMYB41 PEQU_09363 304 2 PeMYB42 PEQU_09491 563 2 PeMYB43* PEQU_09503 289 11 PeMYB44 PEQU_10362 247 2 PeMYB45* PEQU_10550 287 0 PeMYB46* PEQU_10664 251 2 PeMYB47 PEQU_10683 225 2 PeMYB48* PEQU_10866 237 1 PeMYB49 PEQU_11292 315 3 PeMYB50 PEQU_12592* 271 0 PeMYB51 PEQU_12801* 204 2 PeMYB52 PEQU_13544* 239 2 PeMYB53 PEQU_14547 294 1 PeMYB54 PEQU_14880 245 2 PeMYB55 PEQU_14919 221 2 PeMYB56 PEQU_15257 233 0 PeMYB57 PEQU_15899 180 2 PeMYB58 PEQU_16171 258 1 PeMYB59 PEQU_16389* 231 2 PeMYB60 PEQU_17021* 207 1 PeMYB61 PEQU_17178* 227 1 PeMYB62 PEQU_17258* 239 2 PeMYB63 PEQU_18431 233 2 PeMYB64 PEQU_18783 232 2 PeMYB65 PEQU_18879 358 2 PeMYB66 PEQU_19266 345 2 PeMYB67 PEQU_20514 320 2 PeMYB68 PEQU_21248* 309 2 PeMYB69 PEQU_21694 320 1 PeMYB70 PEQU_23041* 333 1 PeMYB71 PEQU_23940* 312 1 PeMYB72 PEQU_24421 244 2 PeMYB73 PEQU_25920 260 1 PeMYB74 PEQU_29058 310 2 PeMYB75 PEQU_29143 244 1 PeMYB76 PEQU_29475 250 2 PeMYB77 PEQU_30835 302 2 PeMYB78 PEQU_30836 307 2 PeMYB79 PEQU_30837 313 2 PeMYB80 PEQU_30955 322 2 PeMYB81 PEQU_31568 340 2 PeMYB82 PEQU_32696 380 1 PeMYB83 PEQU_32963 250 2 PeMYB84 PEQU_33555* 188 2 PeMYB85 PEQU_33827 129 2 PeMYB86 PEQU_33908* 310 2 PeMYB87 PEQU_34202 340 2 PeMYB88 PEQU_34697 255 2 PeMYB89 PEQU_36816 290 2 PeMYB90 PEQU_36904 276 1 PeMYB91 PEQU_37625 278 1 PeMYB92 PEQU_39789 270 2 PeMYB93 PEQU_40332 260 1 PeMYB94 PEQU_40608 209 1 PeMYB95 PEQU_40802 214 1 PeMYB96 PEQU_41555 239 2 PeMYB97 PEQU_42161 260 2 注:*氨基酸序列中包含保守的EAR-motif. * There is conserved EAR-motif in the amino acid sequence. -
[1] 吴春霞.植物MYB基因研究进展[J].安徽农业科学, 2009, 20(37):9372-9374. [2] Stracke R, Werber M, Weisshaar B. The R2R3-MYB gene family in Arabidopsis thaliana[J]. Current Opinion in Plant Biology, 2001, 4(5):447-456. doi: 10.1016/S1369-5266(00)00199-0 [3] Wilkins O, Nahal H, Foong J, et al. Expansion and Diversification of the Populus R2R3-MYB Family of Transcription Factors[J]. Plant Physiol, 2009, 149(2):981-993. [4] Zhao K, Bartley L E. Comparative genomic analysis of the R2R3-MYB secondary cell wall regulators of Arabidopsis, poplar, rice, maize, and switchgrass[J]. BMC Plant Biology, 2014, 14(1):135. doi: 10.1186/1471-2229-14-135 [5] Matus J T S, Loyola R, Vega A, et al. Post-veraison sunlight exposure induces MYB-mediated transcriptional regulation of anthocyanin and flavonol synthesis in berry skins of Vitis vinifera[J]. Journal of Experimental Botany, 2009, 60(3):853-867 doi: 10.1093/jxb/ern336 [6] Cao Z H, Zhang S Z, Wang R K, et al. Genome wide analysis of the apple MYB transcription factor family allows the identification of MdoMYB121 gene confering abiotic stress tolerance in plants[J]. PloS One, 2013, 8(7):e69955. doi: 10.1371/journal.pone.0069955 [7] He Q, Jones D C, Li W, et al. Genome-Wide Identification of R2R3-MYB Genes and Expression Analyses During Abiotic Stress in Gossypium raimondii[J]. Scientifc Reports, 2016, 6:1-14. doi: 10.1038/s41598-016-0001-8 [8] Li Z, Peng R, Tian Y, et al. Genome-Wide Identification and Analysis of the MYB Transcription Factor Superfamily in Solanum lycopersicum[J]. Plant Cell Physiol, 2016, 57(8):1657-1677. doi: 10.1093/pcp/pcw091 [9] Peng X, Liu H, Wang D, et al. Genome-wide identification of the Jatropha curcas MYB family and functional analysis of the abiotic stress responsive gene JcMYB2[J]. BMC Genomics, 2016, 17(1):251. doi: 10.1186/s12864-016-2576-7 [10] Hsu C, Chen Y, Tsai W, et al. Three R2R3-MYB transcription factors regulate distinct floral pigmentation patterning in Phalaenopsis spp.[J]. Plant Physiol, 2015, 168(1):175-191. doi: 10.1104/pp.114.254599 [11] Abe, H, Urao T, Ito T, et al. Arabidopsis AtMYC2(bHLH) and AtMYB2(MYB) function as transcriptional activators in abscisic acid signaling[J]. Plant Cell, 2003, 15(1):63-78. doi: 10.1105/tpc.006130 [12] Devaiah, B N, Madhuvanthi, R, Karthikeyan, A S, et al. Phosphate starvation responses and gibberellic acid biosynthesis are regulated by the MYB62 transcription factor in Arabidopsis[J]. Mol. Plant, 2009, 2(1):43-58. doi: 10.1093/mp/ssn081 [13] Piljoon S, Xiang F N, Qiao M, et al. The MYB96 transcription factor mediates abscisic acid signaling during drought stress response in Arabidopsis[J]. Plant Physiol, 2009, 151(1):275-289. doi: 10.1104/pp.109.144220 [14] Denekamp M, Smeekens S C. Integration of wounding and osmotic stress signals determines the expression of the AtMYB102 transcription factor gene[J]. Plant Physiol, 2003, 132(3):1415-1423. doi: 10.1104/pp.102.019273 [15] Lippold F, Sanchez D H, Musialak M, et al. AtMyb41 regulates transcriptional and metabolic responses to osmotic stress in Arabidopsis[J]. Plant Physiol, 2009, 149:1761-1772. doi: 10.1104/pp.108.134874 [16] Dai, X., Xu Y, Ma Q, et al. Overexpression of an R1R2R3 MYB gene, OsMYB3R-2, increases tolerance to freezing, drought, and salt stress in transgenic Arabidopsis[J]. Plant Physiol, 2007, 143(4):1739-1751. doi: 10.1104/pp.106.094532 [17] Yang A, Dai X, Zhang W H. A R2R3-type MYB gene, OsMYB2, is involved in salt, cold, and dehydration tolerance in rice[J]. J Exp Bot, 2012, 63(7):2541-2556. doi: 10.1093/jxb/err431 [18] Liao, Y, Zou H F, Wang H W, et al. Soybean GmMYB76, GmMYB92, and GmMYB177 genes confer stress tolerance in transgenic Arabidopsis plants[J]. Cell Res, 2008, 18(10):1047-1060. doi: 10.1038/cr.2008.280 [19] Wang Z, Tang J, Hu R, et al. Genome-wide analysis of the R2R3-MYB transcription factor genes in Chinese cabbage (Brassica rapa ssp. pekinensis) reveals their stress and hormone responsive patterns[J]. BMC Genomics, 2015, 16(1):1-21. doi: 10.1186/1471-2164-16-1 [20] Stracke, R, Ishihara H, Huep G, et al. Differential regulation of closely related R2R3-MYB transcription factors controls flavonol accumulation in different parts of the Arabidopsis thaliana seedling[J]. Plant J, 2007, 50(4):660-677. doi: 10.1111/j.1365-313X.2007.03078.x [21] Lepiniec L, Debeaujon I, Routaboul JM, et al. Genetics and biochemistry of seed flavonoids[J]. Annu Rev Plant Biol, 2006, 57(1):405-430. doi: 10.1146/annurev.arplant.57.032905.105252 [22] Song SS, Qi TC, Huang H, et al. The Jasmonate-ZIM domain proteins interact with the R2R3-MYB transcription factors MYB21 and MYB24 to affect Jasmonate-regulated stamen development in Arabidopsis[J]. Plant Cell, 2011, 23(3):1000-1013. doi: 10.1105/tpc.111.083089 [23] 牛义岭, 姜秀明, 许向阳.植物转录因子MYB基因家族的研究进展[J].分子植物育种, 2016, 14(8):2050-2059. [24] Ma H, Pooler M. Anthocyanin regulatory:structural gene expression in Phalaenopsis[J]. Journal of the American Society for Horticultural Science, 2009, 134(1):88-96. doi: 10.21273/JASHS.134.1.88 [25] 孙叶, 包建忠, 刘春贵, 等.兰花花色基因工程研究进展[J].核农学报, 2015, 29(9):1701-1710. [26] 许传俊, 黄瑶梅, 雯黄, 等.不同花色品种蝴蝶兰花色素苷含量分析及相关基因表达研究[J].华南师范大学学报:自然科学版, 2015, 47(3):93-99. [27] Wu X, Lim S, Yang W. Characterization, expression and phylogenetic study of R2R3-MYB genes in orchid[J]. Plant Molecular Biology, 2003, 51(6):959-972. doi: 10.1023/A:1023050110077 [28] Cai J, Liu X, Vanneste K, et al. The genome sequence of the orchid Phalaenopsis equestris[J]. Nature Genetics, 2015, 47(1):65-72. doi: 10.1038/ng.3149 [29] Chen Y, Yang X, He K, et al. The MYB Transcription Factor Superfamily of Arabidopsis: Expression Analysis and Phylogenetic Comparison with the Rice MYB Family[J]. Plant Molecular Biology, 2006, 60(1):107-124. doi: 10.1007/s11103-005-2910-y [30] 王希庆, 陈柏君, 印莉萍.植物中的MYB转录因子[J].生物技术通报, 2003(2):22-25. doi: 10.3969/j.issn.1002-5464.2003.02.006 [31] Yang C, Xu Z, Song J, et al. Arabidopsis MYB26/MALE STERILE35 regulates secondary thickening in the endothecium and is essential for anther dehiscence[J]. The Plant Cell, 2007, 19(2):534-548. doi: 10.1105/tpc.106.046391 [32] Griesbacb R J, Klein T M. In situ genetic complementation of a flower color mutant in Doritis pulcherrima (Orchidaceae)[J]. Lindleyana, 1993, 8:223-226. [33] Chiou C, Yeh K. Differential expression of MYB gene (OgMYB1) determines color patterning in floral tissue of Oncidium Gower Ramsey[J]. Plant molecular Biology, 2008, 66(4):379-388. doi: 10.1007/s11103-007-9275-3 [34] Kazan K. Negative regulation of defence and stress genes by EAR-motif-containing repressors[J]. Trends Plant Sci, 2006, 11(3):109-112. doi: 10.1016/j.tplants.2006.01.004 [35] 张健飞, 权瑞党, 黄荣峰. EAR转录抑制子结构及功能的研究[J].中国农业科技导报, 2011, 13(4):53-57. doi: 10.3969/j.issn.1008-0864.2011.04.08 [36] Preston J, Wheeler J, Heazlewood J, et al. AtMYB32 is required for normal pollen development in Arabidopsis thaliana[J]. The Plant Journal, 2004, 40(6):979-995. doi: 10.1111/tpj.2004.40.issue-6 [37] Huang C K, Lo P C, Huang L F, et al. A single-repeat MYB transcription repressor, MYBH, participates in regulation of leaf senescence in Arabidopsis[J], Plant Mol Biol, 2015, 88(3):269-286. doi: 10.1007/s11103-015-0321-2 [38] Jin H, Cominelli E, Bailey P, et al. Transcriptional repression by AtMYB4 controls production of UV-protecting sunscreens in Arabidopsis[J]. The EMBO Journal, 2000, 19(22):6150-6161. doi: 10.1093/emboj/19.22.6150 [39] Romero I, Fuertes A, Benito M J, et al. More than 80 R2R3-MYB regulatory genes in the genome of Arabidopsis thaliana[J]. The Plant Journal, 1998, 14(3):273-284. doi: 10.1046/j.1365-313X.1998.00113.x -




 下载:
下载: