-
实时荧光定量PCR (real-time fluorescent quantitative PCR, qRT-PCR)技术兴起于20世纪90年代,目前已经被普遍用于分析检测基因转录水平表达。相对于常规半定量RT-PCR而言,qRT-PCR具有实时、特异性强及灵敏度高等优点,已广泛应用于分子生物学、医学和诊断学等诸多研究领域。
qRT-PCR从荧光组分角度上划分常用的主要有2种:DNA结合染料(SYBR Green Ⅰ)和Taqman探针法。SYBR Green Ⅰ法相较于后者具有实验简单、成本低等特点,从而更多地被应用于低通量、单重实验。从实验目的角度qRT-PCR技术还可以分为绝对定量和相对定量2种。绝对定量主要通过建立已知量的标准品的标准曲线,并利用未知待测样品量的CT值推算某个基因的绝对数值(拷贝数)。相对定量则主要比较不同组织之间或处理样品与未处理样品中目的基因的表达差异。相对定量数据计算方法主要有2-ΔΔCT法(Livak法)、2-ΔCT法和Pfaffl法[1-2]。2-ΔΔCT法(Livak法)是检测基因相对表达量数据分析中最为普遍采用的计算方法,但是该方法要求目的基因和内参基因的扩增效率接近100%,否则,倘若两者扩增效率相差较大,就会导致实验结果不能真实反映基因的表达水平,因此,必须对实验体系进行优化或者采用Pfaffl法计算。
红豆杉(Taxus L.)为裸子植物,属于红豆杉科,因可以从该树种中提取抗癌药物紫杉醇从而引起国际上的广泛关注。目前,有关紫杉醇生物合成的分子机理研究已经较为深入,已经发现了合成途径中19步的14个关键酶基因[3],也有多个转录因子基因以及其它相关基因如JAZs、细胞色素P450等被分离克隆[4-8],并且研究者也都利用qRT-PCR的方法研究了这些基因转录水平的表达。然而,目前尚未见到有关这些基因qRT-PCR中相对定量法的最佳反应体系建立及优化的研究。本研究针对前期工作中克隆得到1个TbAP2基因,构建适用于qRT-PCR实验的最佳反应体系,为更加准确获取TbAP2基因在转录水平不同时空条件下的表达研究提供行之有效的方法。
-
本研究中采用曼地亚红豆杉(Taxus × media)细胞系Tm3为实验材料,采用6, 7-V固体培养基25℃暗培养,每隔2530 d转接1次。
-
将100 mg红豆杉细胞进行液氮研磨,按照柱式植物RNAOUT2.0试剂盒(北京天恩泽基因科技有限公司)说明书操作,提取的总RNA经1%琼脂糖凝胶电泳和NanoDrop 8000分光光度计(Thermo,美国)检测其浓度和质量。取0.6 μg总RNA用于反转录合成cDNA,反转录体系以及操作步骤参照购自天根生化科技(北京)有限公司的Fast Quant RT Kit (with gDNase)说明书进行。
-
根据课题组前期克隆获得的TbAP2基因序列[4],利用Primer 6.0分别设计了3对引物,并根据上下游引物在基因上的位置,将不同上下游引物进行两两组合出多对引物,TbAP2基因引物组合结果为:F1R1、F1R3、F2R1、F2R2、F2R3、F3R1、F3R3。具体信息见表 1。选择TBC41基因为内参基因[9],并由生工生物工程(上海)股份有限公司合成。
表 1 PCR引物序列
Table 1. The information of PCR primers
引物名称Primer name 上游引物5’-3’ Forward primer 下游引物5’-3’ Reverse primer TBC41 F:CAAGAAGAAAGAGTCAGCAAA R:GGAACGACATGACATTATGAATAG TbAP2-1 F1:CTGCAATTGCTCTGGACAAA R1:TCAACAGCTCCTCCTCCTGT TbAP2-2 F2:CAAGGGTGTGAGAATGAGGAG R2:GCAGCTTCTGCAGTTGAATATG TbAP2-3 F3:TCCTCTGCTGCTCTGAAT R3:CTCCTCCTCCTGTAACCAT 将上述7对引物组合首先进行普通PCR扩增,采用25 μL反应体系:12.5 μL 2×Taq PCR Master Mix,1 μL引物(浓度为5 μmol·L-1),1 μL cDNA模板(原液稀释5倍),加水补足至25 μL。PCR反应循环为:94℃预变性3 min;94℃变性30 s,55℃退火30 s,72℃延伸1 min,共进行30个循环;72℃延伸5 min。以水为模板作为阴性对照。反应结束后取5 μL PCR扩增产物,通过2%的琼脂糖凝胶电泳检测引物的特异性和目的片段的扩增长度,初步筛选出较好的引物。
-
荧光定量PCR实验采用正交试验设计,选择三因素三水平试验,故选择L9(34)的正交表来安排实验(表 2),其中,A为PCR体系(5、10、20 μL),B为cDNA模板用量(0.5、1.0、1.5 μL),C为引物用量(0.3、0.5、1.0 μL),每个试验方案至少重复3次。
-
用常规PCR初步筛选出的较好的引物,以反转录合成的cDNA为模板,荧光定量PCR试剂采用KAPA SYBR® FAST qPCR Kit Master Mix(2×)Universal(北京普凯瑞生物科技有限公司),按照正交试验列出的方案进行试验,荧光定量PCR反应的程序为:95℃预变性3 min;95℃变性10 s,55℃退火20 s,72℃延伸30 s,45次循环,每次循环第3步采集荧光;最后95℃变性5 s,退火至60℃后保温1 min。实验中分别以灭菌ddH2O和去除DNA污染未反转录的RNA为模板作为NTC和NRC阴性对照。
表 2 L9(34)正交试验设计
Table 2. L9(34) orthogonal experimental design
实验号
No.因素Factors 方案
ProgramA (PCR体系
/μL)
PCR reactionB (cDNA模板
/μL)
cDNA templateC (引物用量
/μL)
Primer volume1 1(5) 1(0.5) 1(0.3) A1B1C1 2 1 2(1.0) 2(0.5) A1B2C2 3 1 3(1.5) 3(1.0) A1B3C3 4 2(10) 1 2 A2B1C2 5 2 2 3 A2B2C3 6 2 3 1 A2B3C1 7 3(20) 1 3 A3B1C3 8 3 2 1 A3B2C1 9 3 3 2 A3B3C2 注:cDNA模板浓度为0.6 μg总RNA反转录合成的cDNA原液稀释5倍; 引物浓度为5 μmol·L-1
Note: With 0.6 μg total RNA the first strand cDNA was synthesized, and then diluted 5 times as PCR template; the primer concentration was 5 μmol·L-1 -
先将反转录成的cDNA稀释5倍,以其为原始液,按3倍梯度稀释成5个浓度梯度的标准品,进行荧光定量PCR扩增,利用Excel绘制标准曲线,以CT值为纵坐标,以稀释倍数的对数值为横坐标,并标注线性方程和R2,计算扩增效率。
-
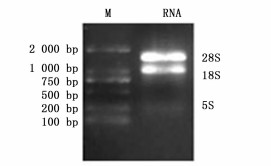
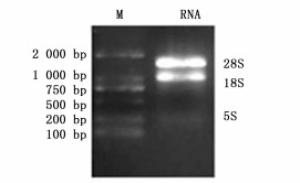
提取高质量总RNA是做好荧光定量PCR的基础,本研究中提取的总RNA经1%的琼脂糖凝胶电泳检测,28S和18S 2个条带清晰明亮,并无拖尾现象(图 1),表明提取的RNA完整程度高,无降解,无DNA和蛋白质污染,用于反转成cDNA。
-
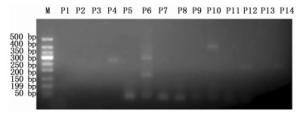
根据课题组前期研究所克隆所得到的TbAP2基因序列设计特异性引物,将所有引物组合经常规PCR扩增。图 2显示:TbAP2基因的7对引物组合中,TbF1R1、TbF1R3、TbF3R1和TbF3R3引物组合表现较好(P14、P12、P4、P2),均扩出单一条带且无二聚体产生,阴性对照也没有扩增出二聚体等杂带,且TbF3R1引物组合(P4)目的条带亮度最高,引物TbF3R3扩增的目的条带亮度最低(P2)。其它引物组合中,TbF2R1引物组合扩出1条目的条带(P10),但PCR产物以及阴性对照均有二聚体产生(P10、P9);TbF2R2引物组合未扩增出目的条带(P8),且阴性对照有二聚体产生(P7);TbF2R3引物组合扩出3条条带(P6),且阴性对照有二聚体产生(P5)。
此外,将以上7对引物中表现较好的引物TbF1R1、TbF1R3、TbF3R1和表现较差的TbF2R1引物以及TBC41基因引物在Roche 480仪器上进行引物特异性验证和重复性检测,结果发现:引物TBC41、TbF1R1、TbF1R3和TbF3R1特异性较好(图 3A、B、C、E),都只有1个明显单峰,而TbF2R1引物特异性确实较差(图 3D),峰形不锐利,且存在1个不明显的肩峰,表明确实存在非特异性扩增条带。
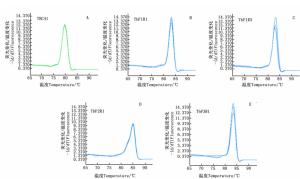
图 3 TBC41 (A)、TbF1R1 (B)、TbF1R3(C)、TbF2R1(D)和TbF3R1(E)引物熔解曲线图
Figure 3. Melting curve of primer pairs of TBC41 (A), TbF1R1 (B), TbF1R3(C), TbF2R1 (D) and TbF3R1(E)
从引物扩增重复性角度分析,发现以上5对引物的重复性均表现良好,CT值大小都在2030之间,且TbF3R1的CT值平均值最小(21.72),而且每个基因均实行3复孔平行实验, 发现CT值标准差最大为0.28 (表 3)。综合考虑,本研究选择引物TbF3R1和TBC41基因引物进行后续荧光定量PCR实验。
表 3 引物重复性检测
Table 3. The detection of the repeatability of primers
引物组合
Primer pairs重复1
Repeat1重复2
Repeat2重复3
Repeat3CT平均值
CT Mean Value标准差
Std. DevTBC41 22.17 22.52 22.73 22.47 0.28 TbF1R1 26.57 26.94 26.80 26.77 0.19 TbF1R3 24.64 24.40 24.55 24.53 0.12 TbF2R1 22.36 22.61 22.54 22.50 0.13 TbF3R1 21.81 21.53 21.83 21.72 0.17 -
将引物TBC41和TbF3R1按照正交试验方案同时进行荧光定量PCR并设置阴性对照。表 4表明:最优方案的选择与K(k)值有关,在本实验中,CT值越小,引物的扩增效率越高,因此,选择K1(k1)、K2(k2)、K3(k3)最小的水平,即TBC41扩增效率最高的方案为A1B3C3。同理,可以看出TbF3R1引物组合扩增效率最高的方案也为A1B3C3(表 5)。此外,极差越大,表明因素越重要,可以看出,本研究中3个因素影响力大小为:PCR反应体系>引物量>模板用量。引物TBC41和TbF3R1在常用的10 μL体系下最佳方案为A2B2C3,20 μL体系下最佳的组合方案分别为A3B1C3和A3B3C2。由于TbF3R1的方案A3B3C2的CT值与方案A3B1C3相近,为使在同一方案中同时使2对引物具有相近的扩增效率,因此,后续确定扩增体系扩增效率的实验中,20 μL体系采用方案A3B1C3。
表 4 引物TBC41正交试验方案结果
Table 4. The scheme of orthogonal experimental of primer TBC41 with qRT-PCR method
序号
No.因素Factors 方案
ProgramCT平
均值
Mean CTA (PCR体
系/μL)
PCR reactionB (cDNA模
板/μL)
cDNA templateC (引物用
量/μL)
Primer volume1 1 1 1 A1B1C1 26.34 2 1 2 2 A1B2C2 23.89 3 1 3 3 A1B3C3 22.42 4 2 1 2 A2B1C2 28.33 5 2 2 3 A2B2C3 25.36 6 2 3 1 A2B3C1 27.72 7 3 1 3 A3B1C3 29.10 8 3 2 1 A3B2C1 33.48 9 3 3 2 A3B3C2 29.93 K1 72.65 83.77 87.54 K2 81.41 82.73 82.15 K3 92.51 80.07 76.88 k1 24.22 27.92 29.18 k2 27.14 27.58 27.38 k3 30.84 26.69 25.63 R 6.62 1.23 3.55 注:Ki:水平效应,即任一列上水平号为i所对应的实验结果之和;ki:平均效应,即水平和除以水平数;R:极差,即在任一列上最大K(k)值与最小K(k)值之差。cDNA模板浓度为0.6 μg总RNA反转录合成的cDNA原液稀释5倍; 引物浓度为5 μmol·L-1。下同
Note: Ki: horizontal effect, that is, the sum of the experimental results corresponding to level number i in any column; ki: the average effect, that is, the sum of the horizontal divided by the quantity of the horizontal; R: Range, the different between the maximum value K (k) and the minimum K (k) value. With 0.6 μg total RNA the first strand cDNA was synthesized, and then diluted 5 times as PCR template; the primer concentration was 5 μmol·L-1.The same below.表 5 TbF3R1正交试验方案结果
Table 5. The scheme of orthogonal experimental of primer TbF3R1 with qRT-PCR method
序号
No.因素Factors 方案
ProgramCT平
均值
Mean CTA (PCR体
系/μL)
PCR reactionB (cDNA模
板/μL)
cDNA templateC (引物用
量/μL)
Primer volume1 1 1 1 A1B1C1 21.59 2 1 2 2 A1B2C2 20.41 3 1 3 3 A1B3C3 19.37 4 2 1 2 A2B1C2 22.58 5 2 2 3 A2B2C3 20.72 6 2 3 1 A2B3C1 21.48 7 3 1 3 A3B1C3 23.33 8 3 2 1 A3B2C1 23.91 9 3 3 2 A3B3C2 22.43 K1 61.37 67.50 66.98 K2 64.78 65.04 65.42 K3 69.67 63.28 63.42 k1 20.46 22.50 22.33 k2 21.59 21.68 21.81 k3 23.22 21.09 21.14 R 2.77 1.41 1.19 将模板cDNA依次稀释3倍,绘制5、10、20 μL体系下TBC41和引物TbF3R1在A1B3C3、A2B2C3和A3B1C3方案下的标准曲线,计算扩增效率。图 4表明:尽管此3种方案下,6条标准曲线的回归系数R2都符合>0.980的条件,但是TBC41基因的扩增效率却只有70%80%,引物TbF3R1的扩增效率在A1B3C3和A2B2C3方案中分别为98%和91%,而在A3B1C3方案中仅为74%(表 6)。可以看出,3个方案中没有任何一个方案达到内参基因和目的基因扩增效率为90%105%的标准,因此,需要对此3种方案进行继续优化。
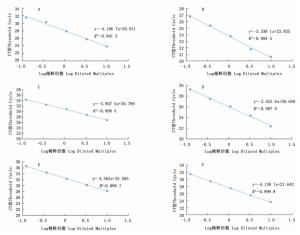
图 4 TBC41和TbF3R1引物在A1B3C3、A2B2C3和A3B1C3中的标准曲线
Figure 4. The standard curves of the primer pairs of TBC41 and TbF3R1in A1B3C3、A2B2C3和A3B1C3 programs before optimization
表 6 优化前A1B3C3、A2B2C3和A3B1C3方案中TBC41和TbF3R1的扩增效率
Table 6. The amplification efficiency of TBC41 and TbF3R1 primer pairs under A1B3C3, A2B2C3 and A3B1C3 programs before optimization
反应体系
Reaction system/μL方案
Program引物组合
Primer扩增效率
PCR efficiency/%回归系数
R2斜率
Slope5 A1B3C3 TBC41 73 0.991 2 -4.196 7 TbF3R1 98 0.994 5 -3.338 1 10 A2B2C3 TBC41 80 0.998 5 -3.907 5 TbF3R1 91 0.997 0 -3.553 6 20 A3B1C3 TBC41 70 0.999 7 -4.363 0 TbF3R1 74 0.999 8 -4.138 7 -
一般来说,实验中扩增效率较难准确达到100%,因此,在实际操作时,保证扩增效率为90%105%,线性回归系数R2>0.980,即可视为满足2-ΔΔCT法计算要求。本实验通过改变体系中模板和引物的体积(表 7),以达到内参基因和目的基因的扩增效率都在100%左右的标准。表 8表明:经过优化后,5、10、20 μL体系下,内参基因TBC41和目的基因TbAP2的扩增效率均可以保持在93%99%之间,且R2都在0.980以上(图 5)。
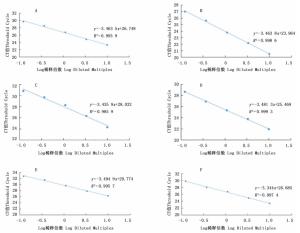
图 5 TBC41和TbF3R1引物在优化后5、10、20 μL体系下的标准曲线
Figure 5. The standard curves of the primer pairs of TBC41 and TbF3R1in 5, 10 and 20 μL reaction volumes after optimization
表 7 优化后的荧光定量PCR体系
Table 7. The optimized qRT-PCR composition
组成成分
Composition5 μL体系
5 μL reaction
system/μL10 /μL体系
10 μL reaction
system/μL20 μL体系
20 μL reaction
system/μLMix(2×)Universal 2.5 5 10 cDNA 1.0 1.2 0.5 正、反引物体积 1.5 1.3 1.5 ddH20 0 2.5 8 注:cDNA模板浓度为0.6 μg总RNA反转录合成的cDNA原液稀释5倍;引物浓度为5 μmol·L-1 。
Note: 0.6 μg total RNA is reverse transcribed to cDNA stock solution and then diluted 5 times as cDNA template; the primer concentration was 5 μmol·L-1。表 8 不同PCR反应体系下引物TBC41和TbF3R1的扩增效率
Table 8. The amplification efficiency of primers TBC41 and TbF3R1 in different PCR reactions after optimization
反应体系/μL
reaction system引物组合
Primer扩增效率
PCR efficiency/%回归系数
R2斜率
Slope5 TBC41 94 0.993 8 -3.483 5 TbF3R1 94 0.998 6 -3.463 8 10 TBC41 95 0.985 9 -3.435 9 TbF3R1 94 0.999 3 -3.481 3 20 TBC41 93 0.995 7 -3.494 9 TbF3R1 99 0.997 4 -3.344 0 -
在qRT-PCR的相对定量实验中,引物设计、模板浓度和质量以及退火温度等都是影响实验结果的重要因素[10-12]。SYBR Green Ⅰ是当前qRT-PCR实验最常用的试剂,具有较强的灵敏性,但是却也存在不能专一识别目的基因双链DNA的缺点,实验中经常会出现引物二聚体或非特异性扩增出现的荧光值而影响实验的准确性。因此,设计出高效且特异性扩增的引物至关重要。在本研究中,笔者针对TbAP2基因设计、组合出的7对引物进行普通PCR筛选,TbF1R1、TbF1R3、TbF3R1和TbF3R3引物组合均扩增出单一条带,其他3对引物均有引物二聚体或者杂带。由于TbF3R3引物扩增效率最低,因此利用Roche480针对TbF1R1、TbF1R3和TbF3R1进行特异性和重复性分析。熔解曲线分析显示:3种引物组合只扩增出唯一单峰,未产生引物二聚体等非特异性扩增,表明3种引物组合均具有良好特异性。此外,3种引物组合还具有良好的重复性,技术重复CT值误差均小于1。
鉴于TbF3R1的CT值最小,且标准差较小(SD=0.17),因此,选择该引物组合以及内参基因TBC41进行后续正交实验进行qRT-PCR最佳体系建立。通过表 6可以看出:2个基因未优化正交实验给出的最佳方案在5、10、20 μL体系下扩增效率绝大部分未能达到标准,并且5 μL和10 μL体系下TBC41和TbF3R1的扩增效率相差19%和25%。可见,建立和优化qRT-PCR反应体系十分有必要;而优化后的3种qRT-PCR体系引物TBC41和TbF3R1的扩增效率在93%99%之间,因而更适合用于2-ΔΔCT法计算。
此外,从本研究结果还可以看出qRT-PCR反应体系对于扩增效率的影响力最高,其次是引物用量和模板用量(表 4、5)。目前,国内实验室大多采用10~25 μL体系,而市场上商业公司大多采用384孔5 μL反应体系。减少qRT-PCR反应体系,最大好处是能够节约经费降低成本,还有利于检测低拷贝基因的表达,但也存在降低稳定性的风险。已有研究者针对转录水平低表达基因,在甘蓝(Brassica oleracea)和叶用莴苣(Lactuca sativa)中建立了小体积(10 μL)的qRT-PCR反应体系[13-14]。本研究中建立的5 μL和10 μL小体积反应体系中,目的基因和内参基因的扩增效率均在94%左右,同样为检测TbAP2以及其他红豆杉中低拷贝基因的表达提供了有益参考。此外,一般认为,最佳的引物浓度应该在0.1~0.5 μmol·L-1,较高的引物浓度会导致非特异性产物扩增;而高质量的cDNA模板也是影响qRT-PCR定量结果的重要因素。因此,必须保证提取的RNA完整且无基因组DNA污染[11]。
-
为了更准确地检测红豆杉TbAP2基因的表达量,本研究对内参基因TBC41和目的基因TbAP2的最佳荧光定量PCR体系进行了建立和优化,成功建立了红豆杉TbAP2基因和内参基因TBC41在5 μL小体系、10 μL和20 μL体积下的最佳qRT-PCR反应体系,且3个体系扩增效率均在90%105%之间,回归系数R2>0.980。本研究建立的qRT-PCR反应体系,为今后检测TbAP2基因以及红豆杉其他相关基因在转录水平不同时空条件下的表达提供了有效的方法和理论指导。
红豆杉TbAP2基因荧光定量PCR体系的建立及优化
Optimization of Fluorescent Quantitative Real-time PCR System of Taxus chinensis var. mairei
-
摘要:
目的 建立稳定可靠的、适合检测红豆杉(Taxus L.)TbAP2基因表达量的荧光定量PCR实验体系。对于检测该物种中基因的组织特异性表达具有重要意义。 方法 以曼地亚红豆杉细胞为试材,提取总RNA并反转录为cDNA,根据TbAP2基因序列设计多对引物,合成内参基因TBC41的引物,采用正交试验L9(34)方法分别筛选以上2个基因5 μL和10 μL小反应体系及20 μL常用体系中的最佳组合,并通过cDNA模板用量和引物用量等方面进行优化,以确保基因扩增效率在90%~105%之间。 结果 本研究建立了TBC41和TbAP2基因在5、10、20 μL体系下的荧光定量最佳PCR反应体系,在优化后的5 μL体系下,加入Mix(2×)Universal 2. 5 μL,cDNA模板1.0 μL,正反引物共1.5 μL,内参基因TBC41和目的基因TbAP2的扩增效率均为94%;在优化后的10 μL下,加入Mix(2×)Universal 5 μL,cDNA模板1.2 μL,正反引物共1.3 μL,TBC41和TbAP2的扩增效率分别为95%和94%。在优化后的20 μL下,加入Mix(2×)Universal 10 μL,cDNA模板0.5 μL,正反引物共1.5 μL,TBC41和TbAP2的扩增效率分别为93%和99%,以上各扩增体系回归系数R2均大于0.980。 结论 在以上3种反应体系下,内参基因和目的基因均具有接近100%的扩增效率,表明本研究成功建立了适合检测红豆杉TbAP2基因表达量的荧光定量PCR实验体系,并为红豆杉其它基因的表达研究提供参考。 Abstract:Objective To establish a stable and suitable real-time fluorescent quantitative PCR (qRT-PCR) experiment system of TbAP2 in Taxus L. Method Total RNA was extracted from the cell line of Taxus×media and used to reverse transcript cDNA. According to the sequence of TbAP2 gene obtained previously, 7 primer pairs were designed and synthesized with the TBC41 gene as the housekeeping gene. The orthogonal test L9 (34) method were used to choose the stable and suitable qRT-PCR experiment system with the cDNA as template. The volume of qRT-PCR reaction included 5, 10 and 20μL. The amplification efficiency would be assured between 90%-105% by adjusting the dosage of cDNA template and primer pairs, respectively. Result This study established the optimal qRT-PCR reaction system of TBC41 TbAP2 and gene in 5μL, 10μL and 20μL volume. In the optimized 5μL system, the amplification efficiency of both TBC41 and TbAP2 were 94%. In the optimized 10μL system, the amplification efficiency of TBC41 and TbAP2 were 95% and 94%, respectively. In the optimized 20μL system, the amplification efficiency of TBC41 and TbAP2 were 93% and 99%, respectively. The regression coefficient R2 in all the three amplification system were greater than 0.980. Conclusion All the reaction systems mentioned above show that the amplification efficiency of TBC41 and TbAP2 close to 100%, indicating that these detection program are suitable to investigate the TbAP2 gene expression by qRT-PCR method. -
Key words:
- Taxus L.
- / AP2
- / orthogonal test
- / qRT-PCR
-
表 1 PCR引物序列
Table 1. The information of PCR primers
引物名称Primer name 上游引物5’-3’ Forward primer 下游引物5’-3’ Reverse primer TBC41 F:CAAGAAGAAAGAGTCAGCAAA R:GGAACGACATGACATTATGAATAG TbAP2-1 F1:CTGCAATTGCTCTGGACAAA R1:TCAACAGCTCCTCCTCCTGT TbAP2-2 F2:CAAGGGTGTGAGAATGAGGAG R2:GCAGCTTCTGCAGTTGAATATG TbAP2-3 F3:TCCTCTGCTGCTCTGAAT R3:CTCCTCCTCCTGTAACCAT 表 2 L9(34)正交试验设计
Table 2. L9(34) orthogonal experimental design
实验号
No.因素Factors 方案
ProgramA (PCR体系
/μL)
PCR reactionB (cDNA模板
/μL)
cDNA templateC (引物用量
/μL)
Primer volume1 1(5) 1(0.5) 1(0.3) A1B1C1 2 1 2(1.0) 2(0.5) A1B2C2 3 1 3(1.5) 3(1.0) A1B3C3 4 2(10) 1 2 A2B1C2 5 2 2 3 A2B2C3 6 2 3 1 A2B3C1 7 3(20) 1 3 A3B1C3 8 3 2 1 A3B2C1 9 3 3 2 A3B3C2 注:cDNA模板浓度为0.6 μg总RNA反转录合成的cDNA原液稀释5倍; 引物浓度为5 μmol·L-1
Note: With 0.6 μg total RNA the first strand cDNA was synthesized, and then diluted 5 times as PCR template; the primer concentration was 5 μmol·L-1表 3 引物重复性检测
Table 3. The detection of the repeatability of primers
引物组合
Primer pairs重复1
Repeat1重复2
Repeat2重复3
Repeat3CT平均值
CT Mean Value标准差
Std. DevTBC41 22.17 22.52 22.73 22.47 0.28 TbF1R1 26.57 26.94 26.80 26.77 0.19 TbF1R3 24.64 24.40 24.55 24.53 0.12 TbF2R1 22.36 22.61 22.54 22.50 0.13 TbF3R1 21.81 21.53 21.83 21.72 0.17 表 4 引物TBC41正交试验方案结果
Table 4. The scheme of orthogonal experimental of primer TBC41 with qRT-PCR method
序号
No.因素Factors 方案
ProgramCT平
均值
Mean CTA (PCR体
系/μL)
PCR reactionB (cDNA模
板/μL)
cDNA templateC (引物用
量/μL)
Primer volume1 1 1 1 A1B1C1 26.34 2 1 2 2 A1B2C2 23.89 3 1 3 3 A1B3C3 22.42 4 2 1 2 A2B1C2 28.33 5 2 2 3 A2B2C3 25.36 6 2 3 1 A2B3C1 27.72 7 3 1 3 A3B1C3 29.10 8 3 2 1 A3B2C1 33.48 9 3 3 2 A3B3C2 29.93 K1 72.65 83.77 87.54 K2 81.41 82.73 82.15 K3 92.51 80.07 76.88 k1 24.22 27.92 29.18 k2 27.14 27.58 27.38 k3 30.84 26.69 25.63 R 6.62 1.23 3.55 注:Ki:水平效应,即任一列上水平号为i所对应的实验结果之和;ki:平均效应,即水平和除以水平数;R:极差,即在任一列上最大K(k)值与最小K(k)值之差。cDNA模板浓度为0.6 μg总RNA反转录合成的cDNA原液稀释5倍; 引物浓度为5 μmol·L-1。下同
Note: Ki: horizontal effect, that is, the sum of the experimental results corresponding to level number i in any column; ki: the average effect, that is, the sum of the horizontal divided by the quantity of the horizontal; R: Range, the different between the maximum value K (k) and the minimum K (k) value. With 0.6 μg total RNA the first strand cDNA was synthesized, and then diluted 5 times as PCR template; the primer concentration was 5 μmol·L-1.The same below.表 5 TbF3R1正交试验方案结果
Table 5. The scheme of orthogonal experimental of primer TbF3R1 with qRT-PCR method
序号
No.因素Factors 方案
ProgramCT平
均值
Mean CTA (PCR体
系/μL)
PCR reactionB (cDNA模
板/μL)
cDNA templateC (引物用
量/μL)
Primer volume1 1 1 1 A1B1C1 21.59 2 1 2 2 A1B2C2 20.41 3 1 3 3 A1B3C3 19.37 4 2 1 2 A2B1C2 22.58 5 2 2 3 A2B2C3 20.72 6 2 3 1 A2B3C1 21.48 7 3 1 3 A3B1C3 23.33 8 3 2 1 A3B2C1 23.91 9 3 3 2 A3B3C2 22.43 K1 61.37 67.50 66.98 K2 64.78 65.04 65.42 K3 69.67 63.28 63.42 k1 20.46 22.50 22.33 k2 21.59 21.68 21.81 k3 23.22 21.09 21.14 R 2.77 1.41 1.19 表 6 优化前A1B3C3、A2B2C3和A3B1C3方案中TBC41和TbF3R1的扩增效率
Table 6. The amplification efficiency of TBC41 and TbF3R1 primer pairs under A1B3C3, A2B2C3 and A3B1C3 programs before optimization
反应体系
Reaction system/μL方案
Program引物组合
Primer扩增效率
PCR efficiency/%回归系数
R2斜率
Slope5 A1B3C3 TBC41 73 0.991 2 -4.196 7 TbF3R1 98 0.994 5 -3.338 1 10 A2B2C3 TBC41 80 0.998 5 -3.907 5 TbF3R1 91 0.997 0 -3.553 6 20 A3B1C3 TBC41 70 0.999 7 -4.363 0 TbF3R1 74 0.999 8 -4.138 7 表 7 优化后的荧光定量PCR体系
Table 7. The optimized qRT-PCR composition
组成成分
Composition5 μL体系
5 μL reaction
system/μL10 /μL体系
10 μL reaction
system/μL20 μL体系
20 μL reaction
system/μLMix(2×)Universal 2.5 5 10 cDNA 1.0 1.2 0.5 正、反引物体积 1.5 1.3 1.5 ddH20 0 2.5 8 注:cDNA模板浓度为0.6 μg总RNA反转录合成的cDNA原液稀释5倍;引物浓度为5 μmol·L-1 。
Note: 0.6 μg total RNA is reverse transcribed to cDNA stock solution and then diluted 5 times as cDNA template; the primer concentration was 5 μmol·L-1。表 8 不同PCR反应体系下引物TBC41和TbF3R1的扩增效率
Table 8. The amplification efficiency of primers TBC41 and TbF3R1 in different PCR reactions after optimization
反应体系/μL
reaction system引物组合
Primer扩增效率
PCR efficiency/%回归系数
R2斜率
Slope5 TBC41 94 0.993 8 -3.483 5 TbF3R1 94 0.998 6 -3.463 8 10 TBC41 95 0.985 9 -3.435 9 TbF3R1 94 0.999 3 -3.481 3 20 TBC41 93 0.995 7 -3.494 9 TbF3R1 99 0.997 4 -3.344 0 -
[1] Livak K, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. doi: 10.1006/meth.2001.1262 [2] 刘正霞, 徐阳, 徐进梅, 等.不同引物及数据分析方法对定量PCR结果的影响[J].南京医科大学学报:自然科学版, 2009, 29(8): 1112-1117. [3] Croteau R, Ketchum R E, Long R M, et al. Taxol biosynthesis and molecular genetics[J]. Phytochem Rev, 2006, 5(1): 75-97. doi: 10.1007/s11101-005-3748-2 [4] 杨艳芳, 刘洪伟, 邱德有.欧洲红豆杉AP2/ERF转录因子基因序列分析[J].生物资源, 2014, 36(4):54-59. [5] 李艳艳, 杨艳芳, 王俊青, 等.红豆杉TcLBDs基因的克隆与表达分析[J].林业科学, 2015, 51(10): 126-133. [6] Nasiri J, Naghavi M R, Alizadeh H, et al. Seasonal-based temporal changes fluctuate expression patterns of TXS, DBAT, BAPT and DBTNBT genes alongside production of associated taxanes in Taxus baccata[J]. Plant Cell Rep, 2016, 35(5):1103-1119. doi: 10.1007/s00299-016-1941-y [7] Liao W F, Zhao S Y, Zhang M, et al. Transcriptome assembly and systematic identification of novel cytochrome P450s in Taxus chinensis[J]. Front Plant Sci, 2017, 8: 1468. doi: 10.3389/fpls.2017.01468 [8] Zhang M, Chen Y, Nie L, et al. Molecular, structural, and phylogenetic analyses of Taxus chinensis JAZs[J]. Gene, 2017, 620: 66-74. doi: 10.1016/j.gene.2017.04.005 [9] Ramirez-Estrada K, Osuna L, Moyano E, et al. Changes in gene transcription and taxane production in elicited cell cultures of Taxus × media and Taxus globosa[J]. Phytochemistry, 2015, 117:174-184. doi: 10.1016/j.phytochem.2015.06.013 [10] Nolan T, Hands R E, Bustin S A. Quantification of mRNA using real-time PCR[J]. Nat Protoc, 2006, 1(3): 1559-1582. doi: 10.1038/nprot.2006.236 [11] Udvardi M K, Czechowski T, Scheible W R. Eleven golden rules of quantitative RT-PCR[J]. Plant Cell, 2008, 20(7): 1736-1737. doi: 10.1105/tpc.108.061143 [12] Baker M. qPCR: quicker and easier but don't be sloppy[J]. Nat Methods, 2011, 8(3): 207-212. doi: 10.1038/nmeth0311-207 [13] 王效维, 刘海霞, 杨颖丽, 等.低表达甘蓝花药发育相关基因小体积荧光定量反应体系的简历和可靠性分析[J].西北农业学报, 2011, 20(4): 111-115. doi: 10.3969/j.issn.1004-1389.2011.04.023 [14] 李婷, 韩莹琰, 范双喜, 等.叶用莴苣荧光定量PCR体系的优化[J].中国农学通报, 2013, 29(13): 106-113. doi: 10.11924/j.issn.1000-6850.2012-3317 -




 下载:
下载: