-
兰科(Orchidaceae)植物为多年生草本种子植物[1-2],约6个亚科800属35 000种[3-4]。兰科植物不仅有很高的观赏价值,还具有很高的药用和经济价值[5-7]。小兰屿蝴蝶兰(Phalaenopsis equestris (Schauer) Rchb.)[8]和铁皮石斛(Dendrobium officinale Kimura et Migo)[9-10]全基因组测序工作的完成,为科学工作者深入研究兰科植物在形态进化、生理适应和共生关系等方面的分子机制提供了海量的信息资源和前所未有的机遇。
Ca2+是植物细胞在发育和应答外界信号中功能最广泛的第二信使。植物经长期进化,在细胞内形成一种独特的蛋白,这种蛋白拥有高度保守性结构(EF-hand motif),可以结合Ca2+,行使生物学功能,即:钙调蛋白(CaMs)和类钙调蛋白(CMLs)[11]。在植物细胞中,CaM和CML两类蛋白与Ca2+结合后形成的复合物参与多种生物学反应[12-13],是目前被研究最多的Ca2+受体蛋白[14],且在结构上非常保守,具有调控植物生长发育基因和生物胁迫条件下应答基因的表达作用[15-16]。目前,对兰科植物中CaM及CML基因家族的研究尚未见报道,本文利用生物信息学的方法预测了小兰屿蝴蝶兰和铁皮石斛中的CaM及CML基因家族成员,同时分析了这些成员的蛋白保守结构域,系统进化关系以及基因表达模式,为兰科植物CaM及CML基因家族功能验证提供数据基础。
HTML
-
通过OrchidBase数据库[8](http://orchidbase.itps.ncku.edu.tw/)下载小兰屿蝴蝶兰和铁皮石斛的全基因蛋白序列文件;通过TAIR基因组序列数据库[17](http://www.arabidopsis.org/)下载拟南芥(Arabidopsis thaliana)CaM和CML蛋白序列。将拟南芥的CaM和CML蛋白序列作为查询序列,利用BLASTP程序对小兰屿蝴蝶兰和铁皮石斛蛋白序列文件进行本地化比对检索,手动去除错配序列后,依据与拟南芥序列的同源性对其进行命名[18];利用DNAMAN软件[19]计算CaM及CML蛋白序列的分子量及等电点等;通过在线分析服务GSDS[20]对小兰屿蝴蝶兰和铁皮石斛CaM及CML基因家族内含子及外显子结构进行分析。
-
将小兰屿蝴蝶兰和铁皮石斛的候选蛋白序列与拟南芥CaM蛋白序列通过软件ClustalX 2.0.10 [17]进行序列比对。利用在线数据库SMART[18]和Pfam[19]对候选蛋白序列的保守结构域进行分析和鉴定。通过在线软件Myristoylator[20](http://web.expasy.org/myristoylator/)确定是否存在潜在豆蔻酰化位点。
-
利用软件MEGA6.05[19]分别构建小兰屿蝴蝶兰和铁皮石斛CaM及CML蛋白家族系统进化树,建树方法为最大似然法(Maximum Likelihood,ML)[19],Bootstrap运行次数设置为1 000,其他参数均为默认。
-
从兰科植物基因组数据库(http://www.nature.com/ng/journal/v47/n1/full/ng.3149.html)下载小兰屿蝴蝶兰不同组织(花、叶、茎、根)[8]以及利用本实验室获得的石斛种子与美胞胶膜菌互作转录组数据[21]。将RPKM(Reads Per Kilo bases per Million reads)值通过HemI(Heatmap Illustrator)1.0.3.3软件进行作图分析[22],分别统计CaM及CML家族基因表达情况。
1.1. 兰科植物CaM及CML家族基因的筛选及确定
1.2. 兰科植物CaM蛋白的多重序列比对及蛋白序列分析
1.3. 兰科植物CaM及CML蛋白系统进化分析
1.4. 兰科植物CaM及CML基因表达分析
-
从小兰屿蝴蝶兰和铁皮石斛基因组中均筛选获得4个CaM(OPeCaM1~4,ODcCaM1~4)和54个CML(OPeCML1~54,ODcCML1~54),其中,CaM蛋白序列全长为148~149 aa,分子量为11.96~17.96 kDa,等电点为3.67~3.95,都具有4个手性分子结构域(EF-hand);CML蛋白序列全长为72~337 aa(除 ODcCML53 为691aa),分子量为6.95~76.94 kDa,等电点为3.52~8.85,分别具有1~5个手性分子结构域;小兰屿蝴蝶兰CaM及CML家族基因中39个基因没有内含子,19个基因具有内含子;铁皮石斛CaM及CML基因家族中有41个基因没有内含子,17个基因具有内含子(表 1、表 2)。小兰屿蝴蝶兰OPeCaM1~4的相似度为91.95%~95.97%,相差的氨基酸个数为6~12个;铁皮石斛ODcCaM1~4的相似度为92.00%~98.66%,相差的氨基酸个数为2~12个(表 3、表 4)。
名称
Name基因ID
Gene ID亚家族
subfamily蛋白序列长度
Length of protein/aaEF hands 分子量
Mol Wt/kDa等电点
pI内含子
Intron甲硫氨酸的含量
percentage methionine/%OPeCaM1 PEQU_18438 1 149 4 16.96 3.89 2 6.02 OPeCaM2 PEQU_10717 1 149 4 16.96 3.89 2 6.01 OPeCaM3 PEQU_12537 1 148 4 16.96 3.88 2 6.00 OPeCaM4 PEQU_26335 1 149 4 17.96 3.95 1 6.05 OPeCML01 PEQU_04925 1 148 4 12.96 3.63 4 4.82 OPeCML02 PEQU_07491 10 155 4 16.96 3.95 1 5.01 OPeCML03 PEQU_06469 10 166 4 16.96 4.02 0 5.90 OPeCML04 PEQU_16519 1 135 3 20.96 3.80 0 4.11 OPeCML05 PEQU_34003 10 199 4 30.98 4.21 6 4.67 OPeCML06 PEQU_29891 1 120 3 14.96 3.52 2 2.83 OPeCML07 PEQU_25947 7 190 4 24.95 4.16 1 6.36 OPeCML08 PEQU_22407 6 155 4 19.97 3.98 0 3.42 OPeCML09 PEQU_40659 5 154 4 18.95 3.95 0 3.71 OPeCML10 PEQU_35611 1 72 2 8.96 3.73 1 2.44 OPeCML11 PEQU_15751 10 141 3 17.96 3.83 4 2.12 OPeCML12 PEQU_06469 10 166 4 16.97 4.19 0 6.23 OPeCML13 PEQU_41809 6 219 4 24.95 4.38 0 5.22 OPeCML14 PEQU_30031 8 137 4 20.96 3.88 0 5.01 OPeCML15 PEQU_40424 2 193 4 23.97 4.07 0 4.93 OPeCML16 PEQU_18134 6 199 4 25.97 4.18 0 5.22 OPeCML17 PEQU_24120 8 134 4 18.96 3.80 0 5.33 OPeCML18 PEQU_32857 8 140 4 17.95 3.81 0 4.31 OPeCML19 PEQU_32861 8 141 4 16.97 3.82 0 5.46 OPeCML20 PEQU_11970 8 140 4 17.96 3.87 0 6.82 OPeCML21 PEQU_32860 8 141 4 15.96 3.87 0 5.11 OPeCML22 PEQU_11969 8 237 5 25.95 4.28 1 6.30 OPeCML23 PEQU_25577 8 143 3 18.95 4.38 0 4.41 OPeCML24 PEQU_28290 8 143 4 15.96 3.77 0 5.02 OPeCML25 PEQU_05240 3 143 3 16.97 3.88 0 4.92 OPeCML26 PEQU_19202 5 160 3 18.97 3.96 0 3.83 OPeCML27 PEQU_17666 5 197 3 22.95 4.19 0 3.77 OPeCML28 PEQU_35797 6 110 3 11.96 3.81 0 3.61 OPeCML29 PEQU_11304 8 152 3 19.96 3.85 0 4.16 OPeCML30 PEQU_20039 6 114 3 15.96 3.77 0 3.03 OPeCML31 PEQU_28291 8 110 3 11.96 3.78 0 4.23 OPeCML32 PEQU_09901 8 111 3 16.96 3.79 0 3.19 OPeCML33 PEQU_29894 2 101 3 6.95 3.78 0 2.68 OPeCML34 PEQU_32859 8 115 3 14.96 3.78 0 3.46 OPeCML35 PEQU_09640 9 183 4 38.96 3.87 0 3.10 OPeCML36 PEQU_15088 4 166 2 23.95 3.85 0 2.89 OPeCML37 PEQU_28312 4 176 2 20.97 3.83 0 2.54 OPeCML38 PEQU_00748 7 168 3 7.96 3.85 0 3.88 OPeCML39 PEQU_39569 10 108 2 14.95 3.81 0 3.16 OPeCML40 PEQU_40143 10 127 2 18.96 3.77 0 1.93 OPeCML41 PEQU_09077 4 103 2 13.96 3.88 0 2.42 OPeCML42 PEQU_09518 4 158 2 21.96 3.89 0 2.02 OPeCML43 PEQU_24484 10 225 4 34.95 4.28 3 5.33 OPeCML44 PEQU_00516 7 106 2 7.97 3.77 0 2.11 OPeCML45 PEQU_11010 1 304 2 30.96 4.75 4 1.66 OPeCML46 PEQU_04326 1 253 2 23.96 4.56 0 3.35 OPeCML47 PEQU_41809 6 219 4 24.96 4.23 5 4.26 OPeCML48 PEQU_17666 5 197 3 22.97 4.06 0 3.28 OPeCML49 PEQU_01306 10 153 3 18.95 3.99 1 3.66 OPeCML50 PEQU_12715 10 147 3 17.97 4.20 2 2.82 OPeCML51 PEQU_17236 10 211 4 17.96 4.12 1 4.21 OPeCML52 PEQU_00999 1 113 4 13.96 4.32 0 3.16 OPeCML53 PEQU_12714 10 153 2 19.95 4.41 1 3.19 OPeCML54 PEQU_01183 1 121 3 13.96 3.52 0 2.47 Table 1. The characteristics of OPeCaM/OPeCML protein sequence in P.equestris
名称
Name基因ID
Gene ID亚家族
subfamily蛋白序列长度
Length of protein/aaEF hands 分子量
Mol Wt/kDa等电点
pI内含子
Intron甲硫氨酸的含量
percentage methionine /%ODcCaM1 DG_10048944 1 149 4 16.96 3.89 1 6.01 ODcCaM2 DG_10042177 1 149 4 16.96 3.88 1 6.03 ODcCaM3 DG_10042221 1 149 4 16.96 3.88 3 6.00 ODcCaM4 DG_10069718 1 148 4 11.96 3.67 3 6.02 ODcCML01 DG_10099596 8 147 4 16.96 3.63 3 5.11 ODcCML02 DG_10026132 6 154 4 22.96 4.20 0 3.96 ODcCML03 DG_10032884 6 152 4 18.96 4.12 0 4.88 ODcCML04 DG_10102530 6 154 4 18.97 4.32 0 4.46 ODcCML05 DG_10001096 6 165 4 22.96 4.41 0 5.52 ODcCML06 DG_10086740 6 142 4 12.95 3.52 0 4.26 ODcCML07 DG_10112859 6 154 4 21.96 4.16 0 4.14 ODcCML08 DG_10069652 6 140 4 20.15 3.78 0 3.82 ODcCML09 DG_10048052 6 143 4 16.96 3.75 0 6.26 ODcCML10 DG_10013983 6 145 4 12.96 3.73 0 3.93 ODcCML11 DG_10087227 6 145 4 11.96 3.73 0 3.74 ODcCML12 DG_10120493 10 113 3 13.95 3.79 0 2.43 ODcCML13 DG_10127394 2 112 3 13.96 3.78 0 2.16 ODcCML14 DG_10045310 2 177 3 17.97 4.88 1 6.29 ODcCML15 DG_10099614 9 124 3 7.96 3.77 2 5.26 ODcCML16 DG_10006683 10 86 2 9.98 3.88 1 1.19 ODcCML17 DG_10099594 9 130 3 14.96 3.89 2 3.95 ODcCML18 DG_10012949 8 107 2 11.97 3.80 1 2.27 ODcCML19 DG_10018482 6 110 3 13.96 3.82 0 3.36 ODcCML20 DG_10068885 6 206 4 25.96 4.67 0 4.42 ODcCML21 DG_10080246 6 206 4 25.96 4.67 0 5.16 ODcCML22 DG_10107219 7 195 4 27.95 4.28 0 4.82 ODcCML23 DG_10115967 6 219 4 24.96 4.38 0 5.56 ODcCML24 DG_10045416 7 165 4 25.96 3.77 0 6.01 ODcCML25 DG_10131684 7 151 2 21.95 3.88 0 2.44 ODcCML26 DG_10013708 8 87 2 6.98 3.72 1 2.22 ODcCML27 DG_10086551 8 147 3 21.96 3.89 0 4.01 ODcCML28 DG_10057311 4 141 3 10.97 3.85 0 2.82 ODcCML29 DG_10047181 7 104 2 10.96 3.78 0 1.71 ODcCML30 DG_10112504 3 231 3 39.95 4.67 0 3.29 ODcCML31 DG_10114938 8 303 3 37.97 5.88 6 4.15 ODcCML32 DG_10061210 5 107 3 12.96 3.79 3 3.23 ODcCML33 DG_10067799 7 100 3 7.96 3.78 0 4.53 ODcCML34 DG_10089833 7 195 4 28.97 4.58 0 5.14 ODcCML35 DG_10069653 6 116 3 18.95 3.87 0 2.96 ODcCML36 DG_10112846 6 133 3 21.96 3.85 0 3.54 ODcCML37 DG_10112841 6 128 3 20.96 3.83 0 3.26 ODcCML38 DG_10122957 6 133 3 21.95 3.85 0 2.80 ODcCML39 DG_10012468 7 190 4 24.97 4.58 0 2.51 ODcCML40 DG_10113507 6 185 4 25.97 4.17 0 3.82 ODcCML41 DG_10112861 6 337 4 48.95 5.88 2 3.99 ODcCML42 DG_10140024 6 148 2 16.96 3.89 1 1.58 ODcCML43 DG_10008482 6 184 3 30.96 4.28 0 3.44 ODcCML44 DG_10088170 6 175 3 31.97 4.02 0 2.15 ODcCML45 DG_10078503 7 189 4 17.96 4.27 0 5.52 ODcCML46 DG_10098264 7 189 4 17.95 4.27 0 4.32 ODcCML47 DG_10078224 7 195 3 24.96 4.99 0 3.66 ODcCML48 DG_10002690 7 107 2 11.95 3.72 0 2.32 ]ODcCML49 DG_10049524 7 176 2 23.93 4.25 0 1.20 ODcCML50 DG_10045416 7 165 2 25.97 4.17 0 2.21 ODcCML51 DG_10024582 7 181 2 24.96 4.28 0 3.03 ODcCML52 DG_10116046 7 327 2 29.97 5.88 4 2.52 ODcCML53 DG_10064998 7 691 2 76.94 8.85 10 2.23 ODcCML54 DG_10098265 5 191 4 25.95 4.58 0 4.19 Table 2. The characteristics of ODcCaM/ODcCMLprotein sequence in D. officinale
OPeCaM1 OPeCaM2 OPeCaM3 OPeCaM4 OPeCaM1 6 9 12 OPeCaM2 95.97 10 11 OPeCaM3 93.96 93.29 12 OPeCaM4 91.95 92.62 91.95 注:数字分别表示相似度(100%)和氨基酸差异数。
Note: The numbersaresimilarity(100%) and quantity difference amino acids.Table 3. Alignment of protein sequence of OPeCaMs
ODcCaM1 ODcCaM2 ODcCaM3 ODcCaM4 ODcCaM1 3 3 12 ODcCaM2 97.99 2 11 ODcCaM3 97.99 98.66 11 ODcCaM4 92.00 92.67 92.67 注:数字分别表示相似度(100%)和氨基酸差异数。
Note: The numbersaresimilarity(100%) and quantity difference amino acids.Table 4. Alignment of protein sequence of ODcCaMs
-
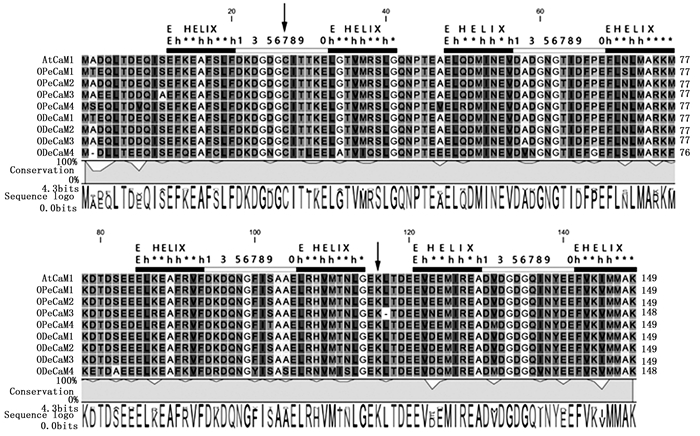
利用ClustalX 2.0.10软件将OPeCaM和ODcCaM蛋白序列与拟南芥AtCaM1进行多重序列比对(图 1)。兰科植物CaM蛋白具有能与Ca2+结合的4个手性分子结构域(EF-hand),每个EF-hand都是由30个氨基酸组成的螺旋-环-螺旋结构(Helix-Loop-Helix)。第1至9位氨基酸组成E螺旋(E-helix),第22至30位氨基酸组成的F螺旋(F-helix),中间的12个氨基酸为结合Ca2+的环(Loop),均为9个氨基酸长度的E螺旋与F螺旋结合于钙离子环的两侧。E螺旋由1个谷氨酸(E)起始,疏水氨基酸依“h**h**h”的规则在序列分布,其中,“h”代表疏水氨基酸,“*”为任意一种氨基酸。这种特殊的分布模式同样适用于EF-hand1和EF-hand3手性分子的F螺旋,而EF-hand2和EF-hand4手性分子的F螺旋的氨基酸分布模式与该模式有细微的差别。中间Loop环上的1、3位点是非常保守的天冬氨酸(D);第5位点通常为天冬氨酸(D)或者天冬酰胺(N),但占据第7、9位点的氨基酸有多种可能;谷氨酸(E)在0位点,功能是连接2个配位点;甘氨酸(G)在6位点,功能是保持环的结构;异亮氨酸(I)在8位点,功能是形成氢键与其他手性分子环结合成对。
在兰科植物CaM蛋白序列27位上存在1个半胱氨酸(C),常见于植物钙调蛋白中,在动物钙调蛋白中却没有被发现[23];在116位上存在1个位于第3个和第4个手性分子之间的赖氨酸(K),这一位点被认为是三甲基作用的潜在位点,存在于除酵母CaM以外的所有真核生物的CaM中[24]。
另外,OPeCaM和ODcCaM的甲硫氨酸的含量均为6%左右。OPeCML5、OPeCML15、OPeCML33、OPeCML35、ODcCML6、ODcCML7、ODcCML8、ODcCML18、ODcCML28、ODcCML42都包含潜在的豆蔻酰化序列(表 1、表 2)。完整的CaM结构是CaM结合Ca2+和靶蛋白结合进而行使功能的前提。
-
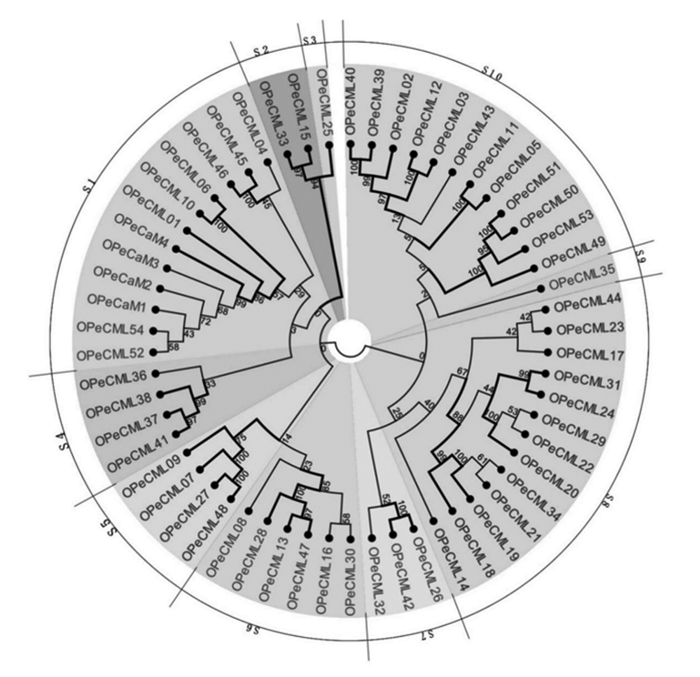
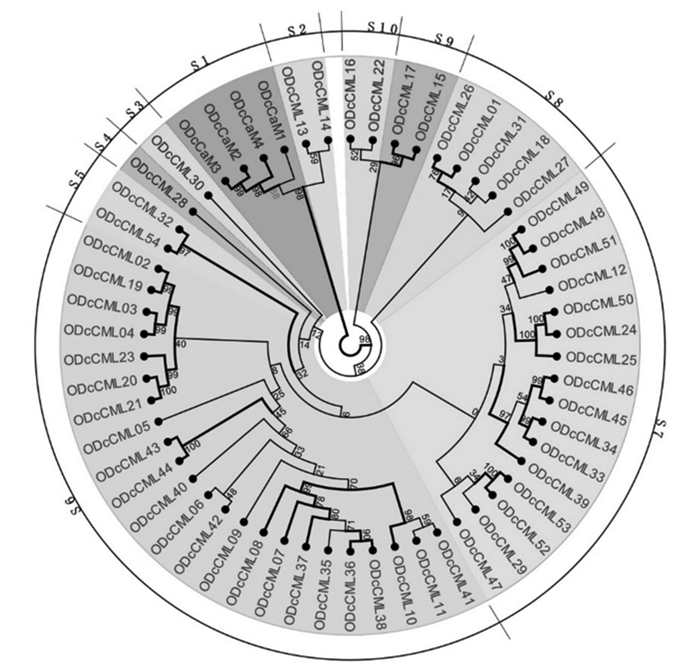
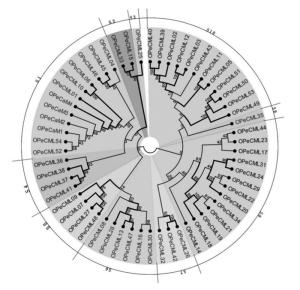
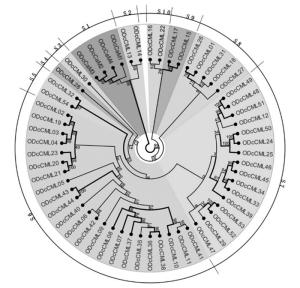
利用最大似然法(ML)构建小兰屿蝴蝶兰和铁皮石斛的CaM及CML氨基酸序列系统进化树(图 2、3)。依据拟南芥CaM及CML亚家族分类方法(9个亚家族)[25],将小兰屿蝴蝶兰和铁皮石斛CaM及CML蛋白各自分成10个亚家族,分别命名为S1~S10,其中,OPeCaM1~4与OPeCML01、OPeCML04、OPeCML06、OPeCML10、OPeCML45、OPeCML46、OPeCML52、OPeCML54聚类在同一个亚家族里,而ODcCaM1~4聚类的亚家族里没有ODcCMLn;OPeCML25、OPeCML35、ODcCML28、ODcCML30均单独聚类到一个亚家族。将Bootstrap≥75%分支加粗,在小兰屿蝴蝶兰系统进化树中S2、S3、S5的Bootstrap值均在75%~100%,并且各分支中Bootstrap值≥75%较多;铁皮石斛系统进化树中S5的Bootstrap值为97%,其他各亚群中Bootstrap值≥75%也较多。说明构建的系统进化树可信值较高。
-
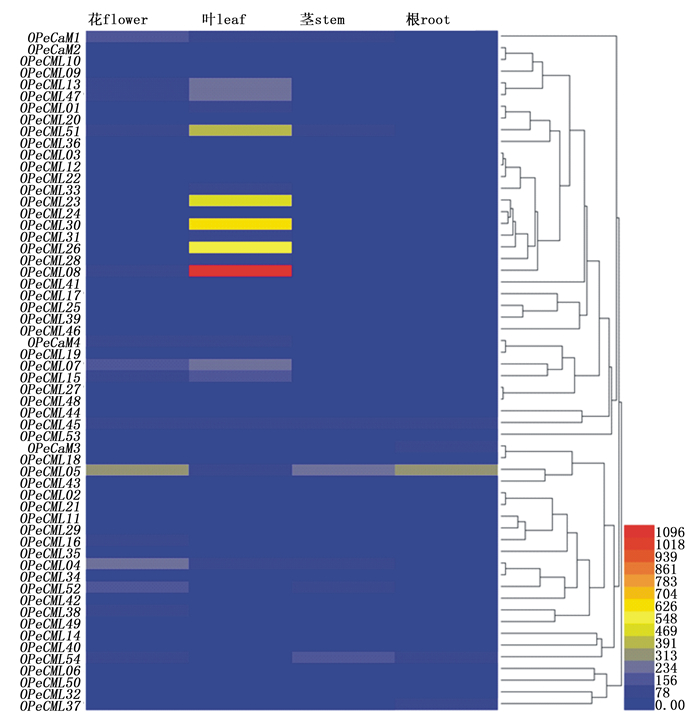
为确定CaM及CML基因在兰科植物不同组织中的表达情况,本文利用小兰屿蝴蝶兰花、叶、茎、根4个组织的转录组测序数据进行基因表达分析,结果(图 4)表明:OPeCML13、OPeCML47、OPeCML51、OPeCML23、OPeCML30、OPeCML26、OPeCML08、OPeCML07和OPeCML15在叶中的表达相对于花、茎、根为上调表达,表达量最高的是OPeCML08(RPKM为1 178);而OPeCML05和OPeCML54在叶中的表达相对于花、茎、根却为下调表达;OPeCaM1、OPeCML16和OPeCML38在花中的表达相对于叶、茎、根为上调表达;OPeCaM4、OPeCML07和OPeCML15在花和叶中的表达相对于茎和根为上调表达。
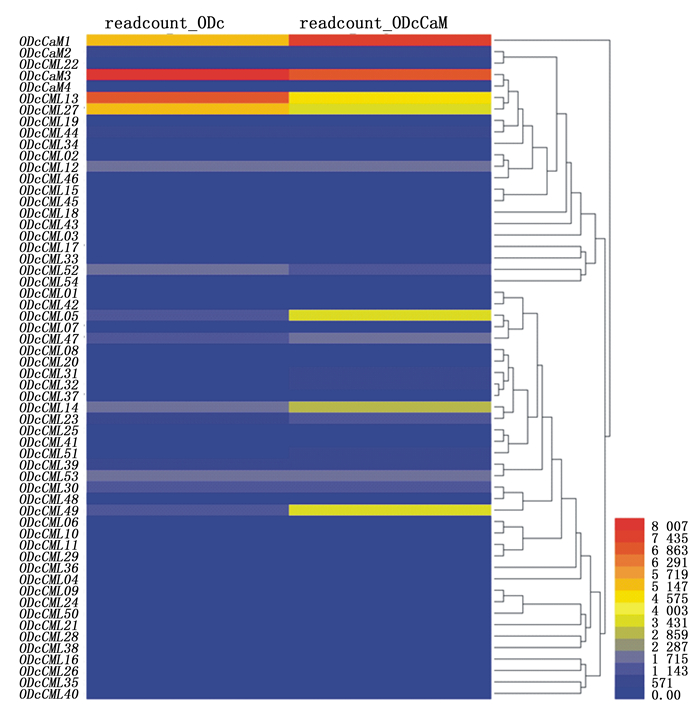
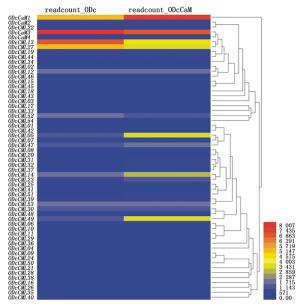
为确定CaM和CML基因在铁皮石斛种子与菌根菌共生萌发过程中的表达水平,本文利用铁皮石斛体外非共生萌发种子(ODc)和共生萌发种子(ODcTcg)转录组数据进行ODcCaM和ODcCML基因的表达分析, 结果(图 5)表明:ODcCaM1、ODcCML05、ODcCML14和ODcCML49在共生萌发文库中的表达相对于非共生文库为上调表达,表达量最高的是ODcCaM1(RPKM为8 007);而ODcCaM3、ODcCML13、ODcCML27和ODcCML52在共生萌发文库中的表达相对于非共生文库为下调表达。
2.1. 兰科植物CaM及CML家族蛋白的鉴定
2.2. 兰科植物CaM蛋白的多重序列比对
2.3. 兰科植物CaM及CML蛋白系统进化分析
2.4. 兰科植物CaM及CML基因表达分析
-
钙是植物生长发育所必需的营养元素,也是一种转导多种生理过程的重要信号分子。钙调蛋白(CaM)以及类钙调蛋白(CML)是细胞内两种重要的钙结合靶蛋白,开展植物中钙调蛋白和类钙调蛋白的研究,对了解上述基因在植物生长发育、细胞代谢、环境胁迫以及生物胁迫等方面的作用具有重要意义。目前,CaM基因和CML基因已在多种植物中开展研究,如拟南芥(Arabidopsis thaliana (L.) Heynh.)基因组中发现6个AtCaM基因和50个AtCML基因[25];水稻(Oryza sativa L.)基因组中发现5个OsCaM基因和32个OsCML基因[26];毛果杨(Populus trichocarpa Torr. & Gray)基因组中发现9个PtCaM基因和50个PtCML基因[27];胡杨(Populus euphratica)基因组中发现8个PeCaM基因和51个PeCML基因[27]。Zhao等[28]于2013年完成了对茄属(Solanum L.)植物CaM基因的鉴别和功能分析,分别于番茄(Lycopersicon esculentum Miller),烟草(Nicotiana tabacum L.)和马铃薯(Solanum tuberosum L.)的基因组中鉴定到6、7和4个CaM基因。本研究利用生物信息学方法预测分析了小兰屿蝴蝶兰和铁皮石斛的CaM和CML基因家族,发现2种兰科植物基因组中均含有4个CaM基因和54个CML基因,较上述植物中CaM和CML基因数量少。对比分析NCBI所发表的基因组数据发现,拟南芥、水稻、毛果杨和胡杨分别有33 583、30 534、45 555和34 279个蛋白编码基因,而小兰屿蝴蝶兰和铁皮石斛基因组分别有29 431个和35 567个蛋白编码基因,推测该基因家族的数量可能不与基因组蛋白编码基因的数量成正比。有研究表明,在拟南芥、水稻和杨树中,片段复制和串联复制可能是导致CaM和CML基因家族扩张的主要原因[25-27]。
2003年Mccormack等[25]对拟南芥的CaM及CML基因家族进行了分析,共找到了6个CaM基因和50个CML编码基因,其中,6个CaM基因编码的蛋白均含有4个手性分子结构域,推测6个CaM基因并不足以维持植株的功能,分布于50个CML中的手性分子结构域与CaM中手性分子作用类似;同时与CaM基因家族相比,CML基因家族在手性分子的数量和部分功能上有改变。本研究发现,在2种兰科植物基因组中均含有4个CaM基因和54个CML基因,CML基因数量远大于CaM基因数量,且CML基因具有1~5个数量不等的手性分子结构域。推测CML基因家族是CaM基因在进化过程中演变产生的,这一演变结果体现出明显的基因进化作用,同时CML中手性分子在序列上的差异可能反映了不同的CML基因家族成员间对Ca2+信号响应的微小差别[29]。
甲硫氨酸通过侧链异常的灵活性和极化性影响钙调蛋白的结构和功能,当CaM结合钙离子时,CaM形成开放构象,使结构更加稳定[30]。本研究发现,小兰屿蝴蝶兰和铁皮石斛中的CaM与拟南芥、水稻、杨树等植物的CaM甲硫氨酸含量相同,均为6%左右[25-27];豆蔻酰化序列的存在影响到对信号转导的调节,在拟南芥中就发现有4个AtCML蛋白序列存在潜在的豆蔻酰化位点[25],而水稻中只有1个[26],本研究在小兰屿蝴蝶兰和铁皮石斛中分别找到了4个和6个。
Mccormack等[25]通过对拟南芥CaM及CML蛋白家族进行系统进化分析,将CaM及CML蛋白分成了9个亚家族;Boonburapong等[26]在2007年对水稻的CaM及CML蛋白进行了分析,将其分为6个亚家族。本研究依据拟南芥CaM及CML亚家族分类方法,将小兰屿蝴蝶兰和铁皮石斛分别分成了10个亚家族, 并通过Bootstrap值≥75%分支较多,推测出构建的系统进化树可信度较高。研究发现,AtCML49/50通过影响脱落酸介导(ABA-mediated)途径调控拟南芥在盐胁迫下的耐受性[25],OPeCML11和ODcCML05/30与AtCML49/50聚类到同一分支,推测它们在功能上存在相似性。拟南芥AtCML37通过茉莉酸信号途径结合钙离子从而调节其抗虫害胁迫的耐受性[25],同时OPeCML40/45和ODcCML52/53与AtCML37聚类到同一分支,由此推测, 他们可能调控兰花在抗虫害胁迫方面的耐受性。庞俊峰等[31]于2010年通过生物信息学分析了木榄(Bruguiera gymnorrhiza (L.) Poir.)的CaM及CML基因家族,并与拟南芥、花生、大麦、榉木及甘薯等植物进行了比较,发现聚类到同一个亚家族的蛋白,不仅在结构上基本相同,所含有的外显子和内含子数也基本一致。通过本文CaM及CML基因家族系统进化树和结构图分析,也可以得到相同的推测。
大量研究表明,CaM及CML家族基因参与植物不同组织在生长发育过程中的响应机制[32-34]。芒果(Mangifera indica L.)在花瓣分化、雄蕊分化、雌蕊分化时期时花中Ca2+的含量相比其它组织中的含量增加,CaM/CML含量也随之升高[33];梅花(Armeniaca mume Sieb.)中花芽的CaM/CML含量相比于其他组织也较高[34]。杨树在叶发育过程中,高表达量的PtCaM及PtCML家族基因均参与钙调蛋白的积累过程[27]。在拟南芥矮小突变体和正常植株的研究中发现,AtCaM及AtCML的含量不同,说明CaM/CML参与调控植物茎的生长发育[35-36]。Yang等[37]在种子生根过程的研究中发现,CaM/CML表达量随着根的发育逐渐降低。本研究对CaM及CML基因在小兰屿蝴蝶兰花、叶、茎、根中表达水平分析发现,9个基因在叶中的表达相对于花、茎、根为上调表达,其中,OPeCML08表达量最高,RPKM为1 178;而OPeCML05和OPeCML54在叶中的表达相对于花、茎、根却为下调表达。推测它们可能在小兰屿蝴蝶兰的叶中发挥着某些重要作用。OPeCaM1、OPeCML16和OPeCML38在花中的表达相对于叶、茎、根为上调表达;OPeCaM4、OPeCML07和OPeCML15在花和叶中的表达相对于茎和根为上调表达。推测这些基因可能在兰科植物不同组织过程中起到关键性作用。
前期研究结果发现,在植物受到生物及非生物胁迫时,CaM及CML家族基因具有重要作用[24]。Boonburapong等[26]针对水稻叶、根、花、未成熟的种子和愈伤组织PCR扩增反转录得到的cDNA,结果发现生物胁迫下的水稻叶子中OsCaM1-1基因表达量显著增多。这与“细胞中Ca2+的变化是高等植物对病虫害侵染的早期反应之一[38]”的结论吻合。大豆SCaM4和SCaM5的表达受到病原真菌的强烈诱导,这一诱导事件也可以通过外施Ca2+来实现,然而用Ca2+螯合剂处理后抑制了大豆钙调蛋白基因在病原菌诱导下的表达[39]。Yamakawa等[40]通过沉默NtCaM2和NtCaM6基因,发现烟草(Nicotiana tabacum L.)对脆裂病毒、瓜果腐霉菌和真菌病原体这三种常见病害的抵抗力显著降低,推测NtCaM基因家族在抗生物胁迫方面发挥着显著的作用。烟草NtCaM4和NtCaM5基因在正常环境下表达水平很低,但在病原体侵入的条件下表达量明显提高,表现出高诱导性[40]。马尾松(Pinus massoniana Lamb.)接种松材线虫后,马尾松苗为响应松材线虫侵染,根、茎、叶器官中的PmCaM基因均不同程度地上调表达[41]。Leba等[42]对拟南芥CML基因在多种胁迫条件下研究发现,AtCML9受到致病菌、鞭毛蛋白和水杨酸的诱导后上调表达。Harrison等[43]研究植物在受到丛枝菌根真菌侵染后,胞内Ca2+浓度迅速升高,推测Ca2+-CaM/CML信号通路被激活,CaM及CML基因活跃表达。兰科植物在自然条件下必须从特定的真菌中获得营养才能成功萌发,并在随后的生活史中与真菌建立稳定的共生关系[5-6]。这些特定的真菌被定义为兰科菌根真菌,均属于丛枝菌根真菌[7]。因此,对兰科植物与兰科菌根真菌的共生关系建立过程中CaM和CML基因家族的的表达情况进行分析,对进一步阐明共生机制具有重要意义。本研究发现,在非共生和共生的铁皮石斛种子中ODcCaM1、ODcCML05、ODcCML14和ODcCML49在共生萌发文库中的表达相对于非共生文库为上调表达;而 ODcCaM3、ODcCML13、ODcCML27和ODcCML52在共生萌发文库中的表达相对于非共生文库为下调表达。推测这些基因可能参与铁皮石斛种子与美孢胶膜菌共生萌发中的生物学调控。
-
本研究依据已发表的拟南芥CaM和CML基因家族蛋白序列,利用生物信息学方法对小兰屿蝴蝶兰和铁皮石斛CaM和CML基因家族进行分析。在小兰屿蝴蝶兰和铁皮石斛全基因组中均预测到4个CaM蛋白和54个CML蛋白;小兰屿蝴蝶兰CaM及CML家族基因中39个基因没有内含子,19个基因有内含子;铁皮石斛CaM及CML基因家族中有41个基因没有内含子,17个基因有内含子;系统进化分析将其各分为10个亚家族;小兰屿蝴蝶兰中9个基因在叶中的表达相对于花、茎、根为上调表达,2个基因在叶中的表达相对于花、茎、根却为下调表达,3个基因在花中的表达相对于叶、茎、根为上调表达,3个基因在花和叶中的表达相对于茎和根为上调表达。说明CaM及CML家族基因在兰科植物不同组织中具有重要作用;铁皮石斛中4个基因在共生萌发文库中的表达相对于非共生文库为上调表达,4个基因在共生萌发文库中的表达相对于非共生文库为下调表达。说明CaM和CML基因家族可能参与铁皮石斛种子与美孢胶膜菌共生萌发中的生物学调控。本研究为进一步研究兰科植物中CaM和CML基因家族的潜在生物学功能提供数据基础。






 DownLoad:
DownLoad: